中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 贾仲君, 蔡元锋, 贠娟莉, 杜文斌. 2017
- Zhongjun Jia, Yuanfeng Cai, Juanli Yun, Wenbin Du. 2017
- 单细胞、显微计数和高通量测序典型水稻土微生物组的技术比较
- Comparison of soil microbiome by single cell technology, classical microscope methods and high-throughput MiSeq sequencing
- 微生物学报, 57(6): 899-919
- Acta Microbiologica Sinica, 57(6): 899-919
-
文章历史
- 收稿日期:2017-02-16
- 修回日期:2017-04-07
- 网络出版日期:2017-04-14
2. 中国科学院微生物研究所, 微生物资源前期开发国家重点实验室, 北京 100101
2. State Key Laboratory of Microbial Resources, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
土壤是地球上异质性最强的历史自然体,孕育着地球最高的微生物多样性,是地球最大的基因资源库[1]。然而,受限于微生物分类学理论的发展,迄今尚未有一种方法能真实反映土壤中微生物的物种组成和准确数量。卡尔•伍思提出的rRNA为基础的生物三域分类理论尽管未能完全解决地球环境中微生物分类的理论难题[2],但提供了一种事实上可操作的、针对海量微生物的分类学策略[3]。因此,20世纪90年代以来,随着DNA测序技术的快速发展,针对复杂环境如土壤、海洋、动物和植物等体系的微生物组研究成为不同学科、不同领域实验室的一种常规策略,从根本上改变了传统微生物生态学的研究理念,发现了大量前所未知微生物,微生物组研究成为工业、农业和医药等行业的重要学科交叉前沿。
土壤微生物的定量一直是微生物学研究难点。理论上最佳的定量策略包括:有效分离土壤中的非生物颗粒与微生物细胞,随后对微生物细胞进行准确计数。然而,目前尚未有被广泛认可的普适性土壤微生物细胞提取策略,准确定量土壤微生物的真实数量理论上仍不可行。主要原因是:土壤母质类型多样、发生过程复杂,并且土壤中绝大部分物质尚未有明确的化学结构[1],因此,很难设计一种普适性的提取缓冲液,实现复杂土壤中微生物细胞与土壤颗粒的完全分离。同时,传统的微生物细胞计数方法通常依赖于细胞染色和生长,涉及的一系列操作过程也存在较大的计数偏差。(1) 基于细胞染色的计数方法通常具有较大误差[4-5]。例如,经典的DAPI (4, 6-diamidino-2-phenylindole)染色计数法、荧光原位杂交(fluorescence in situ hybridization,FISH)、酶联荧光原位杂交CARD-FISH (catalyzed reporter deposition-FISH)方法,皆采用特异的化学物质,通过对细胞内核酸DNA直接染色或杂交放大外源荧光信号并在特定波长下显影成像,直接计数细胞数量。但细胞提取的效率、非生物颗粒干扰、细胞通透性及其对染色物质的吸收、显微设备的检测效率等,均会对微生物计数结果产生较大影响。(2) 经典的平板培养计数法或最大或然数法(most probable number,MPN)则依赖于细胞的生长,计数结果与自然环境中的微生物真实数量相去甚远[6-7]。事实上,这类方法主要根据可培养细胞在固体培养基生长形成肉眼可见的菌落,或者在液体培养基中产生可检测的浑浊度变化,结合稀释倍数推算环境中的微生物细胞数量。尽管在微生物学发展早期曾发挥重要作用,但这类方法很难模拟原位环境条件,并且依赖于细胞的生长,因此事实上仅能大致反映土壤中可培养微生物的多寡及其在不同环境中的相对优势度。此外,这些传统微生物研究方法操作繁琐,流程复杂,需要专业技术人员和特殊实验室,在很大程度上限制了微生物学的发展及其与相关领域的跨学科交叉研究。
基于核酸序列的物种分类已经成为土壤微生物组研究的重要内容。传统微生物分类主要基于细胞形态、结构和生理生化特征等属性。20世纪80年代后期,随着测序技术的快速发展,基于核酸DNA/RNA的分子生物学技术逐渐成为土壤微生物数量和组成研究的主要策略[8]。关键流程包括复杂环境中微生物基因组总核酸DNA/RNA提取,进而对其16S rRNA基因或微生物标靶基因进行特异扩增,随后对PCR产物进行测序并分析微生物组成[9];或者针对特定靶标基因开展qPCR分析微生物数量[10]。特别是2007年以来高通量测序技术发展迅猛,已经成为主流的分子生态学研究手段,逐渐替代了传统的分子指纹图谱技术,如克隆文库技术、变性梯度凝胶电泳DGGE (denaturing gradient gel electrophoresis)[11]、末端限制性片段长度多态性分析T-RFLP (terminal-restriction fragment length polymorphism)。此外,新兴的单细胞筛选及分析技术代表着未来微生物学研究的一个重要方向。细胞是生命的基本单元,利用各种提取策略获得复杂环境中微生物细胞混合物之后,理论上不仅可对单个细胞计数,也可对单个细胞基因组进行分析,研究同种或近源物种的生态进化规律。单细胞技术同时能够规避传统MPN和平板计数法研究难培养微生物的内在缺陷,辅之以高效的土壤微生物细胞提取技术,可能是未来研究原位环境中微生物生理功能和物种分化的重要手段[12]。
总体而言,无论传统显微计数还是现代分子方法,土壤微生物组的研究对象包括:可提取的土壤微生物细胞(土壤细胞)、微生物细胞DNA (细胞DNA)、直接从土壤中提取微生物组总DNA (土壤DNA)。然而,目前土壤微生物组不同技术优势的比较研究较少,许多技术问题仍处于定性描述阶段。例如:(1) 土壤微生物组的数量比较。如基于土壤细胞的DAPI染色、CARD-FISH杂交计数,基于细胞DNA、土壤DNA的qPCR数量之间的相互比较。(2) 土壤微生物组的物种组成比较。如基于土壤DNA、细胞DNA测序的微生物组成是否一致。土壤细胞经单细胞筛选及全基因组扩增后,基于单细胞DNA测序的微生物多样性似乎未见报道。此外,针对复杂土壤环境中的特定微生物生理过程如好氧甲烷氧化,不同研究方法能否准确反映好氧甲烷氧化菌的群落变化规律,目前仍不清楚。据此,本研究采用DAPI染色、CARD-FISH杂交、qPCR分析了一种典型水稻土中的微生物数量;采用高通量测序16S rRNA基因的PCR扩增产物研究了微生物的物种组成;基于显微计数和分子方法,比较分析了水稻土好氧甲烷氧化的微生物生理生态过程。
1 材料和方法 1.1 土壤采集及实验技术路线 1.1.1 土壤采集: 水稻土于2012年11月采自江苏扬州宗村良种场(119°42′0"E, 32°35′5"N),隶属于中国FACE (free air CO2 enrichment)大气二氧化碳浓度升高全球变化平台。该区域属于典型的中国亚热带单季稻麦轮作农业区。土壤类型为钙质潜育土(calcaric glevsols),植稻历史大于50年。用不锈钢土钻采集表层15 cm土壤,土样风干后经过研磨,并过2 mm筛除去大颗粒杂质,4 ℃保存备用。土样最大持水量(maximum water holding capacity,WHC)为55%,总有机碳15 g/kg,总氮1.59 g/kg,总磷1.23 g/kg,pH 7.4 (水土比2.5)[13]。 1.1.2 实验技术路线: 如图 1所示,每个土壤可获得4种不同的样品:直接提取的土壤微生物细胞(土壤细胞)、从土壤细胞悬液进一步提取的细胞DNA (细胞DNA)、从土壤细胞悬液经单细胞分选及全基因组扩增后获得的DNA (单细胞DNA)和直接提取的土壤微生物总DNA (土壤DNA)。(1) 针对水稻土微生物组的数量:采用DAPI染色和CARD-FISH杂交两种显微计数方法,分析土壤细胞数量;采用qPCR分子技术分析细胞DNA和土壤DNA中微生物基因数量。(2) 针对水稻土微生物的物种组成:采用高通量测序16S rRNA和pmoA基因的方法,分析土壤DNA、细胞DNA、单细胞DNA中的微生物组成。水稻土样品则来自高浓度甲烷氧化前后,即零时刻的背景土壤(Day 0) 和氧化了600 μmol甲烷的水稻土(Day-17)。最后比较传统显微计数、现代分子方法研究土壤微生物组的数量、组成及好氧甲烷氧化微生物生理生态过程的技术特点。
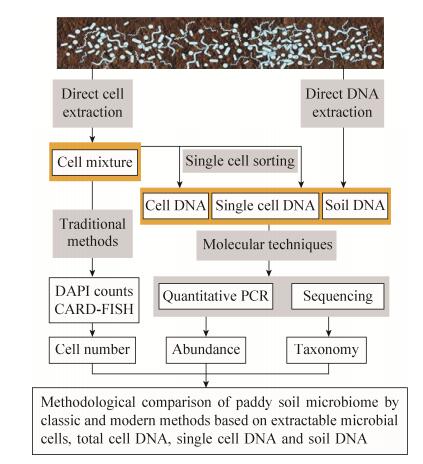
|
| 图 1. 实验整体思路和技术流程 Figure 1. Flow diagram of the study. |
1.1.3 水稻土好氧甲烷氧化微生物过程: 调整土壤含水量至最大持水量的60% (实际含水量33%),在无菌封口袋中充分混匀后,28 ℃预培养4 d,使土壤微生物充分活化。取8 g作为0时刻样品(Day 0),–20 ℃保存用于总DNA及土壤细胞的提取。其余土样用于微宇宙培养。在120 mL血清瓶中加入8 g水稻土,设置3个平行,用丁基橡胶塞密封后用铝盖封口,抽真空后,用注射器注入14.4 mL CH4,36 mL O2,并用N2补充至1个大气压,甲烷初始浓度约85.7 g/m3,28 ℃避光培养。前10 d每天监测1次CH4浓度,10 d后每3–4 d监测1次。待瓶内甲烷浓度降至7.14 g/m3以下(第17天)终止实验,取出土壤保存于–20 ℃用于总DNA及土壤细胞的提取。培养0时刻(Day 0) 及培养17 d (Day 17) 的土壤样品用于后续土壤微生物组和甲烷氧化菌的数量和组成分析。 1.2 土壤微生物组及好氧甲烷氧化菌细胞数量分析 1.2.1 土壤微生物细胞提取: 利用差速离心结合Nycodenz密度梯度离心的方法从土壤中提取微生物细胞[14],方法略有改动,步骤如下:(1) 称取1 g土壤样本至10 mL无菌离心管,加入0.1 mm的玻璃珠(Sigma) 0.5 g、0.2%焦磷酸钠(sodium pyro-phosphate,pH 7.5) 溶液5 mL,涡旋2 min后250×g低速离心2 min。(2) 准备新的10 mL离心管,加入4 mL Nycodenz溶液(密度1.3 g/mL,8 g Nycodenz+10 mL ddH2O),将低速离心得到的上清液贴壁缓缓加到Nycodenz溶液上方(切勿扰动上清液与Nycodenz之间形成的界面)。4 ℃,9800×g离心50 min (离心条件:Acc:4,Del:3)。(3) 离心结束后,吸取水层和Nycodenz层之间的灰白色细胞层转移至2 mL离心管中,4 ℃下12000×g离心10 min,弃上清。然后用1×PBS缓冲液洗涤菌体细胞2次(12000×g离心2 min)。(4) 细胞沉淀最终用0.5 mL 4%多聚甲醛重悬,置于4 ℃过夜固定及保存。 1.2.2 土壤微生物组的DAPI染色计数: 将固定后的细胞悬液过滤到0.22 μm的聚碳酸酯滤膜上(GTTP,Millipore,直径25 mm),滤膜用1×PBS缓冲液清洗,晾干,在无菌培养皿中切取部分滤膜,在滤膜上滴加1 μg/mL的DAPI水溶液,室温下避光染色10 min,随后用过量双蒸水(ddH2O)清洗后晾干。将滤膜正反面用Citiflour有机油浸润,并借助有机油将滤膜固定在载玻片上,盖上盖玻片在荧光显微镜(Axio Imager A2,ZEISS)下观察并拍照。随机挑选10个视野统计细胞数量。根据视野及滤膜面积计算每克土壤中提取的细胞数量。 1.2.3 土壤微生物组的CARD-FISH计数: 用于总细菌计数的16S rRNA探针为EUB Ⅰ-Ⅲ [15],杂交步骤如下:(1) 将固定后的细胞悬液过滤到0.22 μm的聚碳酸酯滤膜上,滤膜用1×PBS缓冲液清洗,晾干,在培养皿中切取部分滤膜,背面做上标记。(2) 将滤膜置于0.01 mol/L HCl中室温下浸泡10 min,使细胞内过氧化氢酶失活,然后用1×PBS缓冲液清洗,晾干。(3) 将滤膜浸入新配制的溶菌酶缓冲液[溶菌酶10 mg/mL,并含0.05 mol/L EDTA,0.1 mol/L Tris-HCl (pH 8.0)]中,37 ℃下处理30 min,以增加细胞的通透性。随后用ddH2O清洗后晾干。(4) 取600 μL杂交缓冲液[0.9 mol/L NaCl,20 mmol/L Tris-HCl (pH 8.0),20% dextran sulfate (W/V),0.02% (W/V) sodium dodecyl sulfate (SDS),35% (V/V) formamide,1%(W/V) Blocking Reagent]加入1.5 mL的离心管中,加入2 μL探针(50 ng/μL);将滤膜全部浸入,46 ℃杂交2 h。(5) 将滤膜从杂交缓冲液中取出,浸入50 mL预热至48 ℃的探针洗涤液[70 mmol/L NaCl,5 mmol/L EDTA (pH 8.0),20 mmol/L Tris-HCl (pH 8.0),0.01% (W/V) SDS],在48 ℃下继续水浴10 min;随后取出滤膜,将其浸入1×PBS缓冲液中常温下放置15 min,之后用滤纸吸掉滤膜上多余的液体。(6) 配制酪胺信号扩增体系:10 μL 0.15% H2O2 (现用现配)、1 μL Alexa Fluor™ 488 Tyramide Reagent (B40953,150 slides,150 μL DMSO溶解)、989 μL扩增缓冲液[在1×PBS缓冲液中包含2 mol/L NaCl,10% dextran sulfate (W/V),1% (W/V) Blocking Reagent],将滤膜浸入其中,37 ℃避光水浴30 min。然后在1×PBS缓冲液中浸泡10 min,用过量ddH2O清洗。(7) 将滤膜正反面用Citiflour有机油浸润,并借助有机油将膜固定在载玻片上,盖上盖玻片在荧光显微镜下观察、拍照并计数。 1.2.4 土壤好氧甲烷氧化菌的CARD-FISH计数: 好氧甲烷氧化菌主要分为type Ⅰ及type Ⅱ两大类,分别属于γ-和α-Proteobacteria,对它们进行CARD-FISH杂交用的16S rRNA探针分别为Mγ84/705和Mα450[16]。具体步骤和土壤微生物组的CARD-FISH计数步骤基本相同,两处不同之处为:杂交缓冲液中formamide的比例为20%,探针洗涤液中NaCl的浓度为215 mmol/L。 1.3 土壤微生物组及好氧甲烷氧化菌基因数量分析 1.3.1 土壤DNA的提取: 采用FastDNA Spin Kit for Soil (MP Bio)试剂盒提取土壤总DNA,土壤用量为0.5 g,提取步骤严格按照试剂盒说明书进行。利用微量紫外分光光度计(NanoDrop® ND-1000 UV-Vis)测定提取的总DNA浓度及质量,随后将DNA置于–20 ℃保存,用于后续定量PCR及高通量测序分析。 1.3.2 细胞DNA的提取: 同1.2部分提取土壤微生物细胞,用10 mL 1×PBS缓冲液重悬细胞,随后将细胞悬液过滤到孔径0.22 μm的聚碳酸酯滤膜上(GTTP,Milipore),将滤膜用无菌剪刀剪碎,收集至2 mL无菌离心管中,用FastDNA Spin Kit for Soil (MP Bio)试剂盒提取细胞DNA,细胞DNA置于–20 ℃保存,用于后续定量PCR及高通量测序分析。 1.3.3 qPCR分析土壤总微生物数量: 土壤微生物的qPCR定量所用引物为原核微生物16S rRNA基因V4-V5区通用引物对515F (5′-GTGCCAGCMGCCGCGG-3′)/907R (5′-CCGTCAATTCMTTTRAGTTT-3′)。反应总体积为20 μL,包括10 μL SYBR Premix Ex TaqTM (TaKaRa)、0.25 μL正反向引物(50 μmol/L)、1 μL DNA模板以及8.5 μL无菌双蒸水。反应程序如下:95 ℃ 3 min;40×(95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,with plate read);melt curve 65 ℃ to 95 ℃,increment 0.5 ℃,0:05+plate read[17]。反应在CFX96 Optical Real-Time PCR System (Bio-Rad, Laboratories Inc.,Hercules,CA,USA)上进行。阴性对照采用灭菌ddH2O代替样品作为反应模板。标准曲线的制作简述如下:用相应的引物扩增任意一个土壤样品的DNA,PCR产物回收后进行TA克隆,选阳性克隆子培养后提取质粒,用微量紫外分光光度计(NanoDrop® ND-1000 UV-Vis)测定质粒浓度,根据含目标片段的质粒长度及质量浓度计算得出每微升的目标基因拷贝数。用无菌去离子水对质粒进行10倍梯度稀释,作为标准样品用于构建标准曲线。 1.3.4 qPCR分析土壤好氧甲烷氧化菌的数量: 土壤好氧甲烷氧化菌的qPCR定量所用引物为甲烷单加氧酶β亚基编码基因pmoA扩增通用引物对:A189F (5′-GGNGACTGGGACTTCTGG-3′)/mb661R (5′-CCGGMGCAACGTCYTTACC-3′)[18-19],反应程序如下:95 ℃ 3 min;40×(95 ℃ 10 s,55 ℃ 30 s,72 ℃ 30 s,80 ℃ 5 s,with plate read);melt curve 65 ℃ to 95 ℃,incremental 0.5 ℃,0:05+plate read。反应体系组成及标准曲线的制作过程同16S rRNA基因的qPCR。 1.4 单细胞DNA的获取 1.4.1 土壤微生物单细胞的分选: 首先提取土壤微生物细胞,方法同1.2部分。细胞沉淀最终用1 mL 1×PBS缓冲液重悬。然后,用血球计数板在显微镜(OLYMPUS PT-005) 下对提取的细胞进行计数,根据计数结果将细胞悬液稀释至104–105个/mL。最后,将1 mL稀释后的细胞悬液用1×SYBR Green室温避光染色30 min,用流式细胞仪(Moflo XDP,Beckman)将染色完毕的细胞分选到96孔板中。 1.4.2 单细胞全基因组扩增: 使用MDA (multiple displacement amplification)试剂盒(Qiagen REPLI-g Single Cell Kit)对单细胞进行全基因组扩增,使用前将各成分进行分装以避免污染。首先向96孔板中加入40 μL石蜡油(sigma aldrich),然后使用微量注射泵(harvard apparatus)加入裂解液(DLB),每孔30 nL,阳性对照中加入40 nL大肠杆菌基因组DNA (≤10 ng/μL),阴性对照孔中不加任何模板[20-21]。将96孔板放入预热至65 ℃的金属浴(H2O3-PRO)中热裂解10 min,结束后冰上冷却。随后,向96孔板中加入30 nL终止液中和反应。将Reaction buffer、ddH2O、φ29 Polymerase、50×SYBR Green从冰上取出,按照说明书配制MDA Mix,然后在每个孔中加入400 nL MDA Mix,2500 r/min离心30 s。MDA总反应体积为500 nL,反应条件为30 ℃恒温扩增10 h (RT-PCR仪,ABI 7500),每10 min采集1次荧光信号。Day 0样品检测到阳性孔84个,Day 17样品检测到阳性孔196个。在每个阳性孔内加入20 μL ddH2O对MDA扩增产物进行稀释。Day 0和Day 17样品每个样品的每个孔吸取2 μL混合,作为单细胞DNA的PCR扩增模板,用于后续的高通量测序分析。 1.5 土壤微生物组及好氧甲烷氧化菌群落的基因组成分析 1.5.1 高通量测序分析土壤微生物组16S rRNA基因组成: 土壤微生物16S rRNA基因V4-V5区扩增引物对为515F/907R,其中515F的5′端连接有12个碱基的barcode序列,用于区分不同的样品。PCR反应程序为:95 ℃ 3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,32个循环;72 ℃ 10 min。PCR扩增产物通过切胶回收试剂盒(MiniBEST DNA Fragment Purification Kit Ver.3.0,TaKaRa)进行纯化,测定浓度后等质量混合,然后建库测序。建库试剂盒为VAHTSTM Nano DNA Library Prep Kit for Illumina®(南京诺唯赞生物科技有限公司),测序试剂盒为MiSeq Reagent Kit v3 (600 cycles,Illumina)。Mothur (version 1.33.3) 软件用于测序结果的处理和分析[22]。“make.contigs”命令(deltaq=5) 用于双末端序列的拼接,“trim.seqs”命令用于原始序列的质量控制。“classify.seqs”命令用于高质量序列的taxonomic分类,比对数据库文件为trainset14_032015.rdp.fasta和trainset14_032015. rdp.tax,cutoff值设为80%。 1.5.2 高通量测序分析土壤好氧甲烷氧化菌16S rRNA基因的相对丰度: 在土壤总微生物16S rRNA基因分类完成后,从属水平的分类结果中筛选好氧甲烷氧化菌种类,统计其在总微生物中的相对丰度。进一步根据相对丰度数据计算各个甲烷氧化菌种类在整体甲烷氧化菌中的相对含量。 1.5.3 高通量测序分析土壤好氧甲烷氧化菌pmoA功能基因组成: 好氧甲烷氧化菌pmoA基因扩增引物为A189F和mb661R,其中A189F的5′端连接有6个碱基的barcode序列,用于区分不同的样品。反应程序为94 ℃ 3 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,32个循环;72 ℃ 10 min。PCR产物的纯化、建库及测序过程同16S rRNA基因。测序完成后,使用Mothur软件中的“trim.seqs”命令对序列进行质量控制,“classify.seqs”命令用于高质量序列的taxonomic分类,比对数据库文件为pmoa.mothur.2013.fasta和pmoa.mothur.2013.tax,cutoff值设为80%[23]。 1.6 统计分析
Alpha多样性和Beta多样性指数的计算使用Mothur (version 1.33.3) 软件完成[24]。16S rRNA基因按照97%的序列相似度划分OTU (Operational Taxonomic Unit),命名为OTU0.03,pmoA基因按照90%的核酸序列相似度分OTU,命名为OTU0.10。样品测序量进行归一化以避免测序深度的差异对多样性计算的影响。“rarefaction.single”命令用于计算样品OTU数量随测序量不断加深的变化过程。“unifrac.weighted”命令用于统计不同分组样品群落结构是否具有显著差异,样品间的差异性通过“pcoa”命令进行PCoA (principal coordinates analysis,主坐标分析)分析进行展示。不同处理间平均值差异的显著性采用SPSS 17.0软件中的one way-ANOVA或T-TEST进行检验。
2 结果和分析 2.1 土壤微生物组的研究方法比较及水稻土好氧甲烷氧化微生物过程传统显微计数和现代分子方法研究土壤微生物数量的问题包括:(1) 土壤微生物细胞的提取效率。目前技术条件下,一般认为土壤DNA来自于土壤微生物组中所有细胞的DNA,而直接提取的微生物仅反映了土壤中易与土壤颗粒分离的细胞。因此,如图 1所示,土壤DNA的qPCR定量在一定程度上可代表土壤中所有微生物的基因数量,而细胞DNA的qPCR定量则代表了土壤中可提取微生物的基因数量,两者之间的比例即为土壤微生物的细胞提取效率。(2) 可提取细胞DNA与直接提取土壤微生物DNA的物种差异比较。一般认为目前的提取技术很难获得所有微生物细胞。因此,通过测序土壤DNA、细胞DNA和单细胞DNA,即可初步推测可提取微生物在多大程度上能够代表土壤微生物组的全貌;可提取细胞悬液经单细胞分选后,其单细胞DNA分析是否具有代表性。
本研究针对2个土壤样本(Day 0,Day 17) 开展了上述分析。如图 2-A所示,与零时刻(Day 0) 相比,Day 17水稻土氧化了大约600 μmol的高浓度甲烷,相应地,培养瓶中零时刻甲烷浓度从85.7 g/m3降至6.14 g/m3左右。

|
| 图 2. 水稻土氧化甲烷的动力学过程(A)及土壤总微生物(B)与甲烷氧化菌(C)的数量变化 Figure 2. Consumption dynamics of methane by paddy soil (A), copy number changes of 16S rRNA (B) and pmoA genes (C) in 1 g wet weight soil (wws) or in cells exracted from 1 g wet weight soil before and after methane incubation. Error bars represent standard deviations (n=3), and columns with the different letters are significantly different (P < 0.05) by one-way ANOVA. |
2.2 基于qPCR的土壤微生物组及甲烷氧化菌数量比较
通过qPCR比较了土壤DNA和细胞DNA中的16S rRNA基因拷贝数,结果表明:土壤微生物细胞的提取效率约为2.0%。如图 2-B所示,Day 0土壤DNA中的16S rRNA基因数量在8.7×1010 copies/g,而细胞DNA中16S rRNA基因的数量在1.7×109 copies/g,两者相差50倍(ANOVA,P < 0.05)。类似的,Day 17土壤DNA中16S rRNA基因数量为1.6×1011 copies/g,相应的细胞DNA中16S rRNA基因数量为3.8×109 copies/g,两者相差41倍,土壤细胞提取效率(2.4%)和Day 0土壤样本基本一致。此外,通过比较Day 0和Day 17土壤DNA的qPCR定量结果,发现整体微生物数量略微增加1倍左右,基于土壤DNA和基于细胞DNA的qPCR结果均发现相同的增加趋势(图 2-B)。
通过对土壤DNA和细胞DNA中的pmoA基因进行qPCR定量,结果表明土壤甲烷氧化菌细胞的提取效率由零时刻的0.9%左右提高到高浓度甲烷培养后的6.1%左右(ANOVA, P < 0.05)。如图 2-C所示,零时刻土壤DNA和细胞DNA中甲烷氧化菌pmoA基因数量分别为2.1×107 copies/g和1.8×105 copies/g,细胞提取效率仅0.9%;高浓度甲烷培养之后,分别增加到1.2×109 copies/g和7.0×107 copies/g,土壤甲烷氧化菌细胞提取效率也增大到了6.1%左右(图 2-C)。
通过比较Day 0和Day 17土壤DNA、细胞DNA的qPCR结果,均发现高浓度甲烷培养导致甲烷氧化菌大量增长。土壤DNA的定量分析表明高浓度甲烷培养导致土壤甲烷氧化菌增加了54倍,而基于细胞DNA的qPCR分析表明这一增加量约为388倍。尽管两种方法的具体增加量不同,但基于土壤DNA和细胞DNA的定量结果均表明水稻土氧化高浓度甲烷后其中的甲烷氧化菌大量增加,两种方法均能较好表征水稻土中甲烷氧化菌的生长过程(图 2-C)。
2.3 基于DAPI染色和CARD-FISH杂交的土壤微生物组及甲烷氧化菌数量比较通过DAPI染色及CARD-FISH计数对土壤可提取微生物细胞计数。如图 3-A所示,Day 0样本中总微生物细胞的DAPI计数为5.8×107 cells/g,Day 17样本中略微增加至7.4×107 cells/g。而CARD-FISH的计数结果分别为1.7×107 cells/g (Day 0) 和1.9×107 cells/g (Day 17) (图 3-B)。DAPI染色的灵敏度更高,是CARD-FISH计数结果的3–4倍。

|
| 图 3. 高浓度甲烷培养前后土壤总微生物及甲烷氧化菌细胞经过DAPI染色(A)及CARD-FISH杂交(B,C,D)后的显微图像 Figure 3. Abundance changes of DAPI and CARD-FISH-stained cells extracted from 1 g wet weight paddy soils before and after high concentration methane incubation. A: DAPI-stained total cell; B: CARD-FISH of total bacteria using probes EUB338 Ⅰ-Ⅲ; C: CARD-FISH of type Ⅰ methanotrophs using probes Mγ84/705; D: CARD-FISH of type Ⅱ methanotrophs using probes Mα450. Cell numbers were obtained by classical microscopic counting method. *: t-test (n=10), P < 0.05; **: P < 0.01. |
利用Type Ⅰ和Type Ⅱ甲烷氧化菌的特异探针进行CARD-FISH计数,结果表明高浓度甲烷氧化过程中,好氧甲烷氧化菌的数量显著增加(图 3-C,D)。零时刻甲烷氧化菌数量较低,Type Ⅰ甲烷氧化菌约1.6×104 cells/g,Type Ⅱ甲烷氧化菌约2.1×104 cells/g;而高浓度甲烷培养后(Day 17),两类甲烷氧化菌数量均显著增加(t-test,P < 0.01),Type Ⅰ甲烷氧化菌数量增加至1.7×106 cells/g,增加100倍左右(图 3-C)。Type Ⅱ甲烷氧化菌增加到4.1×104 cells/g,增加1倍左右(图 3-D)。Day 0水稻土中甲烷氧化菌总量约3.7×104 cells/g;Day 17则增加至1.7×106 cells/g,增幅约45倍。
2.4 基于高通量测序的土壤微生物组成分析在微生物分类学门的水平,高通量测序土壤DNA和细胞DNA的16S rRNA基因组成基本一致,表明可提取细胞中涵括了栖息于土壤中的主要微生物类群。如图 4-A所示,在Day 0土壤DNA中含有30个已知的门,而细胞DNA中则检测到了25个门,且未检测到的5个门均为丰度非常低的门类,在土壤DNA中仅占0.14%。然而,同一个微生物门在土壤DNA和细胞DNA中的相对丰度具有明显差异,例如,Planctomycetes门在土壤DNA中仅占3.1%,但在细胞DNA中比例上升至19.6%。Acidobacteria门在土壤DNA中占12.2%,而在细胞DNA中仅占1.1%。此外,土壤中存在大量的未知微生物类群(unclassified bacteria),在土壤DNA中占比高达20.1%,而在细胞DNA中仅占4.0%,表明这些未知微生物细胞很难被提取。针对土壤细胞悬液的单细胞分选结果则表明,尽管也发现了20个门类的细胞,但分离出来的细胞绝大部分属于Proteobacteria门,可占95%以上,具有非常明显的偏好性(图 4-A)。

|
| 图 4. 高浓度甲烷培养前后土壤DNA、细胞DNA及单细胞DNA中总微生物的群落结构及多样性的变化 Figure 4. Community composition and diversity changes of the total soil microbiome in paddy soils before and after high concentration methane incubation on the basis of 16S rRNA gene analysis. A: Phyla detected in soil DNA, cell DNA and single cell DNA; B: Rarefaction curves of OTU number; C: Principal coordinate analysis (PCoA) of bacterial communities from soil DNA, cell DNA and single cell DNA. |
针对Day 17的样品分析表明,水稻土好氧甲烷氧化过程在一定程度上也可能影响门水平的微生物类群。在土壤DNA中,Proteobacteria门相对丰度从Day 0的27.5%增至Day 17的54.5%;而在细胞DNA中,则从50.6%增至66.0%。此外,Actinobacteria门在土壤DNA中从Day 0的16.6%降至Day 17的7.7%;在细胞DNA中从20.4%降低至9.3% (图 4-A)。基于单细胞DNA的测序结果未能展示这一趋势。
但值得注意的是,在微生物分类学种的水平(DNA序列相似度大于97%),土壤DNA、细胞DNA及单细胞DNA的高通量测序结果差异较大。经过测序深度的归一化后,发现土壤DNA的微生物多样性指数最高,而细胞DNA中的微生物多样性指数明显降低,单细胞DNA中的微生物多样性最低(图 4-B),这一结果极可能源于分离的单细胞数量过少(<200个)。基于OTU0.03组成相似性,PCoA分析进一步展示了不同研究方法获得的微生物群落结构间的差异,所有样品按照3种方法聚成3个明显分离的簇(weighted unifrac,P < 0.005),表明在种的水平,土壤DNA、细胞DNA和单细胞DNA中微生物组成差异较大,本研究采取的细胞提取策略及单细胞分选技术具有明显的偏好性(图 4-C)。
2.5 基于高通量测序的土壤好氧甲烷氧化菌组成分析高通量测序水稻土微生物组16S rRNA基因后,通过筛选好氧甲烷氧化菌的16S rRNA序列并在科和属的水平计算不同甲烷氧化菌的相对丰度,比较了水稻土氧化甲烷过程中甲烷氧化菌的组成变化规律。如图 5-A所示,水稻土中检测到的所有好氧甲烷氧化菌可分为Type Ⅰ和Type Ⅱ两个类型,分别对应Methylococcaceae和Methylocystaceae两个科。在土壤DNA中,Type Ⅰ和Type Ⅱ在Day 0的相对百分比分别为0.7%和0.3%;而在Day 17的相对百分比分别为18.4%和1.0%,增幅为25.3倍和2.3倍。类似的,在细胞DNA中,Type Ⅰ的百分比从2.0%增加至35.2%,增幅达16.6倍,而Type Ⅱ甲烷氧化菌则从0.9%增加至4.9%,增幅约为4.4倍。这一结果和基于CARD-FISH显微计数基本一致(图 3-C, D)。单细胞DNA测序结果未检测到足量的甲烷氧化菌16S rRNA序列,因此未能反映这一趋势。

|
| 图 5. 高浓度甲烷富集前后甲烷氧化菌在整体微生物中的相对含量(A)及群落组成变化(B) Figure 5. Changes of relative abundance of methanotrophs in total microbiome (A) and methanotroph community composition (B) before and after high concentration methane incubation. |
进一步在属的水平分析好氧甲烷氧化菌的组成。在土壤DNA中,Day 0样品中的优势种为一些未分类甲烷氧化菌(Unclassified type Ⅰ or type Ⅱ),占比近70%,高浓度甲烷富集后Day 17样品中优势种转变为Type Ⅰ中的Methylobacter和Methylosarcina,占比分别达37.4%和34.1%。而在细胞DNA中,Day 0样品中的优势种为Type Ⅰ中的Methylobacter,占比为54.4%,在Day 17样品中优势种仍为Methylobacter,占比增加到80.3%,而Methylosarcina的占比仅为1.6%。单细胞DNA中也检测到了5种不同的甲烷氧化菌16S rRNA基因型,但Day 0和Day 17样品中的优势种没有发生变化,均为Methylobacter,占比分别为66.2%和61.3% (图 5-B)。
通过分析pmoA基因高通量测序数据,在微生物分类学种的水平(pmoA基因相似度大于90%),研究了高浓度甲烷培养前后土壤DNA、细胞DNA、单细胞DNA中好氧甲烷氧化菌的多样性变化及群落结构聚类特征。如图 6-A所示,尽管Day 0的土壤DNA、细胞DNA、单细胞DNA中pmoA基因多样性不尽相同,但均明显高于Day 17,表明高浓度甲烷氧化过程中部分种群快速增加并导致整体多样性降低。进一步的群落结构聚类分析表明,与Day 0相比,水稻土氧化高浓度甲烷后(Day 17),土壤DNA、细胞DNA中的甲烷氧化菌的物种组成也发生了明显的分异(weighted unifrac,P < 0.005),而在单细胞DNA中,没有检测到甲烷氧化菌在高浓度甲烷富集前后的群落结构分异趋势(图 6-B),这和图 5-B基于16S rRNA基因的测序结果一致。

|
| 图 6. 高浓度甲烷培养前后土壤DNA、细胞DNA及单细胞DNA中甲烷氧化菌群落pmoA基因多样性及组成变化 Figure 6. Diversity and genotype composition of methanotrophs in soil DNA, cell DNA and single cell DNA based on MiSeq sequencing of pmoA gene. A: Rarefaction curves of OTU number; B: Principal coordinate analysis (PCoA) of methanotroph communities in soil DNA, cell DNA and single cell DNA; C: Methanotroph genus or pmoA genotypes detected in soil DNA, cell DNA and single cell DNA. |
进一步在甲烷氧化菌属的水平,通过测序土壤DNA、细胞DNA、单细胞DNA中的pmoA基因发现7个甲烷氧化菌属,并且各个属在水稻土甲烷氧化过程中的变化规律并不一致(图 6-C)。在Day 0土壤DNA中,RPCs是优势的Type Ⅰ甲烷氧化菌,其相对丰度为22.3%,次优势属为Methylosarcina和Methylobacter,相对丰度分别为12.1%和11.6%。而Methylocystis则是优势的Type Ⅱ甲烷氧化菌,其相对丰度为36.4%。在Day 0细胞DNA中,Type Ⅱ则成为优势类群,其中Methylocystis-Methylosinus、Methylocystis、Methylosinus的相对丰度分别为32.4%、19.4%和13.9%。在水稻土氧化高浓度甲烷后,Day 17土壤DNA中Methylosarcina、Methylobacter的百分比大幅增加至58.6%和25.6%,而在细胞DNA中,仅Methylobacter发生显著增加,从Day 0的0.1%增至Day 17的69.2%。单细胞DNA的测序结果则表明,在水稻甲烷氧化过程中,Type Ⅰ中的Methylosarcina和RPCs属发生少量增加,而Type Ⅱ中尚未有明确分类地位的甲烷氧化菌属也有少量增加。
在甲烷氧化菌科的水平,基于pmoA基因和基于16S rRNA基因的高通量测序结果非常一致,均表明在高浓度甲烷富集后Type Ⅰ甲烷氧化菌相对含量大幅增加,并且土壤DNA和细胞DNA中检测到一致的甲烷氧化菌变化趋势。在土壤DNA中,Type Ⅰ甲烷氧化菌相对百分比从Day 0的53.3%增至Day 17的89.8%,增幅约为68%;而在细胞DNA中,则从24.8%增至71.8%,增幅高达190%。相反,与Day 0相比,土壤DNA和细胞DNA中Type Ⅱ甲烷氧化细菌相对百分比在Day 17均明显降低。单细胞DNA的pmoA测序结果也表现出了类似的趋势(图 6-C)。
3 讨论核酸DNA是分子方法研究土壤微生物组的核心,而传统方法则聚焦于土壤中可提取的微生物细胞。据此,我们针对土壤DNA、土壤中可提取微生物细胞(土壤细胞)和土壤细胞DNA (细胞DNA),采用qPCR方法、显微计数方法(DAPI染色、CARD-FISH杂交)及高通量测序PCR产物方法,比较分析了不同方法研究土壤微生物组的特点。结果表明,不同方法获得的土壤微生物数量范围变化较大,在107–1010 cells/g之间,DAPI染色和CARD-FISH杂交的计数结果最低,而qPCR定量土壤DNA的结果最高,相差最高可达3个数量级。这些结果表明,不同方法研究土壤微生物组数量的结果差异显著,不具备可比性。进一步的高通量测序结果也发现,尽管在微生物分类学门的水平,土壤DNA、细胞DNA、单细胞DNA中的主要类群基本一致,但在种的分类水平,土壤微生物组群落结构分异明显。然而值得注意的是,尽管传统方法和分子技术的检测分辨率具有明显差异,但均能较好地表征水稻土甲烷氧化过程的好氧甲烷氧化菌变化规律。需要特别指出的是,本研究仅针对一种水稻土开展研究,并且主要的研究对象为好氧甲烷氧化菌这一类功能微生物类群。因此,研究结果对旱地等不同类型土壤及其他功能微生物类群的普适性仍待更多试验验证。
实时荧光定量PCR的结果明显高于传统微生物计数法,相差在1–3个数量级之间。然而,基于土壤DNA的qPCR方法极可能高估微生物数量。主要原因包括:(1) 土壤中胞外DNA干扰。这些胞外DNA可能是活体微生物细胞分泌物,也可能来自于死亡细胞裂解后的游离DNA[25-26]。据估算,每克土壤中附着于土壤颗粒或游离的胞外DNA含量可能高达1.9 μg[27],而本研究中每克水稻土总DNA的提取量约在4–6 μg之间,导致土壤微生物量被高估可能达1倍之多。(2) 不同微生物16S rRNA基因拷贝数差异巨大。根据对已知2013个微生物基因组(130个古菌、1883个细菌)的分析,发现单个细菌细胞中16S rRNA基因拷贝变幅为1–15,而古菌的变幅为1–4,并且超过80%已知细菌种类含有2个以上的16S rRNA基因拷贝[28]。此外,已知的好氧甲烷氧化菌细胞通常含有2–3个pmoA基因拷贝[29-30]。因此,基于16S rRNA或其它靶标基因的qPCR极可能高估土壤中的微生物细胞数量。据此,假设每个微生物细胞含有6个16S rRNA基因拷贝,每个好氧甲烷氧化菌含有2个pmoA基因拷贝,基于土壤DNA qPCR的微生物数量约为1.5×1010–2.7×1010 cells/g,基于细胞DNA qPCR的土壤总微生物数量约2.8×108– 6.3×108 cells/g,而基于DAPI染色和CARD-FISH的微生物数量约为1.7×107–7.4×107 cells/g。针对好氧甲烷氧化菌,土壤DNA qPCR的数量约为1.1×107–6.0×108 cells/g,细胞DNA qPCR的数量约为9.0×104–3.5×107 cells/g,而CARD-FISH计数范围则为3.7×104–1.7×106 cells/g,不同方法间的差异最高达到3个数量级。
与qPCR相比,基于细胞提取的显微计数极可能低估土壤微生物的数量。这种低估与土壤微生物细胞提取效率紧密相关,并受很多因素影响,主要包括微生物细胞本身、物理和化学分散策略、土壤类型等。(1) 微生物细胞类群。细胞形态、活性及代谢方式均可能影响土壤微生物细胞提取[31],其中细胞大小可能是最重要的因素。微生物细胞直径范围跨度可达0.2–750.0 μm[32],而古菌细胞更小,与细菌细胞差异可达10倍之多。最近的研究采用不同滤膜分选土壤中的微生物细胞,表明59%–67%的细胞直径均小于1.2 μm,细胞直径越小,其中的难培养微生物包括古菌比例越高,两者之间具有较好的相关性[33]。(2) 物理干扰因子。土壤中不同种类的微生物细胞提取效率具有一定的差异,这可能是由于不同种类微生物细胞与土壤颗粒的粘附程度不同导致的。相当数量的微生物细胞可能与土壤颗粒紧密粘附在一起。因此,首先需要通过超声波、物理匀浆、机械振荡以及化学分散剂等方式将微生物细胞和土壤颗粒分离[34],随后利用差速离心、密度梯度离心或者两相分离技术等将微生物细胞与土壤颗粒分开,而不同的分离方式会对土壤微生物细胞提取造成不同的影响。例如,已有研究表明超声波对细胞损伤最大,而物理旋转搅拌影响较小,并与机械强度和作用时间紧密相关[35]。(3) 化学干扰因子。不同的缓冲溶液配比及不同的离心介质具有完全不同的细胞分离效果[36],如Nycodenz作为离心介质,较之蔗糖或Percoll等其他介质,可以获得纯度更高的微生物细胞,但其损失较大,提取效率较低[37]。(4) 土壤类型影响[38-39]。据估算,地球上高级分类单元的土系高达22000类,这些土壤起源自不同的母质、地形、气候、时间和生物作用,形成了极为复杂的历史自然体。例如,花岗岩、玄武岩和石灰岩等母质在不同地理气候风化条件下,会产生物理化学性质各异的土壤黏粒,这些带有不同电荷的蒙脱石、高岭石等矿物胶体最终形成了各种粒径的土壤团聚体,构成了多相疏松多孔隙土壤环境维持微生物的生长[40]。未来需要发展物理-化学-生物的原位综合观测技术,在不损失活体细胞条件下,尽量消除微生物与土壤颗粒之间相互作用,发展高效的土壤微生物细胞分离技术和评价体系。
高通量测序16S rRNA基因清楚表明土壤微生物细胞提取过程中存在明显的偏差,特别是土壤DNA中Acidobacteria门类微生物占比高达12.2%,但在细胞DNA中该门类微生物仅占1.1%。这一结果与最近的瑞典苔原草地土壤结果类似,土壤DNA中Acidobacteria门含量可达50%左右,而CARD-FISH特异探针杂交则表明Acidobacteria门的相对丰度仅为10%左右[41]。Acidobacteria微生物难以被提取的主要原因可能是其细胞较小,直径通常小于0.8 μm,与土壤颗粒结合比较紧密,常规的涡旋方法难以使其与土壤颗粒充分分离,导致大部分Acidobacteria门细胞随着土壤颗粒离心到离心管底部而远离细胞层[33]。同时,相当数量的Acidobacteria门微生物能够分泌粘性的胞外多糖复合物,增加了细胞和土壤颗粒间的粘附力,降低了该门类细胞的提取效率[42]。此外,Planctomycetes门在土壤DNA中仅占3.1%,但在细胞DNA中比例上升至19.6%,因此,也可能是Planctomycetes类群微生物更加容易提取,间接导致Acidobacteria微生物的比例变低。值得注意的是,细胞DNA中未分类的微生物较少,其原因可能是很多难培养微生物直径小于0.2 μm[43],而这些微小的微生物可能被土壤团聚体包裹而难以被分离。
尽管采用传统显微计数和现代分子方法研究水稻土微生物组的结果具有明显差异,但不同方法均能较好反映好氧甲烷氧化过程的微生物生理生态特征。例如,基于可提取细胞的CARD-FISH甲烷氧化菌计数仅为104 cells/g,而基于土壤DNA的数量高达107 cells/g,两者相差可达数千倍。然而,水稻土甲烷氧化过程中,CARD-FISH计数、土壤可提取细胞DNA的qPCR定量以及土壤DNA定量分析,均表明好氧甲烷氧化菌明显增长,增幅分别为54倍(土壤DNA)、388倍(细胞DNA)和45倍(CARD-FISH)。16S rRNA和pmoA基因测序均表明:高浓度甲烷富集后Type Ⅰ甲烷氧化菌成为甲烷氧化菌中的优势类群,并且这一规律在土壤DNA及细胞DNA中非常一致。因此,至少在微生物分类科的水平,基于细胞DNA的高通量测序结果可以完全反映甲烷氧化菌对高浓度甲烷刺激的响应规律。然而,在甲烷氧化菌各个属中,Methylosarcina在细胞DNA中的丰度远低于其在土壤DNA中的丰度,表明其提取效率明显低于其他甲烷氧化菌种类。Methylosarcina属的细胞常呈现不规则形,并且细胞表面分布有纤毛状结构或者致密的粘液层,能将多个细胞粘附在一起形成团块,而其他种类的甲烷氧化菌则不具备这些特征[44]。因此,这些形态及生理上的特异性可能导致了Methylosarcina属细胞与土壤颗粒结合非常紧密,降低了其细胞提取效率。不过对于整体甲烷氧化菌来说,特别是水稻土氧化了高浓度甲烷后,甲烷氧化菌在细胞DNA中比例(40.1%)要远大于在土壤DNA中的比例(19.4%)(图 5-A),表明大部分新产生的甲烷氧化菌细胞和土壤颗粒的结合并不紧密,比较容易通过常规的涡旋处理与土壤颗粒分离。事实上,水稻土好氧甲烷氧过程中的甲烷氧化菌细胞提取效率从0.9%增加至6.1%,明显高于整体微生物细胞提取效率(2.0%–2.4%),与已有报道较为一致[45-46]。
单细胞是未来土壤微生物组研究的重要内容,而单细胞的研究取决于土壤微生物细胞的提取效率。假设土壤DNA的16S rRNA基因序列能够代表真实的土壤微生物物种组成,那么基于土壤DNA和细胞DNA的高通量测序结果表明:土壤DNA共计检测到30个微生物门,而细胞DNA中检测到25个门,其中细胞DNA未覆盖到的5个门类在本研究水稻土中丰度极低,总占比不到0.14%。因此,本研究获得的细胞DNA可以比较真实地反映水稻土中的微生物组成。然而,在更精细的分类水平(图 4-B,图 6-A),细胞DNA和土壤DNA群落结构具有明显差异,后续的单细胞随机分选则可能产生进一步的偏差。单细胞16S rRNA基因分析表明绝大部分微生物属于Proteobacteria门,偏好性非常明显。同时,尽管细胞DNA中甲烷氧化菌占比已经高达40.1% (图 5-A),但单细胞分选所得到的甲烷氧化菌细胞也较少,这可能与流式细胞仪分选区域的选择及单细胞全基因组MDA扩增的特征有关。流式细胞仪分选时同时考虑细胞大小、荧光强弱、是否单细胞等多方面因素。选定区域不同,得到的细胞类群也就不同。本研究随机选取荧光信号较强的细胞分布区域作为分选目标,由于高浓度甲烷富集后,甲烷氧化菌的活性最强,理论上荧光强的区域可能包含较多的甲烷氧化菌。但是,MDA扩增法本身覆盖度在40%–70%左右,并且对16S rRNA基因的扩增覆盖度只有约1/3左右[47]。因此,MDA扩增后的产物(单细胞DNA)进行16S rRNA基因高通量测序会在很大程度上低估菌群的多样性,并可能导致较明显的物种偏好。从实验结果来看,MDA产物的pmoA功能基因测序与土壤DNA高通量测序结果更为接近,说明通过检验功能基因可以更准确地判定所分离的单细胞是否为目标微生物。这些结果为未来的微生物单细胞分选及鉴定提供了参考。
4 结论直接提取土壤中的微生物总DNA或者微生物细胞,是分子方法和显微计数研究土壤微生物组的核心。我们的结果表明,基于土壤DNA的qPCR得到的水稻土微生物组数量最高,比传统DAPI染色和CARD-FISH杂交计数结果高2–3个数量级。土壤中的胞外DNA使得qPCR定量结果偏高,而微生物细胞粘附于土壤颗粒导致细胞提取效率低,使得传统显微计数结果偏低。因此,水稻土微生物的实际数量应处于2种结果之间。尽管2种方法的检测精度差异显著,但无论是传统显微计数方法还是现代分子定量方法,都表明水稻土好氧甲烷氧化过程中甲烷氧化菌数量显著增加,特别是基于细胞DNA的qPCR结果表明甲烷氧化菌增幅高达388倍。高通量测序16S rRNA和pmoA基因PCR扩增产物的结果表明,基于土壤DNA、细胞DNA和单细胞DNA的高通量测序结果检测到了较一致的微生物种类,并且都表明高浓度甲烷氧化后,Methylobacter/Methylosarcina成为优势甲烷氧化菌,主导了水稻土高浓度甲烷的氧化。Planctomycetes微生物门的细胞易被提取,而Acidobacteria门的微生物则较难被提取,单细胞筛选过程中则选择性偏好Proteobacteria门的微生物细胞。总体而言,尽管传统显微计数方法和现代分子生态学技术在土壤微生物组研究方面的分辨率具有显著差异,但均能较好表征水稻土好氧甲烷氧化的微生物生理生态过程,未来土壤微生物组研究中,应更加重视科学问题本身对技术手段的内在需求,结合显微观察的直观可视优点,更好地发挥分子技术的高分辨率特点,为在单细胞、种群、群落、土体、田块和流域等不同尺度耦合分析土壤微生物组的演化规律及其环境功能提供技术支撑。
| [1] | Young IM, Crawford JW. Interactions and self-organization in the soil-microbe complex. Science, 2004, 304(5677): 1634–1637 DOI:10.1126/science.1097394. |
| [2] | Woese CR, Fox GE. Phylogenetic structure of the prokaryotic domain:the primary kingdoms. Proceedings of the National Academy of Sciences of the United States of America, 1977, 74(11): 5088–5090 DOI:10.1073/pnas.74.11.5088. |
| [3] | Cole JR, Wang Q, Cardenas E, Fish J, Chai B, Farris RJ, Kulam-Syed-Mohideen AS, McGarrell DM, Marsh T, Garrity GM, Tiedje JM. The ribosomal database project:improved alignments and new tools for rRNA analysis. Nucleic Acids Research, 2009, 37(S1): D141–D145 |
| [4] | Amann RI. Fluorescently labelled, rRNA-targeted oligonucleotide probes in the study of microbial ecology. Molecular Ecology, 1995, 4(5): 543–554 DOI:10.1111/j.1365-294X.1995.tb00255.x. |
| [5] | Stahl DA. Application of phylogenetically based hybridization probes to microbial ecology. Molecular Ecology, 1995, 4(5): 535–542 DOI:10.1111/j.1365-294X.1995.tb00254.x. |
| [6] | Haas CN. Estimation of microbial densities from dilution count experiments. Applied and Environmental Microbiology, 1989, 55(8): 1934–1942 |
| [7] | McCrady MH. The numerical interpretation of fermentation-tube results. The Journal of Infectious Diseases, 1915, 17(1): 183–212 DOI:10.1093/infdis/17.1.183. |
| [8] | Olsen GJ, Lane DJ, Giovannoni SJ, Pace NR, Stahl DA. Microbial ecology and evolution:a ribosomal RNA Approach. Annual Review of Microbiology, 1986, 40: 337–365 DOI:10.1146/annurev.mi.40.100186.002005. |
| [9] | Fuhrman JA, McCallum K, Davis AA. Novel major archaebacterial group from marine plankton. Nature, 1992, 356(6365): 148–149 DOI:10.1038/356148a0. |
| [10] | Okano Y, Hristova KR, Leutenegger CM, Jackson LE, Denison RF, Gebreyesus B, Lebauer D, Scow KM. Application of real-time PCR to study effects of ammonium on population size of ammonia-oxidizing bacteria in soil. Applied and Environmental Microbiology, 2004, 70(2): 1008–1016 DOI:10.1128/AEM.70.2.1008-1016.2004. |
| [11] | Muyzer G, de Waal EC, Uitterlinden AG. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA. Applied and Environmental Microbiology, 1993, 59(3): 695–700 |
| [12] | Mazutis L, Gilbert J, Ung WL, Weitz DA, Griffiths AD, Heyman JA. Single-cell analysis and sorting using droplet-based microfluidics. Nature Protocols, 2013, 8(5): 870–891 DOI:10.1038/nprot.2013.046. |
| [13] | Zheng Y, Huang R, Wang BZ, Bodelier PLE, Jia ZJ. Competitive interactions between methane-and ammonia-oxidizing bacteria modulate carbon and nitrogen cycling in paddy soil. Biogeosciences, 2014, 11(12): 3353–3368 DOI:10.5194/bg-11-3353-2014. |
| [14] | Lindahl V, Bakken LR. Evaluation of methods for extraction of bacteria from soil. FEMS Microbiology Ecology, 1995, 16(2): 135–142 DOI:10.1111/fem.1995.16.issue-2. |
| [15] | Daims H, Brühl A, Amann R, Schleifer KH, Wagner M. The domain-specific probe EUB338 is insufficient for the detection of all Bacteria:development and evaluation of a more comprehensive probe set. Systematic and Applied Microbiology, 1999, 22(3): 434–444 DOI:10.1016/S0723-2020(99)80053-8. |
| [16] | Eller G, Stubner S, Frenzel P. Group-specific 16S rRNA targeted probes for the detection of type Ⅰ and type Ⅱ methanotrophs by fluorescence in situ hybridisation. FEMS Microbiology Letters, 2001, 198(2): 91–97 DOI:10.1111/fml.2001.198.issue-2. |
| [17] | Stubner S. Enumeration of 16S rDNA of Desulfotomaculum lineage 1 in rice field soil by real-time PCR with SybrGreenTM detection. Journal of Microbiological Methods, 2002, 50(2): 155–164 DOI:10.1016/S0167-7012(02)00024-6. |
| [18] | Costello AM, Lidstrom ME. Molecular characterization of functional and phylogenetic genes from natural populations of methanotrophs in lake sediments. Applied and Environmental Microbiology, 1999, 65(11): 5066–5074 |
| [19] | Holmes AJ, Costello A, Lidstrom ME, Murrell JC. Evidence that participate methane monooxygenase and ammonia monooxygenase may be evolutionarily related. FEMS Microbiology Letters, 1995, 132(3): 203–208 DOI:10.1111/fml.1995.132.issue-3. |
| [20] |
Du WB, Xu P, Dong LB. Micro-pipeline-based liquid droplet generation method. China:CN201410655191.5. 2014-11-17(in Chinese)
杜文斌, 徐鹏, 董立兵. 基于微管道的液滴的生成方法. 中国: CN201410655191. 5. 2014-11-17. |
| [21] | Xu P, Zheng X, Tao Y, Du WB. Cross-interface emulsification for generating size-tunable droplets. Analytical Chemistry, 2016, 88(6): 3171–3177 DOI:10.1021/acs.analchem.5b04510. |
| [22] | Schloss PD, Westcott SL, Ryabin T, Hall JR, Hartmann M, Hollister EB, Lesniewski RA, Oakley BB, Parks DH, Robinson CJ, Sahl JW, Stres B, Thallinger GG, Van Horn DJ, Weber CF. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 2009, 75(23): 7537–7541 DOI:10.1128/AEM.01541-09. |
| [23] | Dumont MG, Lüke C, Deng YC, Frenzel P. Classification of pmoA amplicon pyrosequences using BLAST and the lowest common ancestor method in MEGAN. Frontiers in Microbiology, 2014, 5: 34 |
| [24] | Schloss PD, Gevers D, Westcott SL. Reducing the effects of PCR amplification and sequencing artifacts on 16S rRNA-based studies. PLoS One, 2011, 6(12): e27310 |
| [25] | Blum SAE, Lorenz MG, Wackernagel W. Mechanism of retarded DNA degradation and prokaryotic origin of DNases in nonsterile soils. Systematic and Applied Microbiology, 1997, 20(4): 513–521 DOI:10.1016/S0723-2020(97)80021-5. |
| [26] | Carini P, Marsden PJ, Leff JW, Morgan EE, Strickland MS, Fierer N. Relic DNA is abundant in soil and obscures estimates of soil microbial diversity. Nature Microbiology, 2016, 2: 16242 DOI:10.1038/nmicrobiol.2016.242. |
| [27] | Niemeyer J, Gessler F. Determination of free DNA in soils. Journal of Plant Nutrition and Soil Science, 2002, 165(2): 121–124 |
| [28] | Sun DL, Jiang X, Wu QL, Zhou NY. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity. Applied and Environmental Microbiology, 2013, 79(19): 5962–5969 DOI:10.1128/AEM.01282-13. |
| [29] | Gilbert B, McDonald IR, Finch R, Stafford GP, Nielsen AK, Murrell JC. Molecular analysis of the pmo (particulate methane monooxygenase) operons from two type Ⅱ methanotrophs. Applied and Environmental Microbiology, 2000, 66(3): 966–975 DOI:10.1128/AEM.66.3.966-975.2000. |
| [30] | Yimga MT, Dunfield PF, Ricke P, Heyer J, Liesack W. Wide distribution of a novel pmoA-like gene copy among type Ⅱ methanotrophs, and its expression in Methylocystis strain SC2. Applied and Environmental Microbiology, 2003, 69(9): 5593–5602 DOI:10.1128/AEM.69.9.5593-5602.2003. |
| [31] | Bakken LR, Olsen RA. DNA-content of soil bacteria of different cell size. Soil Biology and Biochemistry, 1989, 21(6): 789–793 DOI:10.1016/0038-0717(89)90172-7. |
| [32] | Schulz HN, Jørgensen BB. Big bacteria. Annual Review of Microbiology, 2001, 55: 105–137 DOI:10.1146/annurev.micro.55.1.105. |
| [33] | Portillo MC, Leff JW, Lauber CL, Fierer N. Cell size distributions of soil bacterial and archaeal taxa. Applied and Environmental Microbiology, 2013, 79(24): 7610–7617 DOI:10.1128/AEM.02710-13. |
| [34] | Berry AE, Chiocchini C, Selby T, Sosio M, Wellington EMH. Isolation of high molecular weight DNA from soil for cloning into BAC vectors. FEMS Microbiology Letters, 2003, 223(1): 15–20 DOI:10.1016/S0378-1097(03)00248-9. |
| [35] | Ehlers K, Bünemann EK, Oberson A, Frossard E, Frostegård Å, Yuejian M, Bakken LR. Extraction of soil bacteria from a Ferralsol. Soil Biology and Biochemistry, 2008, 40(7): 1940–1946 DOI:10.1016/j.soilbio.2008.04.005. |
| [36] | Lindahl V. Improved soil dispersion procedures for total bacterial counts, extraction of indigenous bacteria and cell survival. Journal of Microbiological Methods, 1996, 25(3): 279–286 DOI:10.1016/0167-7012(95)00102-6. |
| [37] | Eichorst SA, Strasser F, Woyke T, Schintlmeister A, Wagner M, Woebken D. Advancements in the application of NanoSIMS and Raman microspectroscopy to investigate the activity of microbial cells in soils. FEMS Microbiology Ecology, 2015, 91(10): fiv106 DOI:10.1093/femsec/fiv106. |
| [38] | Lombard N, Prestat E, van Elsas JD, Simonet P. Soil-specific limitations for access and analysis of soil microbial communities by metagenomics. FEMS Microbiology Ecology, 2011, 78(1): 31–49 DOI:10.1111/j.1574-6941.2011.01140.x. |
| [39] | Maron PA, Schimann H, Ranjard L, Brothier E, Domenach AM, Lensi R, Nazaret S. Evaluation of quantitative and qualitative recovery of bacterial communities from different soil types by density gradient centrifugation. European Journal of Soil Biology, 2006, 42(2): 65–73 DOI:10.1016/j.ejsobi.2005.08.003. |
| [40] | 熊毅, 李庆逵. 中国土壤. 第2版. 北京: 科学出版社, 1987. |
| [41] | Ushio M, Makoto K, Klaminder J, Takasu H, Nakano SI. High-throughput sequencing shows inconsistent results with a microscope-based analysis of the soil prokaryotic community. Soil Biology and Biochemistry, 2014, 76: 53–56 DOI:10.1016/j.soilbio.2014.05.010. |
| [42] | Eichorst SA, Breznak JA, Schmidt TM. Isolation and characterization of soil bacteria that define Terriglobus gen. nov., in the phylum Acidobacteria. Applied and Environmental Microbiology, 2007, 73(8): 2708–2717 DOI:10.1128/AEM.02140-06. |
| [43] | Hahn MW. Broad diversity of viable bacteria in 'sterile'(0.2μm) filtered water. Research in Microbiology, 2004, 155(8): 688–691 DOI:10.1016/j.resmic.2004.05.003. |
| [44] | Wise MG, McArthur JV, Shimkets LJ. Methylosarcina fibrata gen. nov., sp. nov. and Methylosarcina quisquiliarum sp.nov., novel type 1 methanotrophs. International Journal of Systematic and Evolutionary Microbiology, 2001, 51(2): 611–621 DOI:10.1099/00207713-51-2-611. |
| [45] | Bertrand H, Poly F, Van VT, Lombard N, Nalin R, Vogel TM, Simonet P. High molecular weight DNA recovery from soils prerequisite for biotechnological metagenomic library construction. Journal of Microbiological Methods, 2005, 62(1): 1–11 DOI:10.1016/j.mimet.2005.01.003. |
| [46] | Neveu M, Poret-Peterson AT, Lee ZMP, Anbar AD, Elser JJ. Prokaryotic cells separated from sediments are suitable for elemental composition analysis. Limnology and Oceanography:Methods, 2014, 12(7): 519–529 DOI:10.4319/lom.2014.12.519. |
| [47] | Rinke C, Lee J, Nath N, Goudeau D, Thompson B, Poulton N, Dmitrieff E, Malmstrom R, Stepanauskas R, Woyke T. Obtaining genomes from uncultivated environmental microorganisms using FACS-based single-cell genomics. Nature Protocols, 2014, 9(5): 1038–1048 DOI:10.1038/nprot.2014.067. |
 2017, Vol. 57
2017, Vol. 57