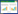中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 张慧敏, Priemé Anders, Faucherre Samuel, Elberling Bo, 贾仲君. 2017
- Huimin Zhang, Priemé Anders, Faucherre Samuel, Elberling Bo, Zhongjun Jia. 2017
- 北极冻土区活跃层与永冻层土壤微生物组的空间分异
- Soil microbiomes modulate distinct patterns of soil respiration and methane oxidation in arctic active layer and permafrost
- 微生物学报, 57(6): 839-855
- Acta Microbiologica Sinica, 57(6): 839-855
-
文章历史
- 收稿日期:2017-02-13
- 修回日期:2017-04-07
- 网络出版日期:2017-04-11
2. 中国科学院南京土壤研究所, 土壤与农业可持续发展国家重点实验室, 江苏 南京 210008;
3. 中国科学院大学, 北京 100049;
4. Center for Permafrost (CENPERM), Department of Geosciences and Natural Resource Management, University of Copenhagen, Øster Voldgade 10, 1350 Copenhagen K, Denmark;
5. Department of Biology, University of Copenhagen, Øster Voldgade 10, 1350 Copenhagen K, Denmark
2. State Key Laboratory of Soil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, Jiangsu Province, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Center for Permafrost(CENPERM), Department of Geosciences and Natural Resource Management, University of Copenhagen, Øster Voldgade 10, 1350 Copenhagen K, Denmark;
5. Department of Biology, University of Copenhagen, Øster Voldgade 10, 1350 Copenhagen K, Denmark
Zhongjun Jia, Tel: +86-25-86881311, Fax: +86-25-86881000, E-mail: jia@issas.ac.cn
永冻土占北半球陆地面积约25%,被认为是对环境变化响应最显著的生态系统之一。永冻土上层通常会发生季节性冻融,其厚度随季节而变化,变幅因地区而异并可达几十倍,从几厘米到200 cm甚至更厚。因此,永冻土表层发生季节性融解的土层常被称为活跃层,而埋藏于活跃层下方长期处于0 ℃以下且至少保持2年以上不融化的冻土则被称为永冻层,其深度可达1500 m,内含物包括土壤、沉积物或岩石层。北冰洋沿岸是地球永冻土分布最为集中的地区,包括欧亚大陆北部、北美大陆北部以及北冰洋的许多岛屿,永冻土面积高达7.2×106 km2[1]。研究表明冻土生态系统对气候变化异常敏感,其气候变暖速度高于地球平均水平,相关长期监测发现部分永冻土地区的活跃层以每年0.7-1.3 cm的速度变厚,多个测点的永冻层消失[2]。全球变暖情景下,永冻层转化为活跃层并伴随着复杂的相态变化,从单一固相的永冻层逐步转化为气、液、固三相的复杂活跃层,其中水分、温度和有机质等要素变化极可能改变其中关键元素的生物地球化学循环,永冻土相关的地质生物学已成为全球变化的研究热点[3]。
全球冻土区碳储量巨大,是气候变化的重要调节阀。据估算,永冻土中碳储量高达16720亿吨,相当于大气与地上植物碳的总和,并且其中绝大部分碳储量位于冻土区季节性融解的活跃层0-3 m深处,约为10240亿吨[4]。有研究表明,随着气候变暖,原先的永冻层不再“永冻”并转化为季节性融化的新活跃层,从而导致其中固定的碳源被微生物分解,通过好氧土壤呼吸产生CO2或者厌氧微生物作用排放甲烷,从而加剧温室效应,形成全球变暖正反馈[5]。此外,气温升高导致永冻土变为活跃土后,原先闭蓄于永冻土中的甲烷和二氧化碳也可能通过物理扩散方式进入大气,进一步加剧温室效应。然而,目前冻土区碳排放通量的估算通常未考虑空间异质性,特别是活跃层和永冻层的垂直梯度差异,导致模型预测结果存在较大的不确定性,例如,有研究表明2040年冻土区的碳排放量可以达到190-450亿吨CO2-C,2.6-8.5亿吨CH4-C,变幅可达3倍之多[4]。因此,在全球变化估算模型中耦合微生物过程模型,量化冻土区活跃层和永冻层的相对贡献及其时空变异规律具有重要意义。
冻土中的微生物群落在土壤碳转化过程中发挥了极为重要的作用。夏季高温期间,活跃层融解并通过土壤微生物呼吸释放大量的CO2,其强度变化受到环境条件如水分、氧气的较大影响。此外,季节性冻融的表层活跃土及深层永久冻土极可能栖息着不同的微生物,导致活跃层和永冻层土壤呼吸强度具有较大的差异。因此,深入解析活跃层和永冻层微生物群落的演替规律及其环境驱动机制,有利于准确预测冻土区温室气体源和汇的强度[6]。但长期以来理论与技术发展滞后并制约了对土壤微生物包括冻土区微生物生态过程的深入认识。近年来以高通量测序为代表的分子方法发展迅猛,根本上改变了土壤真菌、细菌和放线菌的传统区系研究理念,为研究冻土区微生物过程及其生态意义提供了重要支撑。最近,Schostag (2015) 等研究了挪威斯瓦尔巴群岛季节性融解活跃层的微生物年际变化规律,发现活跃层的微生物在夏季和冬季的数量无显著变化,但群落组成发生了明显分异,其中alpha-Proteobacteria变化最为明显,在整个夏季中相对百分比逐渐增加,而在10月到11月秋冬季呈降低趋势。研究同时发现pH和可溶性有机碳含量可能是冻土区微生物群落演替的主控环境因子[7]。然而,由于冻土区极端的气候和环境条件,目前仅有个别样点的相关报道,这些研究能够在多大程度上反映冻土区土壤微生物群落结构和功能,仍需在不同的冻土区开展实验,获得更多的数据支持。
已有的室内模拟实验表明活跃层融解过程中微生物组多样性和功能发生了明显的变化。Mackelprang等采用宏基因组技术,发现阿拉斯加冻土区3个活跃层(35 cm以上表层)和永冻层(85 cm以下深层)的微生物呼吸和甲烷排放具有明显差异。尽管原位3个永冻层土壤微生物组之间存在一定差异,但5 ℃培养1周后这种差异逐渐变小,意味着永冻土融化后,可能发生定向的趋同演替[8]。同时,研究人员通过宏基因组测序获得了1株难培养产甲烷古菌的基因组草图,推测这些古菌能够适应寒冷条件并在冻土中产甲烷[9]。随后在温度上升冻土融解过程中,闭蓄其中的甲烷扩散到活跃层中,一部分被其中的甲烷氧化菌所消耗,其余则逃逸至大气中成为大气甲烷的重要源。最近,研究人员利用先进的多组学(蛋白组、转录组和基因组)技术,比较了原位永冻土的母质喀斯特泥沼、活跃层和永冻层中土壤微生物组的系统发育地位及其遗传潜能,揭示了具有潜在功能的微生物组过程,如厌氧产甲烷和一些特殊的永冻土功能微生物适应机制[7]。然而,不同冻土区及其中的土壤微生物组形成是一个漫长的地质历史过程并可能具有较大的差异,已有的研究大多聚焦于个别样点的原位观测及室内分析。全球变化情景下典型冻土区活跃层和永冻层土壤呼吸和甲烷氧化过程的微生物演替规律及其环境驱动机制尚未见报道。
据此,本研究针对北极冻土区相距2700 km的挪威斯瓦尔巴岛和俄罗斯西伯利亚的典型活跃层和永冻层土壤样品,在10 ℃下模拟全球变暖情景下开展室内培养实验,研究了土壤呼吸强度和好氧甲烷氧化潜力的空间变异规律,利用MiSeq测序16S rRNA基因V4-V5高可变区分析土壤微生物组的群落结构,结合稳定性同位素13C-CH4示踪好氧甲烷碳同化微生物核酸DNA,研究了典型冻土区活跃层和永冻层土壤微生物功能演替规律及其环境驱动机制,以期为预测未来气候变化下北极冻土区微生物组结构与功能变化提供科学依据。
1 材料和方法 1.1 典型冻土区活跃层和永冻层土壤样品采集如图 1-A所示,在相距2700 km的挪威斯瓦尔巴群岛(Svalbard)和俄罗斯西伯利亚勒拿河三角洲(Lena Delta)分别采样,共获得4个活跃层及4个永冻层土壤。挪威斯瓦尔巴岛的2个沉积柱分别来自Adventdalen (AD,78°11'11.6"N,15°55'11.5"E,永冻层形成于全新世,11700年前)和NyAlesund (NA,78°55'13.2"N,11°51'21.1"E,永冻层形成于全新世,约11700年前),其中AD和NA样点的活跃层厚度皆为80 cm (图 1-B)。俄罗斯西伯利亚的2个沉积柱分别来自勒拿河三角洲第一梯度Lena delta First terrace (LF,72°23'16.0"N,126°24'45.4"E,冻土形成于永冻土形成于1000多年前)和第三梯度Kurungnakh Third terrace (KU,72°17'32.0"N,126°12'46.2"E,永冻土形成于更新世,从2588000年前到11700年前),其中LF和KU样点的活跃层厚度分别为40 cm和30 cm (图 1-C)。2个采样点均为典型永冻土区域,其中挪威斯瓦尔巴特群岛采样点的地理气候条件如下:夏季最高温度可达7 ℃,冬季平均温度-12−-16 ℃。俄罗斯西伯利亚勒拿河冻土区域的地理气候条件如下:夏季最高温可达10.1 ℃,平均年温度-12.5 ℃。

|
| 图 1. 挪威斯瓦尔巴群岛和俄罗斯西伯利亚勒拿河三角洲活跃层及永冻层的土壤采样示意图 Figure 1. Location of Sampling sites and soil physiochemical properties. A: The active layer and permafrost soils are collected from four different sites; B: AD and NA stand for samples at Adventdalen and Ny-Alesund of Svalbard islands in Norway, respectively; C: KU and LF stand for soil samples from Kurungnakh Third terrace and Lena delta First terrace of Siberia in Russia, respectively; D: Soil physiochemical properties are shown in heatmap. |
样品采集时间分别为2013年6月和2013年8月,具体采样步骤如下:分别在挪威斯瓦尔巴群岛和俄罗斯西伯利亚勒拿河冻土区各自选择2个采样小区,利用直径4.8 cm、深度1.0 m的土钻,在每个采样小区内获得1个沉积柱,并切割为活跃层和永冻层两部分。本试验中选择挪威斯瓦尔巴群岛10-20 cm的活跃层、80-90 cm永冻层土壤开展后续研究,俄罗斯西伯利亚勒拿河冻土区则选择10-20 cm的活跃层、30-50 cm永冻层土壤开展后续研究。将活跃层和永冻层土壤样品放入无菌塑料袋并现场保存于-18 ℃冰箱后运送到实验室后于-20 ℃保存。4个活跃层和4个永冻层土壤理化性质如前所述[10],包括总碳、总氮、总硫、土壤密度与pH的平均值(图 1-D)。
1.2 活跃层和永冻层土壤呼吸和甲烷氧化过程冻土区土壤呼吸过程:从-20 ℃冷冻柜中取出活跃层和永冻层土壤,根据已知的土壤含水率[9],称取相当于15.0 g dws (dry weight soil)的4个活跃层样品和对应的4个永冻层样品置于100 mL血清瓶中,并在血清瓶内侧安装光化学传感芯片(PreSens,德国),通过Fibox 4检测仪(PreSens,德国)在血清瓶外侧扫描芯片并测定氧气浓度,随后采用丁基橡胶塞密封并用铝盖封口培养瓶,在10 ℃培养箱中避光培养,模拟夏季最高温度下活跃层和永冻层土壤融解过程。整个培养持续30 d并持续测定瓶内CO2和O2浓度变化。同时设置好氧甲烷氧化过程培养实验,整个实验步骤与土壤呼吸培养相似,培养瓶密封后,采用1.0 mL针管向瓶内注入0.5 mL 13CH4 (99 atom% 13C,Sigma-Aldrich),血清瓶内初始CH4浓度约为400 μmol/L (10 mL/L)。整个培养过程持续26 d,规律性测定培养瓶内CO2、O2和CH4浓度。CO2和O2浓度浓度如前所述通过传感器实时监测。CH4和N2O浓度通过气象色谱仪测定,首先从培养瓶内抽出0.5 mL顶部气体,稀释到3 mL的测气瓶(瓶内预先配有2.5 mL稀释气体,组分为氮气和氧气,比例为4:1),稀释后用气相色谱仪测定甲烷与二氧化碳浓度。每天气体样品采集后均向瓶内补充0.5 mL的合成空气平衡气体压力。每隔大约3 d采气1次,培养期间共计采气8次。根据加入甲烷后混匀培养瓶内的气体中甲烷浓度,计算培养瓶内甲烷浓度,结合甲烷浓度随培养时间的动态变化规律,计算外加高浓度甲烷消耗速率,同时结合O2浓度变化规律,计算土壤呼吸速率。土壤呼吸和甲烷氧化培养实验结束后,打开培养瓶盖并破坏性采集土壤样品,部分样品保存于-20 ℃用于DNA提取和分子分析。
1.3 活跃层和永冻层土壤微生物组的数量和组成分析 1.3.1 土壤微生物组总DNA提取: 采用PowerSoil®-DNA Isolation Kit (Mo-Bio Laboratories,Inc.)试剂盒,称取约0.25 g新鲜土样,按照说明书的提取步骤进行,将提取得到的土壤微生物总DNA溶解于100 μL无菌水后,通过微量紫外分光光度计(NanoDrop® ND-1000) 测定DNA浓度和纯度(OD600/OD280和OD260/OD230)。此外,利用1.2%的琼脂糖凝胶电泳分离5 μL的DNA (0.5×TAE缓冲液),分析DNA的完整性和相对浓度。样品包括4个活跃层和4个永冻层土壤及其对应的13C-甲烷标记和对照处理,获得足量DNA后用于后续分析。针对13C-甲烷标记处理KU活跃层土壤及对照组,额外提取3个土壤重复样品DNA用于超高速密度梯度DNA-SIP离心试验。 1.3.2 土壤微生物的数量分析: 活跃层和永冻层的土壤微生物定量PCR引物序列为515F/907R引物[11-12] (515F:5′-GTGCCAGCMGCCGCGG-3′;907R:5′-CCGTCAATTCMTTTRAGTT-3′),定量PCR标线采用16S rRNA基因通用引物515F/907R克隆进行制备。利用通用引物扩增目的基因并构建克隆文库后,将含有目标基因的克隆在LB营养液中过夜培养,利用试剂盒提取质粒纯化并测定质粒浓度,根据摩尔常数计算目标基因的拷贝数,并将质粒连续稀释8个数量级,以获得各目的基因的标准曲线。实时荧光定量PCR于CFX96 Real-Time PCR System (Bio-Rad公司)上完成。定量PCR扩增反应体系为20 μL,包括:10 μL的SYBR Premix EX Taq TM [宝生物工程(大连)有限公司],上、下游引物(10 pmol/μL)各0.5 μL,1.0 μL土壤总DNA模板,8 μL灭菌双蒸水。采用灭菌双蒸水代替DNA作为反应模板,设置阴性对照。反应条件为:94 ℃ 5 min;94 ℃ 45 s,55 ℃ 45 s,72 ℃ 1 min,35个循环;72 ℃ 10 min。 1.3.3 土壤微生物组的组成分析: 利用通用引物515F/907R扩增的PCR产物开展MiSeq高通量测序分析土壤微生物组成。首先采用515F/907R引物扩增土壤样品总DNA的细菌及部分古菌16S rRNA基因V4-V5高变区,修饰后的通用引物含有不同的Tag标签用以区分不同样品,包括4个活跃层和4个永冻层土壤及其对应的13C-甲烷标记处理,每个样品3个重复,共48个样品。每对引物中同时包含adaptor和key序列以提高下游测序效率。DNA水平的PCR扩增体系包含:25 μL EmeraldAmp PCR Master Mix (2×Premix) (TaKaRa),1 μL 505F引物,1 μL 907R引物,加入2.5 μL的DNA模板,补充无菌水至50 μL反应体系。PCR扩增的反应条件为:95 ℃ 3 min;95℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,30个循环;72 ℃ 10 min。获得16S rRNA基因的扩增产物后,利用Agarose Gel DNA Fragment Recovery Kit Ver. 2.0试剂盒(TaKaRa)进行切胶纯化,并将其溶于30 μL DNase-free H2O。将不同样品的PCR扩增产物等摩尔混合后,利用Illumina公司MiSeq测序仪完成序列分析。 1.4 稳定性同位素核酸探针示踪冻土区土壤好氧甲烷微生物核酸DNA如前所述,针对4个活跃层及相应的4个永冻层土壤开展了甲烷氧化培养,但仅有俄罗斯西伯利亚勒拿河活跃层土壤(KU)表现出明显的甲烷氧化潜力,甲烷浓度由初始10 mL/L降为0.25 mL/L,其他培养瓶内甲烷浓度均保持恒定,表明其它所有活跃层土壤和永冻层土壤均无法氧化10 mL/L高浓度甲烷。据此,仅选取13C-甲烷标记培养的KU活跃层土壤DNA及对照的未加甲烷的KU活跃层土壤DNA进行超高速密度梯度离心。DNA-SIP的具体操作细节见已有文献描述[13]。简言之,将2.0 µg DNA与氯化铯溶液混合形成初始密约为1.725 g/mL离心溶液,并转移至6.0 mL的超高速离心试管;利用贝克曼Vti65.2转子在20 ℃下190000×g等密度密度梯度离心44 h;采用本实验室创制的自动分层分离装置获得15个不同浮力密度DNA溶液,通过PEG-6000除去不同浮力密度DNA溶液中的氯化铯离心介质后,进一步采用70%乙醇清洗DNA沉淀,并将其溶于30 µL无菌水-20 ℃保存用于下游分析。
1.5 稳定性同位素13C-DNA鉴别及下游分析13C-DNA的鉴定策略如前所述[13]。超高速密度梯度离心对照处理和13C-甲烷标记处理的土壤总DNA,以不同浮力密度DNA为模板,采用甲烷氧化菌特异的引物(A189F/mb661R) PCR扩增pmoA标耙功能基因,利用荧光定量PCR确定pmoA功能基因在不同浮力密度DNA中的拷贝数分布规律[14],通过比较13C-甲烷和对照处理的结果鉴别13C-DNA。进一步以13C-DNA为模板,选取13C-甲烷标记处理6层到13层,对照处理8层到13层样品,利用加Tag标签的通用引物(515F/907R)对SIP分层DNA样品中的16S rRNA基因进行PCR扩增,利用1.2%的琼脂糖凝胶电泳分离5 μL的PCR产物(0.5×TAE缓冲液),分析其完整性和相对浓度,每个样品设置3个重复分析其16S rRNA序列组成,比较13C-甲烷标记处理的重层DNA与对照组重层DNA中甲烷氧化菌的相对百分比差异,确定活性甲烷碳同化微生物。
1.6 数据的统计分析16S rRNA基因的原始数据利用FLASH v1.2.2[15]进行双端拼接,共计获得高质量序列604437条,再利用QIIME[16]去除Tag标签,利用sumaclust[17]的算法以97%的相似度聚类提取OTU,利用Greengenes数据库进行注释,得到每个样品中门(Phylum)、纲(Class)、目(Order)、科(Family)、属(Genus)水平上物种组成数据后进行注释,按照高质量序列数量最小的样品设置3900条进行抽平,进行多样性分析。NMDS (Non-metricmultidimensional scaling)分析通过R语言Vegan软件包(R Development Core Team,2008) 完成。以97%的相似度对活跃层和冻土层土壤微生物序列OTU注释,发现在属水平上大量微生物无法注释,故在科的水平开展土壤微生物组的群落结构分析。活跃层与永冻层土壤微生物相对组成之间的平均值差异采用t-检验分析,P < 0.05表示显著差异。原始数据NCBI登录号为SRP096268。
2 结果和分析 2.1 北极冻土活跃层和永冻层土壤微生物组的呼吸动力学规律培养瓶内CO2累积量可作为土壤呼吸强度表征指标。如图 2-A所示,挪威斯瓦尔巴岛和俄罗斯西伯利亚4个典型冻土区的活跃层土壤呼吸强度明显高于永冻层,但土壤呼吸强度存在着极大的空间异质性。4个活跃层的土壤呼吸速率范围为61-7293 nmol CO2/(g dws·d),而4个永冻层的土壤呼吸速率范围47-523 nmol CO2/(g dws·d)。西伯利亚冻土区土壤呼吸速率明显高于挪威斯瓦尔巴岛,高达17倍之多,而西伯利亚勒拿河LF活跃层土壤呼吸速率最高,挪威斯瓦尔巴岛NA活跃层土壤呼吸速率最低,两者活跃层之间的差异高达117倍。
|
| 图 2. 冻土区土壤二氧化碳排放速率(A)和氧气消耗速率(B) Figure 2. Soil respiration. A: carbon dioxide accumulation rates during incubation periods (26 days); B: oxygen consumption rates. Boxplot including the information of four sites and the whisker is the largest value and the error bar of barplot comes from methane and non-methane treatment. The abbreviations of AD, NA, KU and LF refer to the sites where soils of active and permafrost layers were collected as shown in Figure 1. |
培养瓶内氧气浓度与土壤呼吸CO2浓度变化负相关,其O2消耗速率和CO2产生速率表现出相同的变化规律,表明土壤微生物消耗氧气利用有机碳源异养呼吸。如图 2-B所示,挪威斯瓦尔巴岛和西伯利亚4个冻土区的活跃层土壤氧气消耗速率明显高于永冻层。但冻土区土壤氧气消耗速率同样存在较大的空间变异性,4个活跃层的土壤呼吸速率范围为82-5292 nmol O2/(g dws·d),而4个永冻层变幅为70-308 nmol O2/(g dws·d)。西伯利亚冻土区土壤氧气消耗速率明显高于挪威斯瓦尔巴岛,高达14倍之多,而西伯利亚勒拿河LF活跃层土壤氧气消耗速率奇高,可达5292 nmol O2/(g dws·d),3 d内即可将培养瓶内氧气消耗殆尽。挪威斯瓦尔巴岛NA土壤氧气消耗速率最低,培养26 d过程中瓶内氧气浓度几乎没有发生变化,在氧气浓度变化最小检测限之内,与较低的土壤微生物呼吸速率一致(图 2-A)。
值得注意的是,根据实际的氧气消耗量计算(图 2-B),土壤CO2实际排放量高于理论计算值(图 2-A),表明固体冷冻状态下活跃层和永冻层土壤中闭蓄了一定CO2,在10 ℃融解过程中通过物理扩散方式进入培养瓶上方。通过实际氧气消耗量推测土壤异养呼吸释放的CO2,进而根据实际CO2排放速率推测被冰冻闭蓄的CO2量。计算表明AD、KU与LF活跃层闭蓄于土壤中的CO2量占其实际排放量贡献分别为40%、36%与27%,永冻层闭蓄于土壤中的CO2量占其实际排放量贡献分别为12%、56.8%与38%,NA的活跃层和永冻层土壤呼吸较低,其氧气的消耗量在检测限附近,无法计算冰冻闭蓄的CO2量。进一步的统计分析表明,冻土区土壤呼吸强度和氧气消耗速率与土壤性质紧密相关,与总碳(P=0.015) 和总氮(P=0.034) 正相关,与土壤是活跃层还是永冻层相关(P=0.024)。
2.2 北极冻土活跃层和永冻层土壤甲烷氧化潜力俄罗斯西伯利亚和挪威斯瓦尔巴岛4个活跃层及相应的4个永冻层土壤中,仅发现西伯利亚勒拿河KU活跃层土壤能够几乎完全氧化10 mL/L的高浓度甲烷。在整个26 d培养过程中,其他7个活跃层和永冻层土壤培养瓶内甲烷浓度始终维持在初始8-15 mL/L水平,表明这些土壤不具备氧化高浓度甲烷能力。相反,KU活跃层土壤在26 d内几乎将瓶内10 mL/L甲烷消耗殆尽,其甲烷氧化速率约为50 nmol CH4/(g dws·d),但KU活跃层下方的永冻层土壤却无明显的甲烷氧化。此外,西伯利亚LF的活跃层和永冻层土壤在培养过程中释放了大量甲烷,导致培养瓶内甲烷浓度持续上升,其甲烷排放速率分别0.3和11.7 nmol CH4/ (g dws·d)。
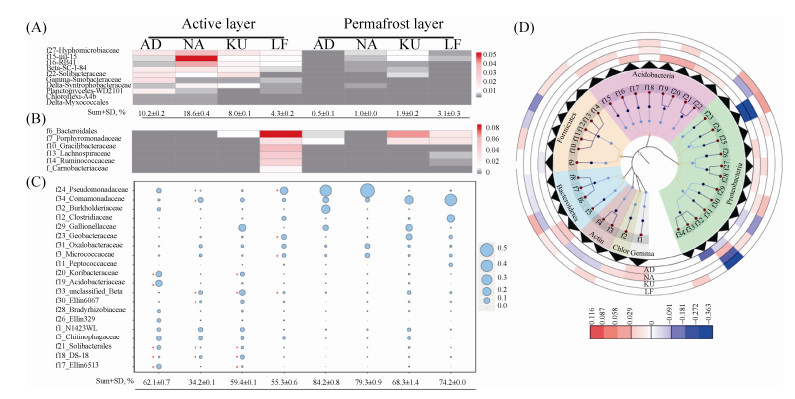
2.3 北极冻土活跃层和永冻层土壤微生物组的比较分析分子生态学分析表明活跃层土壤微生物组的数量、组成和多样性指数明显高于永冻层,并且绝大多数位点均达到了统计显著性(图 2)。首先,实时荧光定量PCR分析表明活跃层土壤微生物组的16S rRNA基因拷贝数明显高于永冻层,除了西伯利亚KU样点外,其他3个样点的差异均达到统计显著。但值得注意的是,活跃层和永冻层微生物数量均存在较大的空间异质性,活跃层土壤中微生物16S rRNA基因拷贝数为7.1×108-3.2×1010 copies/g dws,而永冻层微生物16S rRNA基因拷贝数为6.8×107-7.0×109 copies/g dws。西伯利亚勒拿河LF样点土壤微生物量最高,活跃层和永冻层分别为3.2×1010 copies/g dws与7.0×109 copies/g dws,显著性高于其他冻土层微生物数量。其次,通过谱系多样性指数PD以及稀释曲线分析表明,活跃层的微生物多样性显著高于永冻层,活跃层土壤微生物多样性指数由高到低分别为NA > AD > KU > LF,但KU与LF之间的差异不显著(图 3-B)。永冻层的多样性指数由高到低分别为KU > LF > NA > AD,其中NA与AD之间的多样性不显著。此外,土壤微生物组16S rRNA基因的非度量多维尺度NMDS分析表明(图 3-C),活跃层与永冻层之间的微生物群落结构具有显著差异。相似度分析[Multiple Response Permutation Procedure (MRPP)]结果表明,活跃层和永冻层群落结构差异性由高到低分别是AD (A=0.748,P=0.004)、KU (A=0.712,P=0.002)、NA (A=0.654,P=0.005) 和LF (A=0.507,P=0.002)。挪威斯瓦巴岛(AD,NA)与俄罗斯西伯利亚(KU,LF)冻土之间的微生物群落结构差异显著,而来自同一地区的冻土之间群落结构更为相似。总体而言,冻土区微生物数量变化与总碳显著相关(P=0.041),而冻土区地理位置是土壤微生物群落分异的主导因子。

|
| 图 3. 北极冻土活跃层与冻土层微生物丰度与多样性 Figure 3. Microbial diversity and abundance of active layer and permafrost layer. A: 16S rRNA gene copy number of active layer and permafrost layer with three replicates; B: Rarefaction of each sample; C: Non-metric multidimensional scaling of active layer and permafrost layers at genus level; D: Relative abundance of bacterial taxa at phylum of each sample with three replicates. The abbreviations of AD, NA, KU and LF refer to the sites where soils of active and permafrost layers were collected as shown in Figure 1. |
在微生物分类学门水平(图 3-D),室内模拟培养后活跃层和永冻层土壤微生物群落组成明显不同。变形菌门、酸杆菌门、厚壁菌门、放线菌门和Planctomycetes门普遍存在于4个冻土区样点。变形菌是优势微生物类群,在所有冻土层中显著高于活跃层,在永冻层中的占比由高到低分别为86.3%(AD) > 81.8%(NA) > 61.3%(KU) > 52.1%(LF),在活跃层土壤微生物组中的占比由高到低分别为53.5%(KU) > 45.3%(LF) > 37.9%(NA) > 35.7%(AD)。Acidobacteria和Planctomycetes门的规律正好相反,在活跃层中显著高于永冻层。Acidobacteria在活跃层中的平均丰度为20.0%,永冻层中的平均丰度为2.6%;Planctomycetes在活跃层中的平均丰度为1.8%,在永冻层中的平均丰度为0.2%。此外,活跃层显著高于永冻层的门除了上述2个门之外,在采样点AD还有Armatimonadetes、Elusimicrobia、OD1、TM6和Verrucomicrobia,相对百分比总和达到1.6%。在采样点NA则有Verrucomicrobia、Elusimicrobia、Armatimonadetes与Nitrospirae,相对百分比总和达到1.6%。在采样点KU还发现了Verrucomicrobia、Elusimicrobia、Armatimonadetes和Nitrospirae,相对百分比总和为1.2%。在采样点LF的门水平活跃层与永冻层的细菌差异最小,除Acidobacteria和Planctomycetes门,仅有Verrucomicrobia显著高于其永冻层,相对百分比为0.2%。
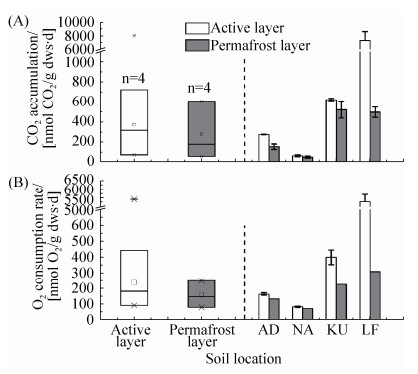
进一步在科的水平对土壤微生物组进行分类后,针对活跃层和永冻层土壤中共同存在或者特异性存在的微生物类群开展分析(图 4)。在所有微生物类群中,发现10个类群普遍存在于活跃层和永冻层中,并在活跃层中的相对丰度显著高于永冻层(Wilcoxon Signed-Rank Test,P < 0.05),可能是活跃层和永冻层分异的特征微生物,平均值分别为10.3%和1.6% (图 4-A)。这些类群在4个活跃层由高到低分别为18.6% (NA)、10.2% (AD)、8.0% (KU)、4.3% (LF);而在永冻层中分别为3.1% (LF)、1.9% (KU)、1.0% (NA)、0.5% (AD)。这10个类群具有明显的地理分异特征,在挪威斯瓦尔巴岛活跃层土壤中明显高于俄罗斯西伯利亚,而在永冻层中则表现出相反规律,导致其在挪威斯瓦德活跃层/永冻层的比例远高于俄罗斯西伯利亚,分别为23.7 (AD)、9.8 (NA)、2.6 (KU)和4.4 (LF)。同时发现俄罗斯西伯利亚LF活跃层土壤中有6个显著优势科,这些科在俄罗斯西伯利亚LF活跃层的相对百分比显著高于其他所有土壤,相对百分比总和达到26.9% (图 4-B)。
|
| 图 4. 北极冻土活跃层与永冻层在科水平上的微生物差异分布 Figure 4. Microbial community structure difference at family level of active layer and permafrost layer. A: Bacterial family of selected sampling sites that is higher in active layer than permafrost layer; B: Bacterial families that are significantly higher in Siberia Lena delta First terrace active layer than other soils; C: Bubble plot of other significantly different families between active layer and permafrost layer, red star marks out the significant higher family realtive abundance in active layer than permafrost layer; D: Taxonomic tree of bacterial 16S rRNA for bacterial families with statistically significant changes between active layer and permafrost layer. Metadata in lanes 1–4 indicate the relative increasing (red) or decreasing (blue) abundance of active layer compared to permafrost layers where difference is presentey by colors intensity. The abbreviations of AD, NA, KU and LF refer to the sites where soils of active and permafrost layers were collected as shown in Figure 1. |
除活跃层中显著高于永冻层的科及俄罗斯西伯利亚LF活跃层的显著优势科外,各采样点的活跃层和永冻层中均存在一些其他优势科(图 4-C),并且多数来自酸杆菌门及变形菌门(图 4-D)。例如,针对挪威斯瓦尔巴岛冻土区AD活跃层中,Acidobacteriaceae及Koribacteraceae科(Acidobacteria门)的相对百分比分别为12.2%及9.0%,显著高于永冻层;而在NA活跃层中,Comamonadaceae及Oxalobacteraceae (Burkholderiales目)的相对丰度分别为7.5%与3.2%,显著高于永冻层。此外,针对俄罗斯西伯利亚冻土,KU活跃层中Gallionellaceae及Koribacteraceae相对丰度分别为14.8%与5.1%,皆为优势科并显著高于永冻层,前者属于β变形菌,后者则隶属Acidobacteria门。勒拿河第一梯度LF活跃层中优势科显著不同于其他3个采样点,Pseudomonadaceae、Bacteroidales和Geobacteraceae的相对百分比分别为17.4%、8.5%和7.2%,可能是勒拿河第一梯度LF活跃层呼吸强度异常高的主要原因。通过直接比较活跃层与永冻层主要科的相对百分比差异(图 4-D),可以看出随着温度升高,微生物组成在冻土融化一个月后仍保留显著的差异,可能是永冻层碳排放速率不同的原因,而不同区域永冻层优势菌结构分异也可能与环境中的可利用碳源变化相关。
2.4 北极冻土活跃层和永冻层土壤甲烷氧化的活性微生物群落结构如前所述,4个样点共计8个活跃层和永冻层土壤中,仅俄罗斯西伯利亚KU活跃层土壤氧化了10 mL/L的13C-高浓度甲烷(100 mL培养瓶上方甲烷浓度从10 mL/L降至0.24 mL/L)并能够开展下一步的稳定性同位素核酸探针研究。根据土壤微生物总DNA的16S rRNA测序结果,发现KU的加甲烷和不加甲烷处理的甲烷碳同化微生物群落结构无显著差异(图 5-A),但各采样点中活跃层甲烷碳同化微生物占土壤微生物组的相对丰度明显高于永冻层(图 5-A)。4个活跃层中甲烷碳同化微生物相对丰度由高到低分别为:2.3% (KU),1.8% (NA),1.4% (AD),0.9% (LF);而4个永冻层分别为:1.4% (KU),1.1% (NA),1.0% (AD),0.8% (LF)。16S rRNA基因的遗传系统发育分析表明,冻土区4个样点的甲烷碳同化微生物主要类群基本一致,包括Methylobacterium、Methylobacteriaceae科、Methylophilaceae、Methylophilales和Crenothrix (图 5-B)。这些甲烷碳同化微生物的相对百分比具有明显的空间异质性。例如,西伯利亚KU活跃层中优势甲烷碳同化微生物为Methylophilaceae (1.3%)、Methylobacterium (0.4%)和Crenothrix属(0.2%),与KU永冻层基本一致。挪威斯瓦尔巴群岛AD活跃层的甲烷碳同化微生物相对百分比为1.35%,以alpha-Proteobacteria (Methylocystaceae科,Methylobacterium属)和beta-Proteobacteria (Methylophilaceae)为主,AD永冻层的甲烷碳同化微生物相对百分比为0.97%,以alpha-Proteobacteria (Methylobacterium属)和beta-Proteobacteria (Methylophilales目)为主。挪威斯瓦尔巴岛总体甲烷碳同化微生物数量低于西伯利亚勒拿河区域。通过比较加13C-甲烷处理与对照组处理的重层DNA中甲烷氧化菌标靶pmoA基因的相对丰度变化,发现13C-标记处理的重层DNA并未发生pmoA基因的明显富集(图 5-C)。然而,进一步利用高通量测序重层DNA中的微生物16S rRNA基因,则发现甲烷碳同化微生物类群得到了明显富集,主要包括Methylobacterium 属、Methylosinus属、beta-Proteobacteria的Methylophilales目的2个属,以及Crenothrix。在对照组重层DNA中这些微生物16S rRNA基因序列占所有16S rRNA基因的相对百分比约为0,但在13C处理的重层DNA中,则分别增至0.25%、0.10%、0.01%、0.15%、0.50%、0.80% (图 5-D),表明这些微生物在甲烷氧化过程中同化了其代谢产物并被13C标记。
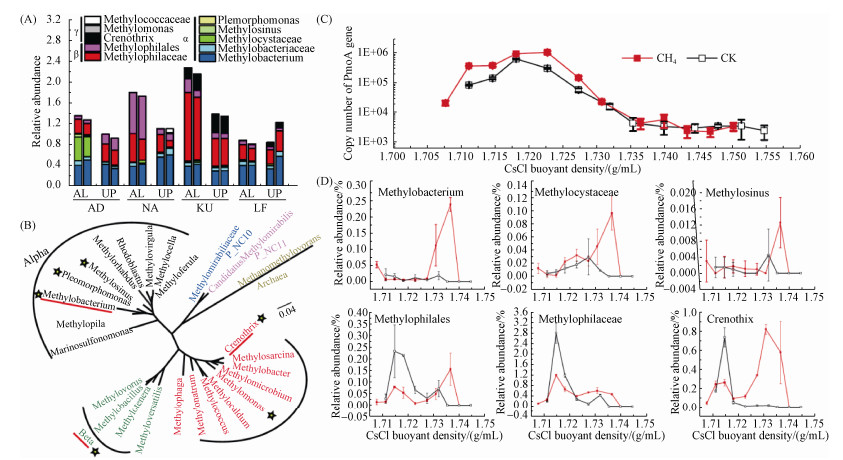
|
| 图 5. 北极冻土区活跃层与永冻层的甲烷碳同化微生物菌与活性甲烷碳同化微生物菌 Figure 5. Methylotrophs and active methylotophs in active layer and permafrost alyer. A: Relative abundance of the 16S rRNA gene sequence reads affiliated with methylotrophs in each sample; B: Phylogenetic tree based on 16S rDNA gene of methylotrophs where "star" represents the dominant methylotrophs among samples and red lines refer to the apprant active methylotrophs; C: Quantitative distributions of the pmoA gene copy numbers across the entire buoyant density gradient of the fractionated DNA from the soil incubated with 13-C labeled methane or without methane; D: Relative frequency of the 16S rRNA gene sequence reads of different active methylotrophs in each fraction in heavier layer after incubation for 26 days. The abbreviations of AD, NA, KU and LF refer to the sites where soils of active and permafrost layers were collected as shown in Figure 1. |
3 讨论
本研究发现北极冻土区季节性融解的活跃层土壤呼吸强度明显高于活跃层,并且土壤微生物组群落结构具有明显的空间异质性。随着全球变暖加剧,活跃层深度向永冻层延伸,使得永冻层中的碳源解冻,更容易被微生物利用。但由于永冻层长期处于固体冰冻状态,其微生物丰度、活性及多样性较低,导致短时间内永冻层融化后,其碳排放速率仍显著低于活跃层。特别是10 ℃培养1个月后,冻土微生物组可能发生一定的演替,不能完全代表原位环境的微生物群落结构,未来仍需强化室内模拟与野外观测的综合研究。我们推测在较大的时间尺度下,永冻层转变为活跃层后也会经历长期的季节性冻融,可能向活跃层发生定向演替并在原位形成稳定的冻土功能微生物组,其中的微生物群落能够迅速在土壤融化后分解碳源,导致碳排放速率升高并形成全球变暖正反馈。
本研究样品来自典型冻土区,包括挪威斯瓦尔巴群岛及俄罗斯西伯利亚勒拿河区域。已有研究表明这些样区域存在永冻层融化、活跃层厚度增加的现象,但相关的冻土区微生物机理研究尚未有报道[18]。我们的结果表明活跃层呼吸强度显著高于永冻层,微生物数量和组成的差异是土壤呼吸变化的主要原因。微生物群落结构分异是不同生物对环境长期适应的进化结果。比较分析各样点活跃层与永冻层细菌群落结构发现,活跃层特征微生物主要来自于Acidobacteria门与Planctomycetes门。此外,也有部分属于变形菌门,如Hyphomicrobiaceae科,仅能在可利用碳源少的贫营养情况下生存,且大部分为好氧化能菌,少部分能通过反硝化或混合酸发酵条件下厌氧生长[19]。Acidobacteria门中ⅲ1-15目,Solibacteraceae科被发现是对水分响应较明显的类别,干旱条件下相对百分比减少,水分较多的条件下增多[20],这种干湿交替模式与活跃层的季节性冻融过程极为类似,导致这些细菌可能具有竞争优势并在碳转化中发挥作用。
挪威斯瓦尔巴群岛AD、NA冻土,俄罗斯西伯利亚勒拿河三角洲KU、LF冻土之间的微生物群落结构具有明显分异,表明活跃层和永冻层微生物群落结构受空间分布约束(图 6)。全球冻土区分布广泛且具有较大的空间异质性,主要体现在历史地理气候因子、关键元素含量,以及活跃层与永冻层分界线形成过程中所带来的理化因子差异[21]。因此,长期地质历史过程中形成的地理环境分异极可能是微生物群落定向演替的重要原因。据此,我们利用原位的土壤理化性质与微生物相对丰度进行主成分分析(图 6)。结果表明总碳、总氮、碳氮比、总硫、土壤密度及样品采样深度这些理化因子中,采样深度能够较好地解释细菌群落的分异规律(R2=0.87,P=0.01)。同时,活跃层与冻土层作为影响因子也具有统计显著性(R2=0.76,P=0.03),各采样点活跃层之间的细菌群落结构差异比活跃层与永冻层之间的细菌群落结构差异更小。然而,由于本研究的样本容量较小,未来仍需开展大数据的整合分析,准确分析在不同空间尺度下驱动环境微生物群落演替的具体环境因子,特别是在活跃层中相对丰度显著高于永冻层的10大类微生物演替过程的主控因子(图 4-A)。

|
| 图 6. 北极冻土区活跃层和永冻层微生物科水平变化规律的环境因子冗余分析(RDA) Figure 6. Redundancy analysis biplot for bacterial commununity on family level. Bold arrow represents soil parameter with significant (Wilcoxon Signed-Rank Test, P < 0.05) infulence on bacterial communities' structure. The abbreviations of AD, NA, KU and LF refer to the sites where soils of active and permafrost layers were collected as shown in Figure 1. |
值得注意的是,已有研究表明pH是北极高寒地区土壤群落结构的主要影响因子[22],但本研究中并未观测到类似的现象,表明未来冻土区微生物地理学研究中应该更加重视冻土垂直空间尺度的研究。在AD、NA、KU三个采样点中,活跃层pH略低于永冻层,而LF样点规律恰好相反。相应的,我们在AD、NA、KU三个样点中也发现Acidobacteria门微生物类群显著高永冻层,而在LF采样点这种规律不明显。事实上,瑞士恩嘎丁西北和东南高海拔冻土区研究也得到了类似结果,活跃层pH低于永冻层,且活跃层中Acidobacteria丰度高于永冻层[5]。然而,尽管Acidobacteria丰度与pH似乎具有一定相关性,但绝大部分Acidobacteria尚未被培养,其生理生态功能未知,这些微生物是否为耐酸或嗜酸菌仍待进一步研究。我们推测不同冻土区可能适应特异的地理环境而演化出特征优势菌(图 4-B、C),这些优势菌的生理代谢策略使之能够更好适应当地的地理环境。例如,俄罗斯西伯利亚勒拿河三角洲的LF活跃层具有异常高的CO2,最高可达其他位点的120倍(图 2-A),尽管我们发现了LF活跃层中6个微生物科显著高于其他所有冻土,相对百分比总和达到26.9% (图 4-B),但仍需通过纯培养或同位素示踪技术研究这些微生物在土壤呼吸过程中的作用,特别是综合利用微生物学、地质学、分子生物学和计算生物学等学科的先进理念和手段,深入探讨冻土区难培养微生物生理代谢特点并推测其功能,解析宏观尺度下微生物驱动的关键元素生物地球化学循环过程及其环境影响。
本研究2个冻土区的供试土壤几乎都不具备高浓度甲烷氧化能力。所有8个活跃层和冷冻层土壤样品中,仅在俄罗斯西伯利亚勒拿河KU活跃层土壤发现了明显的高浓度甲烷氧化。尽管KU永冻层的甲烷碳同化微生物群落结构与其活跃层相似,未发表的pmoA基因实时荧光定量PCR结果表明永冻层中甲烷氧化菌数量低于活跃层,可能是永冻层甲烷氧化潜力低的重要原因。稳定性同位素示踪甲烷碳同化微生物核酸DNA结果表明:KU活跃层主要活性甲烷碳同化微生物为Crenothrix科,与Christiansen (2015) 在格陵兰岛迪斯科岛湿地中发现的优势甲烷氧化菌一致。这一类微生物通常能够适应高浓度甲烷环境,被认为具有r-生长策略。相反,类型Ⅱ甲烷氧化菌具有K-生长策略,具有极高的底物亲和力并通过氧化2.5 mg/m3极低浓度大气甲烷生长,通常存在于北极旱地活跃层土壤中[22]。事实上,KU活跃层氧化高浓度甲烷后,其中的甲烷单加氧酶功能基因pmoA拷贝数增加约1倍,从2.4×106个/g dws增至4.8×106个/g dws。这表明KU样点的甲烷氧化菌具有较强的生理生长活性。挪威斯瓦尔巴岛活跃层和永冻层均不能氧化高浓度甲烷,其主要原因可能与底物甲烷浓度相关,这些冻土层通常含水量较低,属于典型的北极旱地苔原土壤,一般认为其中的甲烷氧化菌通常具有极高的亲和力,利用极低的大气浓度甲烷生长,在长期的环境驯化下,这些甲烷氧化菌可能丧失了高浓度甲烷氧化能力,或者需要更长的适应期以氧化高浓度甲烷。总体而言,KU活跃层甲烷氧化菌数量高于永冻层、前者能够氧化高浓度甲烷而后者不具备这一能力。这些结果在一定程度上表明全球升温条件下,永冻层解冻变为活跃层之后,特定功能微生物类群极可能与环境发生协同进化并发挥重要作用。
4 结论北极冻土区土壤呼吸具有明显的空间分异特征,西伯利亚冻土区高于挪威斯瓦尔巴岛地区,并表现出垂直分布规律,即所有冻土区中活跃层土壤呼吸强度明显高于其下的永冻层。土壤微生物组也表现出类似规律,北极冻土区活跃层的微生物多样性显著高于永冻层。在所有活跃层中,均发现变形菌和酸杆菌门共计10个微生物科的丰度显著高于永冻层,其中Hyphomicrobiaceae、Solibacteraceae和Sinobacteraceae是优势科,在活跃层中的相对丰度约为4.3%-18.6%,是永冻层相应微生物类群的2.6-23.7倍。这些微生物可能是冻土活跃层的特征类群,在土壤呼吸过程中发挥了重要作用。此外,俄罗斯西伯利亚勒拿河三角洲的冻土活跃层中6个微生物科显著高于所有其它活跃层和永冻层,相对丰度为26.9% (主要包括Bacteroidales和Gracilibacteraceae优势科),可能是该冻土活跃层土壤呼吸速率异常高的原因。进一步发现俄罗斯西伯利亚冻土活跃层土壤中活性的甲烷碳同化微生物主要为Methylobacterium和Crenothrix。这些结果暗示未来全球变暖情景下,永冻层逐渐融解并形成活跃层后,其中的微生物类群将可能经历定向演替,并在北极冻土碳转化中发挥重要作用。研究结果为准确预测未来全球变暖情景下冻土区碳转化过程提供了参考。
致谢:感谢丹麦哥本哈根大学冻土中心主任Bo Elberling教授实验室Morten Schostag Nielsen和Christian Nyrop Alber在实验过程中提供的技术帮助。
| [1] | Margesin R. Permafrost soils. Berlin Heidelberg:Springer, 2008. |
| [2] | Åkerman HJ, Johansson M. Thawing permafrost and thicker active layers in sub-arctic Sweden. Permafrost and Periglacial Processes, 2008, 19(3): 279–292 DOI:10.1002/ppp.v19:3. |
| [3] | Liu GX, Ma XJ, Chen T, An LZ, Wang XL. Progress and significance of studies on microorganisms in permafrost sediments. Journal of Glaciology and Geocryology, 2004, 26(2): 188–191 |
| [4] | Schuur EAG, Abbott BW, Bowden WB, Brovkin V, Camill P, Canadell JG, Chanton JP, Chapin Ⅲ FS, Christensen TR, Ciais P, Crosby BT, Czimczik CI, Grosse G, Harden J, Hayes DJ, Hugelius G, Jastrow JD, Jones JB, Kleinen T, Koven CD, Krinner G, Kuhry P, Lawrence DM, McGuire AD, Natali SM, O'Donnell JA, Ping CL, Riley WJ, Rinke A, Romanovsky VE, Sannel ABK, Schädel C, Schaefer K, Sky J, Subin ZM, Tarnocai C, Turetsky MR, Waldrop MP, Anthony KMW, Wickland KP, Wilson CJ, Zimov SA. Expert assessment of vulnerability of permafrost carbon to climate change. Climatic Change, 2013, 119(2): 359–374 DOI:10.1007/s10584-013-0730-7. |
| [5] | Schuur EAG, Bockheim J, Canadell JG, Euskirchen E, Field CB, Goryachkin SV, Hagemann S, Kuhry P, Lafleur PM, Lee H, Mazhitova G, Nelson FE, Rinke A, Romanovsky VE, Shiklomanov N, Tarnocai C, Venevsky S, Vogel JG, Zimov SA. Vulnerability of permafrost carbon to climate change:Implications for the global carbon cycle. Bioscience, 2008, 58(8): 701–714 DOI:10.1641/B580807. |
| [6] | Yergeau E, Hogues H, Whyte LG, Greer CW. The functional potential of high Arctic permafrost revealed by metagenomic sequencing, qPCR and microarray analyses. The ISME Journal, 2010, 4(9): 1206–1214 DOI:10.1038/ismej.2010.41. |
| [7] | Schostag M, Stibal M, Jacobsen CS, Baelum J, Taş N, Elberling B, Jansson JK, Semenchuk P, Prieme A. Distinct summer and winter bacterial communities in the active layer of Svalbard permafrost revealed by DNA-and RNA-based analyses. Frontiers in Microbiology, 2015, 6: 399 |
| [8] | Mackelprang R, Waldrop MP, DeAngelis KM, David MM, Chavarria KL, Blazewicz SJ, Rubin EM, Jansson JK. Metagenomic analysis of a permafrost microbial community reveals a rapid response to thaw. Nature, 2011, 480(7377): 368–371 DOI:10.1038/nature10576. |
| [9] | Hultman J, Waldrop MP, Mackelprang R, David MM, McFarland J, Blazewicz SJ, Harden J, Turetsky MR, McGuire AD, Shah MB, VerBerkmoes NC, Lee LH, Mavrommatis K, Jansson JK. Multi-omics of permafrost, active layer and thermokarst bog soil microbiomes. Nature, 2015, 521(7551): 208–212 DOI:10.1038/nature14238. |
| [10] | Faucherre S, Blok D, Jørgensen CJ, Siewert MB, Hugelius G, Kuhry P, Elberling B. Short and long term controls on permafrost carbon turnover across the Arctic (in prep.). Biogeosciences, 2017 |
| [11] | Lane DJ. 16S/23S rRNA sequencing//Stackebrandt E, Goodfellow M. Nucleic Acid Techniques in Bacterial Systematics. New York, NY:John Wiley and Sons, 1991:125-175. |
| [12] | Stubner S. Enumeration of 16S rDNA of Desulfotomaculum lineage 1 in rice field soil by real-time PCR with SybrGreenTM detection. Journal of Microbiological Methods, 2002, 50(2): 155–164 DOI:10.1016/S0167-7012(02)00024-6. |
| [13] |
Jia ZJ. Principle and application of DNA-based stable isotope probing-a review.
Acta Microbiologica Sinica, 2011, 51(12): 1585–1594 (in Chinese) 贾仲君. 稳定性同位素核酸探针技术DNA-SIP原理与应用. 微生物学报, 2011, 51(12): 1585–1594. |
| [14] |
Zheng Y, Jia ZJ. Next generation sequencing and stable isotope probing of active microorganisms responsible for aerobic methane oxidation in red paddy soils.
Acta Microbiologica Sinica, 2013, 53(2): 173–184 (in Chinese) 郑燕, 贾仲君. 新一代高通量测序与稳定性同位素示踪DNA/RNA技术研究稻田红壤甲烷氧化的微生物过程. 微生物学报, 2013, 53(2): 173–184. |
| [15] | Magoč T, Salzberg SL. FLASH:fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21): 2957–2963 DOI:10.1093/bioinformatics/btr507. |
| [16] | Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Peña AG, Goodrich J, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335–336 DOI:10.1038/nmeth.f.303. |
| [17] | Mercier C, Boyer F, Bonin A, Coissac E. SUMATRA and SUMACLUST:fast and exact comparison and clustering of sequences//Programs and Abstracts of the SeqBio 2013 workshop (Abstract). GdRBIM and gdrIM, Montpellier, France. 2013, 27_29. Available at http://metabarcoding.org/sumatra. |
| [18] | Instanes A. Incorporating climate warming scenarios in coastal permafrost engineering design-Case studies from Svalbard and northwest Russia. Cold Regions Science and Technology, 2016, 131: 76–87 DOI:10.1016/j.coldregions.2016.09.004. |
| [19] | Oren A, Xu XW. The family Hyphomicrobiaceae//Rosenberg E, DeLong EF, Lory S, Stackebrandt E, Thompson F. The Prokaryotes. Berlin Heidelberg:Springer, 2014:247-281. |
| [20] | Barnard RL, Osborne CA, Firestone MK. Responses of soil bacterial and fungal communities to extreme desiccation and rewetting. The ISME Journal, 2013, 7(11): 2229–2241 DOI:10.1038/ismej.2013.104. |
| [21] | Zhang TJ, Frauenfeld OW, Serreze MC, Etringer A, Oelke C, McCreight J, Barry RG, Gilichinsky D, Yang DQ, Ye HC, Ling F, Chudinova S. Spatial and temporal variability in active layer thickness over the Russian Arctic drainage basin. Journal of Geophysical Research:Atmospheres, 2005, 110(D16): D16101 DOI:10.1029/2004JD005642. |
| [22] | Chu HY, Fierer N, Lauber CL, Caporaso JG, Knight R, Grogan P. Soil bacterial diversity in the Arctic is not fundamentally different from that found in other biomes. Environmental Microbiology, 2010, 12(11): 2998–3006 DOI:10.1111/emi.2010.12.issue-11. |
 2017, Vol. 57
2017, Vol. 57