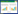中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 杨秀清, 郭兆琳. 2017
- Xiuqing Yang, Zhaolin Guo. 2017
- 红球菌R04苯甲酸转运相关膜蛋白RHOGL009301的生理功能
- Physiological function of membrane protein RHOGL009301 involved in transport of benzoate in Rhodococcus sp. R04
- 微生物学报, 2017, 57(4): 609-620
-
文章历史
- 收稿日期:2016-11-17
- 修回日期:2016-12-28
- 网络出版日期:2017-01-19
红球菌R04是一株分离于华北油田污染土壤中的微生物,可以高效降解苯甲酸钠、联苯和多氯联苯等多种芳香族化合物[1]。红球菌R04通过2, 3-双加氧酶途径降解联苯/多氯联苯[2],具体如下:联苯/多氯联苯经羟基化双加氧酶的催化生成2, 3-二氢二羟基联苯,再被脱氢酶催化成2, 3-二羟基联苯 (DHBP),随后间位断裂形成黄色开环化合物2-羟基-6-氧基-6-苯基-2, 4-己二烯酸 (HOPDA),最后被水解为苯甲酸,从而进入细菌的苯甲酸代谢途径[3]。在此过程中产生的中间代谢产物,如HOPDA、苯甲酸等,会先被红球菌R04排出体外,随后再进入体内[4]。苯甲酸是多种芳香化合物生物降解的关键中间产物[5],在食品、化工及染料工业中的应用广泛,开展苯甲酸的微生物降解[6]及转运机理的研究,对人类的生存和健康有重要的指导作用。
苯甲酸又称安息香酸,是简单的芳香族羧酸。对于芳香酸转运蛋白[7]转运机理的研究报道较少,但在一些微生物体内已经发现苯甲酸的转运蛋白。谷氨酸棒状杆菌中发现的苯甲酸的跨膜转运蛋白有2个家族:MFS转运蛋白超家族和BenE家族,BenE只在BenK缺失情况下才行使跨膜转运苯甲酸的功能[8];Acinetobacter calcoaceticus PHEA-2有3种苯甲酸转运蛋白[9](BenE、BenK、BenP)。有文献报道,Pseudomonas putida PRS2000的趋化性与苯甲酸的运输和降解有关[10]。本课题组红球菌R04基因组测序结果证实红球菌R04基因组中有苯甲酸转运蛋白BenE和BenQ的编码基因[11]。
转运蛋白是膜蛋白的一大类,参与运输营养物质,释放代谢产物等广泛的细胞活动[12]。膜蛋白在生物体内大量存在,是生物生存和代谢的物质承担者,具有多种多样的功能,如物质运输、能量转换、信号转导等,其中,最重要的作用是运输机体所需的营养物质,并向外界排出代谢废物[13]。红球菌R04转录组测序发现,在联苯培养条件下6个注释为膜蛋白功能的基因上调表达,其中上调表达量最高的基因是RHOGL009301,但此基因的生理功能是否与物质运输相关,目前尚不清楚。
本研究为了证实RHOGL009301为膜蛋白基因,将其与绿色荧光蛋白基因进行融合表达定位。同时,通过基因敲除,获取该基因的缺陷型菌株,并对缺陷型菌株和野生型菌株的生长和代谢进行分析,以期确定RHOGL009301基因的生理功能。这对进一步研究联苯/多氯联苯代谢中,膜蛋白的生理功能有着重要的意义,并为研究苯甲酸等芳香化合物的代谢机理和跨膜运输机制奠定基础。
1 材料和方法 1.1 材料 1.1.1 菌株和质粒: Rhodococcus erythropolis,Rhodococcus sp. R04,E. coli DH5α,pETm3c-gfp质粒,pTipQC2质粒和pK18mobsacB质粒由本实验室保存。: 1.1.2 培养基: (1) LB培养基 (g/L):胰蛋白胨10,酵母粉5,NaCl 5,蒸馏水1 L;(2) LBP培养基 (g/L):蛋白胨10,酵母粉5,NaCl 10,蒸馏水1 L;(3) LBG培养基 (g/L):蛋白胨10,酵母粉5,NaCl 5,葡萄糖10,蒸馏水1 L;(4) 红球菌基础培养基 (g/L):K2HPO4·3H2O 5.85,(NH4)2SO4 5.00,KH2PO42.93,MgSO4·7H2O 0.30,NaCl 0.20,CaCl20.03,NiSO4·7H2O 6×10–4,微量元素盐溶液200 μL,蒸馏水1 L。: 1.1.3 主要试剂和仪器: FastPfu DNA Polymerase,pEASY-T3 Cloning Kit购自北京全式金生物技术有限公司;酵母粉,胰蛋白胨购自Oxoid公司;质粒提取试剂盒及胶回收试剂盒购自北京中科瑞泰生物科技有限公司;限制性内切酶EcoR Ⅰ,Hind Ⅲ,BamH Ⅰ购自大连宝生物工程有限公司;T4 DNA Ligase购自Fermentas公司;氨苄青霉素 (Amp),卡那霉素 (Kan),氯霉素 (Chl) 购自美国Sigma-Aldrich公司;联苯购自军事医学科学院试剂站;色谱纯甲醇购自天津科密欧化学试剂有限公司;DYY-6C型电泳仪购自北京市六一仪器厂;EC250-90型电泳仪购自美国Thermo Fisher公司;日立UV-201分光光度计购自美国Hitachi Instruments公司;Delta Vision Deconvolution microscope购自美国Delta Vision公司;高效液相色谱仪购自美国Waters公司,色谱柱为Thermo Hypersil GOLD C18液相色谱柱 (4.6 mm×250 mm)。: 1.2 RHOGL009301基因表达 1.2.1 RHOGL009301基因扩增: 根据RHOGL009301基因设计上下游引物,以表 1中F1和R1为上下游引物,红球菌R04基因组为模板,PCR反应条件:95 ℃ 10 min;95 ℃ 1 min,55 ℃ 30 s,72 ℃ 90 s,30个循环;72 ℃ 5 min,得到PCR产物MP。:| Primer | Oligonucleotide sequence (5′→3′) | Restriction enzyme |
| F1 | TAGAATTCTAAAAAGGTGGAGGCATGACGC | EcoR Ⅰ |
| R1 | TAGGATCCGAGCAGGTGGACCTTCTTGTAG | BamH Ⅰ |
| F2 | TAGGATCCATGAGTAAAGGAGAAGAACT | BamH Ⅰ |
| R2 | TAAAGCTTTTTGTATAGTTCATCCATGCC | Hind Ⅲ |
| F3 | TAGAATTCATGACGGACGATCCGGGCG | EcoR Ⅰ |
| R3 | TAAAGCTTTCAGGGAGTCTCGGGGTCG | Hind Ⅲ |
1.2.2 绿色荧光蛋白基因的获得: 以表 1中F2和R2为上下游引物,pETm3c-gfp为模板,PCR反应条件:95 ℃ 10 min;95 ℃ 1 min,55 ℃ 30 s,72 ℃,得到PCR产物GFP。: 1.2.3 表达载体构建: 将pTipQC2质粒与MP分别双酶切,连接,筛选得到阳性重组质粒pTipQC2-MP;表达载体pTipQC2-MP与GFP分别双酶切,连接,筛选得到阳性重组质粒pTipQC2-MP-GFP。: 1.2.4 蛋白诱导表达: 将pTipQC2-MP-GFP电转化Rhodococcus erythropolis感受态,电转化条件为:电压2.5 kv、电容25 μF、电阻400 Ω,电击时间10 ms,电转化后立即加入800 μL LBP培养基,30 ℃培养8 h,涂在含有氯霉素 (终浓度为25 μg/mL) 的LB平板上,30 ℃培养72 h。将平板所得到的转化子接种于3 mL LB培养基中,30 ℃培养48 h,再转接1 mL培养液于50 mL三角瓶中,当OD600≈1.0时,加入诱导剂硫链丝菌素 (终浓度为0.25 mmol/L),30 ℃诱导表达18 h。: 1.3 显微镜观察
将上述30 ℃诱导培养18 h后的菌液稀释至OD600=0.036,取10 μL滴于载玻片中央,盖玻片直接压片。在Delta Vision去卷积显微镜下于100倍油镜观察,激发波长为488 nm。
1.4 RHOGL009301基因敲除 1.4.1 RHOGL009301基因克隆: 根据基因序列设计上下游引物,以表 1中F3和R3为上下游引物,红球菌R04基因组为模板,PCR反应条件:95 ℃ 10 min;95 ℃ 45 s,55 ℃ 30 s,72 ℃ 120 s,30个循环;72 ℃ 5 min。: 1.4.2 敲除载体构建: 将载体pK18mobsacB和RHOGL009301基因双酶切,连接,筛选得到阳性重组质粒;再用Nru Ⅰ酶对重组质粒进行酶切,将酶切后的质粒进行自连接,筛选得到阳性重组质粒pK18mobsacB-ΔMP。: 1.4.3 电转与筛选: 将pK18mobsacB-ΔMP电转化红球菌R04感受态,电转化条件为:电压2.5 kv、电容25 μF、电阻800 Ω,电击时间8 ms,电转化后立即加入800 μL LBP培养基,30 ℃培养4 h,涂在含有卡那霉素 (终浓度为50 μg/mL) 的LB平板上,30 ℃培养72 h。将平板所得到的转化子,接种于3 mL LBG培养基中,30 ℃培养24 h,取20 μL培养液涂在含有10%蔗糖的LBP平板上,30 ℃培养48 h,挑取单菌落进行菌落PCR筛选。: 1.5 红球菌R04野生型菌株与缺陷型菌株的生物量测定 1.5.1 野生型菌株与缺陷型菌株在不同碳源培养下的生物量: 采用静息细胞法[14]处理红球菌R04野生型菌株与缺陷型菌株,将两种菌株分别接种于含有葡萄糖、联苯、2-羟基-6-氧基-6-苯基-2、4-己二烯酸 (HOPDA) 和苯甲酸 (HOPDA和苯甲酸为联苯代谢中间产物) 的4种基础培养基中,4种碳源终浓度均为10 mmol/L。每隔8 h取样,测定野生型菌株和缺陷型菌株在不同时期的OD600。: 1.5.2 野生型菌株与缺陷型菌株在不同浓度的联苯和苯甲酸培养下的生物量: 采用静息细胞法[14]处理红球菌R04野生型菌株与缺陷型菌株,将2种菌株分别接种于不同联苯和苯甲酸浓度的基础培养基中 (终浓度分别为5、10、20 mmol/L)。联苯培养时每隔5 h取样,测定野生型菌株和缺陷型菌株在不同时期的OD600和OD435;苯甲酸培养时每隔8 h取样,测定野生型菌株和缺陷型菌株在不同时期的OD600。: 1.6 红球菌R04野生型菌株与缺陷型菌株细胞内外代谢物的测定 1.6.1 代谢物的取样: 从三角瓶中周期性地取一定的培养液,然后将其分成2份,作不同处理。一半培养液用等体积的色谱纯甲醇抽提,取上清作为待测样品,得到总的代谢物;另一半培养液离心收集菌体,用缓冲液洗涤细胞2次,用等体积色谱纯甲醇抽提细胞,取上清作为待测样品,得到胞内代谢物 (联苯培养时培养液需经玻璃棉过滤除去联苯固体颗粒后再收集菌体);胞外代谢物=总的代谢物-胞内代谢物。: 1.6.2 测定方法——HPLC分析[15]: 色谱柱为Thermo Hypersil GOLD C18液相色谱柱 (4.6 mm×250 mm);流动相为甲醇:水 (体积比9:1);柱温为室温;检测波长为254 nm;流速为1.0 mL/min;进样量10 μL。: 1.7 数据统计分析所有红球菌R04野生型菌株与缺陷型菌株的生长和代谢实验均重复3次,计算结果取平均值,实验结果以平均值±标准差表示。
2 结果和分析 2.1 RHOGL009301蛋白定位 2.1.1 表达载体构建: 将筛选得到的阳性重组质粒pTipQC2-MP-GFP分别用BamH Ⅰ和Hind Ⅲ,EcoR Ⅰ和BamH Ⅰ进行双酶切鉴定,酶切产物与目的基因大小相当 (结果未显示)。由此可知,阳性重组质粒中含有目的基因,经测序验证pTipQC2-MP-GFP表达载体构建成功。: 2.1.2 蛋白表达: 离心收集诱导表达的菌体,超声波破碎后经SDS-PAGE检测,表明RHOGL009301基因成功表达,蛋白大小为35 kDa (结果未显示),与理论相符。: 2.1.3 RHOGL009301蛋白定位: 由图 1可知,融合蛋白在Rhodococcus erythropolis中成功表达,细胞膜的四周产生了荧光,证实了该基因RHOGL009301确实为膜蛋白基因。:

|
| 图 1. RHOGL009301蛋白定位 Figure 1. Location of RHOGL009301 in Rhodococcus erythropolis. |
2.2 RHOGL009301缺陷型菌株的筛选
筛选得到的阳性重组质粒pK18mobsacB-ΔMP经酶切鉴定表明含有目的基因,测序验证敲除载体构建成功。从电转后得到的LBP平板上挑取单菌落进行菌落PCR筛选,结果如图 2所示。由图 2可知,9泳道的菌株RHOGL009301基因敲除成功。以9泳道菌株的基因组为模板,表 1中F3和R3为上下游引物,进行PCR扩增,将PCR扩增所得条带,与pEASY-T3 Cloning Kit连接后测序,测序结果验证敲除成功,同时将9泳道缺陷型菌株命名为R04ΔMP。
|
| 图 2. 缺陷型菌株菌落PCR的琼脂糖凝胶电泳 Figure 2. Agarose gel electrophoresis of deficient strains Colony PCR. Lane 1: positive control; lane 2–9: suspected deficient strains; M: marker. |
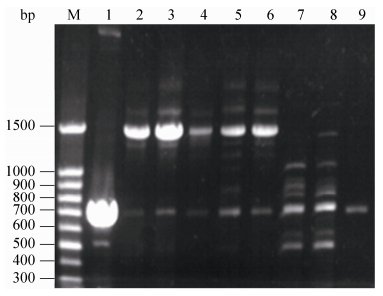
2.3 红球菌R04野生型菌株与缺陷型菌株R04ΔMP的生长 2.3.1 野生型菌株与缺陷型菌株利用不同碳源的生长: 本实验选取野生型菌株与缺陷型菌株分别在4种碳源生长16 h和64 h测定的生物量。如图 3所示,野生型菌株与缺陷型菌株在以葡萄糖和HOPDA为碳源时,生长状况几乎相同,生物量一致,而在联苯和苯甲酸培养条件下,缺陷型菌株的生物量和野生型相比,明显降低。由此可知,RHOGL009301基因的缺失影响了红球菌R04在联苯和苯甲酸中的生长[16-17],但缺陷型菌株仍然可以利用联苯和苯甲酸。:
|
| 图 3. 野生型菌株和缺陷型菌株在葡萄糖/联苯/HOPDA/苯甲酸培养条件下的生物量 Figure 3. Biomass of the wild strain and the deficient strain in different carbon sources. A: 16 h; B: 64 h. |
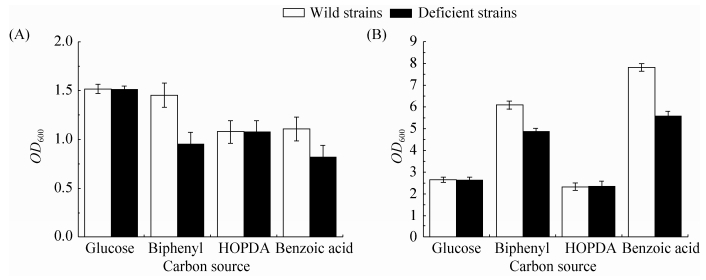
2.3.2 野生型菌株与缺陷型菌株在不同浓度联苯中的生长: 将红球菌R04野生型和缺陷型菌株R04ΔMP分别接种于不同浓度联苯的基础培养基中,测定OD600与OD435,结果如图 4所示。HOPDA为黄色物质[18],在435 nm处有最大光吸收,本实验以OD435表征HOPDA的生成量。在3种不同浓度联苯培养条件下,与野生型菌株相比,缺陷型菌株的生长速度较慢,稳定期延后,且最终生物量减小;在实验中都观察到了黄色物质HOPDA的生成,缺陷型菌株生成最大量的HOPDA的时间延后,培养基呈现黄色的时间延长。在20 mmol/L联苯培养条件下,野生型菌株的最大HOPDA生成量与缺陷型菌株相差明显,最终的生物量相差较大。:
|
| 图 4. 野生型菌株和缺陷型菌株在不同浓度联苯的生物量和HOPDA生成量 Figure 4. Biomass of the wild strain and the deficient strain in different concentrations of biphenyl. A: 5 mmol/L; B: 10 mmol/L; C: 20 mmol/L. |
2.3.3 野生型菌株与缺陷型菌株在不同浓度苯甲酸中的生长: 将红球菌R04野生型和缺陷型菌株R04ΔMP分别接种于不同浓度苯甲酸的基础培养基中,测定OD600,结果如图 5所示。随着苯甲酸浓度的增加,野生型菌株和缺陷型菌株的最终生物量都在增加;但在3种不同浓度苯甲酸培养条件下,缺陷型菌株的最终生物量明显低于野生型菌株,且随着苯甲酸浓度的增加,最终生物量的相差也增大。:
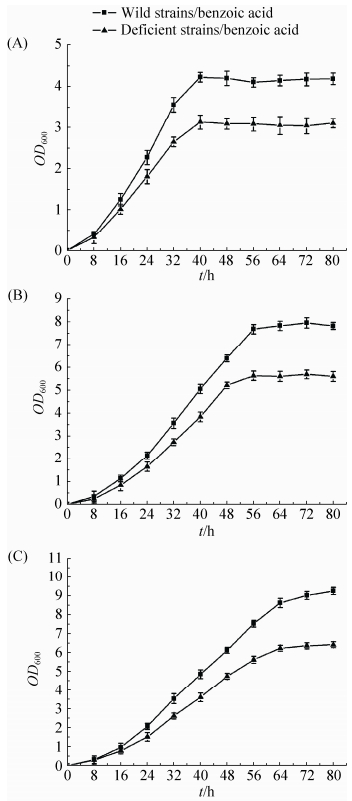
|
| 图 5. 野生型菌株和缺陷型菌株在不同浓度苯甲酸的生物量 Figure 5. Biomass of the wild strain and the deficient strain in different concentrations of benzoic acid. A: 5 mmol/L; B: 10 mmol/L; C: 20 mmol/L. |
2.4 红球菌R04野生型菌株与缺陷型菌株R04ΔMP代谢联苯
将红球菌R04野生型和缺陷型菌株R04ΔMP分别接种于以联苯 (终浓度为10 mmol/L) 为唯一碳源和能源的基础培养基中,在9 h时开始检测到大量中间代谢产物的生成,因此取样从培养9 h时开始。从三角瓶中每隔1 h取1次样,测定总的代谢物浓度和细胞内的代谢物浓度,各测12个值,结果如图 6所示。在代谢物浓度的测定中,与其他代谢物相比,在细胞内检测到了较大量的联苯和苯甲酸,在总的代谢物中除了检测到联苯和苯甲酸,在一开始时还检测到少量的2, 3-二羟基联苯 (DHBP) (结果未显示)。图 6-A中缺陷型菌株细胞外的联苯浓度大于野生型,且缺陷型菌株细胞外的联苯浓度降低较慢,说明缺陷型菌株降解联苯较慢,RHOGL009301基因的缺失确实影响了红球菌R04对联苯的降解[16],这与生物量实验的分析一致;缺陷型菌株和野生型菌株细胞内联苯浓度相差不大,变化几乎一致,这可以说明RHOGL009301基因的缺失并没有影响细胞内联苯的运输[19-20];图 6-B中缺陷型菌株细胞外苯甲酸的浓度一开始低于野生型,后来与野生型达到相同;缺陷型菌株细胞内苯甲酸的浓度达到最大值比野生型晚,这些都说明缺陷型菌株降解联苯速度较慢,生成苯甲酸速度较慢,且缺陷型菌株胞内苯甲酸运输[21-22]速度也较慢。
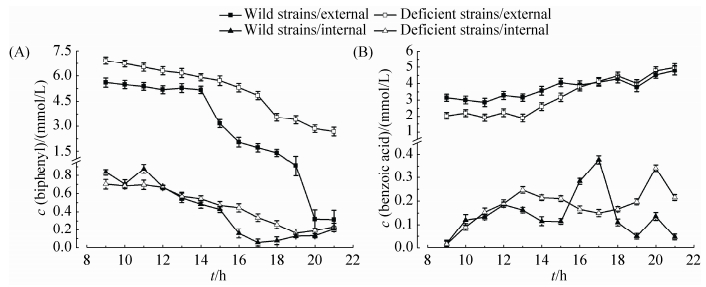
|
| 图 6. 野生型菌株和缺陷型菌株代谢联苯时细胞内外代谢物浓度变化 Figure 6. Internal and external metabolites of the wild strain and the deficient strain when grown on biphenyl. A: biphenyl; B: benzoic acid. |
2.5 红球菌R04野生型菌株与缺陷型菌株R04ΔMP代谢苯甲酸
将红球菌R04野生型和缺陷型菌株R04ΔMP分别接种于以苯甲酸 (终浓度为10 mmol/L) 为唯一碳源和能源的基础培养基中,取样从培养12 h时开始。从三角瓶中每隔1 h取1次样,测定总的苯甲酸浓度和细胞内的苯甲酸浓度,各测12个值,结果如图 7所示。细胞外的苯甲酸,缺陷型高于野生型,在测定的后期,野生型菌株细胞外的苯甲酸与第一次取样时相比几乎已利用完,而缺陷型菌株几乎没有利用。细胞内的苯甲酸,一开始时缺陷型菌株明显低于野生型,后来在野生型菌株利用完苯甲酸之后,缺陷型菌株细胞内苯甲酸浓度高于野生型。这表明缺陷型菌株降解苯甲酸速度较慢,且运输苯甲酸的速度也较慢。说明RHOGL009301基因的缺失影响了细胞内外苯甲酸的运输[21-22]。

|
| 图 7. 野生型菌株和缺陷型菌株代谢苯甲酸时细胞内外苯甲酸浓度变化 Figure 7. Internal and external benzoic acid of the wild strain and the deficient strain when grown on benzoic acid. |
2.6 红球菌R04野生型菌株与缺陷型菌株R04ΔMP对联苯和苯甲酸的共代谢
将红球菌R04野生型和缺陷型菌株R04ΔMP分别接种于以联苯 (终浓度为10 mmol/L) 为唯一碳源和能源的基础培养基中,在培养至9 h时加入苯甲酸 (终浓度5 mmol/L),之后每隔1 h从三角瓶中取1次样,测定总的代谢物浓度和细胞内的代谢物浓度,各测12个值,结果如图 8所示。在代谢物浓度的测定中,仍然以联苯和苯甲酸的量较多。图 8-A中缺陷型菌株细胞外的联苯浓度大于野生型,细胞内的联苯浓度几乎相同,与图 6-A相比,在9 h时加入苯甲酸后野生型菌株和缺陷型菌株细胞外的联苯被利用的速度都减慢,但细胞内的联苯没有明显影响,进一步说明RHOGL009301基因的缺失并没有影响细胞内联苯的跨膜运输[19-20],表明该膜蛋白[23-24]并不是联苯跨膜运输的转运蛋白。图 8-B中缺陷型菌株细胞外的苯甲酸在测定的12 h内几乎没有利用,而野生型菌株细胞外的苯甲酸浓度明显降低;细胞内的苯甲酸浓度一开始时野生型大于缺陷型,在测定的过程中,缺陷型菌株细胞内的苯甲酸也在增加,在后期,大于野生型细胞内苯甲酸。这是因为缺陷型菌株降解联苯慢,生成苯甲酸的速度慢,在加入等量的苯甲酸后,缺陷型菌株运输苯甲酸的转运蛋白减少,所以细胞外苯甲酸浓度减少较慢,细胞内苯甲酸浓度增加较慢。由此表明,膜蛋白[23-24]RHOGL009301可能参与细胞内苯甲酸的运输[21-22],缺陷型菌株降解联苯速度慢,正是因为联苯代谢的中间代谢产物苯甲酸运输的转运蛋白减少,从而影响了联苯的代谢。
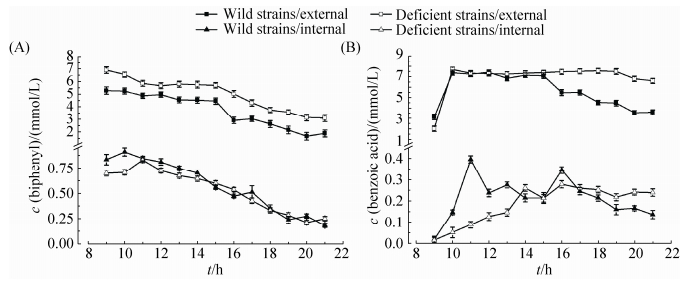
|
| 图 8. 野生型菌株和缺陷型菌株共代谢联苯和苯甲酸时细胞内外代谢物浓度变化 Figure 8. Internal and external metabolites of the wild strain and the deficient strain in mixed culture. A: biphenyl; B: benzoic acid. |
红球菌R04膜蛋白RHOGL009301缺陷型菌株代谢联苯、苯甲酸实验表明,该膜蛋白生理功能与苯甲酸的运输有关,是一种参与苯甲酸跨膜运输的转运蛋白;与之前文献报道的苯甲酸的转运蛋白[8-10]相比,RHOGL009301基因序列不同,蛋白质序列只有较低的相似性 (最高只有13%),说明此膜蛋白是一种新型的苯甲酸转运蛋白。
3 讨论红球菌R04能够利用联苯为唯一碳源生长,对芳香化合物来说,通过转运蛋白的作用跨膜进入细胞,是其降解过程的第一步;在代谢过程中生成的中间产物进出细胞也需要膜蛋白的协助[25-26]。目前在微生物体内发现了苯甲酸的转运蛋白BenE、BenK[8-10]等。
本研究选取的膜蛋白基因RHOGL009301在联苯培养条件下表达量最高,因此推测该膜蛋白可能在联苯的代谢过程中参与物质的运输。通过绿色荧光蛋白的融合表达定位,确定了该基因为膜蛋白基因。实验结果表明该基因缺失确实影响了红球菌R04降解联苯,具体表现为生长速度减慢,稳定期延后,生物量降低。但缺陷型菌株R04ΔMP仍然可以代谢联苯,且细胞内联苯的浓度没有明显变化,说明联苯跨膜进入细胞内并不需要该膜蛋白的转运,该膜蛋白并不是联苯运输的膜蛋白。研究发现缺陷型菌株R04ΔMP在代谢苯甲酸时生物量明显下降,而在代谢HOPDA时生物量与野生型红球菌R04基本一致,生长状况相近,由此我们推测缺陷型菌株R04ΔMP代谢联苯较慢可能是由于中间代谢产物苯甲酸的代谢和转运受到影响。
苯甲酸的降解途径不是单一的,红球菌R04代谢苯甲酸的途径有3条:原儿茶酸途径、儿茶酚的邻位和间位途径[27-30]。已有相关报道表明苯甲酸的转运系统也不止一种,在谷氨酸棒状杆菌中BenE和BenK双敲除的菌株通过诱导仍然可以转运、积累和利用苯甲酸[31]。红球菌R04基因组测序结果证实该菌株基因组中含有苯甲酸转运蛋白BenE和BenQ的编码基因[11],本研究中膜蛋白正是苯甲酸转运相关蛋白的其中一种。实验中我们只对膜蛋白RHOGL009301进行了基因敲除,BenE和BenQ仍然可以转运苯甲酸,所以缺陷型菌株R04ΔMP只是对苯甲酸的转运减少,对苯甲酸和联苯的降解速度减慢,但苯甲酸的代谢没有终止,当然也没有造成红球菌R04停止生长。随着联苯和苯甲酸浓度的增大,缺陷型菌株R04ΔMP生长速度越来越慢。HPLC结果也表明,缺陷型菌株R04ΔMP在代谢联苯和苯甲酸时,细胞外的苯甲酸减少较慢,细胞内的苯甲酸增加较慢,进一步验证了该膜蛋白是一种转运蛋白,与苯甲酸的跨膜转运相关。
本研究通过定位证明RHOGL009301基因为膜蛋白基因,对该基因进行敲除来研究红球菌R04膜蛋白的生理功能,确定了该膜蛋白是一种转运蛋白,与苯甲酸的跨膜运输相关。证明膜蛋白在物质进出细胞中的重要作用,对研究膜蛋白的生理功能具有重要意义,为苯甲酸等相关芳香化合物的代谢机理和转运机制研究提供基础和依据。
| [1] | Yang XQ, Sun Y, Qian SJ. Biodegradation of seven polychlorinated biphenyls by a newly isolated aerobic bacterium (Rhodococcus sp. R04). Journal of Industrial Microbiology and Biotechnology, 2004, 31(9): 415–420 DOI:10.1007/s10295-004-0162-5 . |
| [2] | Bedard DL, Haberl ML, May RJ, Brennan MJ. Evidence for novel mechanisms of polychlorinated biphenyl metabolism in Alcaligenes eutrophus H850. Applied and Environmental Microbiology, 1987, 53(5): 1103–1112 . |
| [3] | Omori T, Ishigooka H, Minoda Y. Purification and some properties of 2-hydroxy-6-oxo-6-phenylhexa-2, 4-dienoic acid (HOPDA) reducing enzyme from Pseudomonas cruciviae S93B1 involved in the degradation of biphenyl. Agricultural and Biological Chemistry, 1986, 50(6): 1513–1518 . |
| [4] | Harwood CS, Nichols NN, Kim MK, Ditty JL, Parales RE. Identification of the pcaRKF gene cluster from Pseudomonas putida: involvement in chemotaxis, biodegradation, and transport of 4-hydroxybenzoate. Journal of Bacteriology, 1994, 176(21): 6479–6488 DOI:10.1128/jb.176.21.6479-6488.1994 . |
| [5] | Caldwell ME, Suflita JM. Detection of phenol and benzoate as intermediates of anaerobic benzene biodegradation under different terminal electron-accepting conditions. Environmental Science & Technology, 2000, 34(7): 1216–1220 . |
| [6] | Zhang XY, Gai ZH, Tai C, Xu P. Advances in benzoic acid degradation by microorganism. Microbiology China, 2012, 39(12): 1808–1816 (in Chinese). 张晓云, 盖忠辉, 台萃, 许平. 微生物降解苯甲酸的研究进展. 微生物学通报, 2012, 39(12): 1808–1816. |
| [7] | Wehrmann A, Morakkabati S, Krämer R, Sahm H, Eggeling L. Functional analysis of sequences adjacent to dapE of Corynebacterium glutamicum reveals the presence of aroP, which encodes the aromatic amino acid transporter. Journal of Bacteriology, 1995, 177(20): 5991–5993 DOI:10.1128/jb.177.20.5991-5993.1995 . |
| [8] | Chaudhry MT, Huang Y, Shen XH, Poetsch A, Jiang CY, Liu SJ. Genome-wide investigation of aromatic acid transporters in Corynebacterium glutamicum. Microbiology, 2007, 153(3): 857–865 DOI:10.1099/mic.0.2006/002501-0 . |
| [9] | Zhan YH, Yu HY, Yan YL, Chen M, Lu W, Li SY, Peng ZX, Zhang W, Ping SZ, Wang J, Lin M. Genes involved in the benzoate catabolic pathway in Acinetobacter calcoaceticus PHEA-2. Current Microbiology, 2008, 57(6): 609–614 DOI:10.1007/s00284-008-9251-4 . |
| [10] | Jeffrey WH, Cuskey SM, Chapman PJ, Resnick S, Olsen RH. Characterization of Pseudomonas putida mutants unable to catabolize benzoate: cloning and characterization of Pseudomonas genes involved in benzoate catabolism and isolation of a chromosomal DNA fragment able to substitute for xylS in activation of the TOL lower-pathway promoter. Journal of Bacteriology, 1992, 174(15): 4986–4996 DOI:10.1128/jb.174.15.4986-4996.1992 . |
| [11] | Yang XQ, Xue R, Shen C, Li SR, Gao C, Wang Q, Zhao XX. Genome sequence of Rhodococcus sp. strain R04, a polychlorinated-biphenyl biodegrader. Journal of Bacteriology, 2011, 193(18): 5032–5033 DOI:10.1128/JB.05635-11 . |
| [12] | André B. An overview of membrane transport proteins in Saccharomyces cerevisiae. Yeast, 1995, 11(16): 1575–1611 DOI:10.1002/(ISSN)1097-0061 . |
| [13] | Vinothkumar KR, Henderson R. Structures of membrane proteins. Quarterly Reviews of Biophysics, 2010, 43(1): 65–158 DOI:10.1017/S0033583510000041 . |
| [14] | Yang XQ, Xi JW. Transcriptomic and benzoate metabolic pathways of Rhodococcus sp. R04 cultured in biphenyl. Acta Microbiologica Sinica, 2015, 55(7): 851–862 (in Chinese). 杨秀清, 席婧雯. 联苯培养条件下红球菌R04转录表达和苯甲酸代谢途径解析. 微生物学报, 2015, 55(7): 851–862. |
| [15] | Hayteas DL, Duffield DA. The determination by HPLC of PCB and p, p′-DDE residues in marine mammals stranded on the Oregon Coast, 1991-1995. Marine Pollution Bulletin, 1997, 34(10): 844–848 DOI:10.1016/S0025-326X(97)00045-3 . |
| [16] | Yang XQ, Zheng Y, Li PL, Wang JR. Influencing factor for the biodegradation of polyhalogenated biphenyls by Rhodococcus sp. R04. China Environmental Science, 2010, 30(5): 694–698 (in Chinese). 杨秀清, 郑媛, 李鹏丽, 王婧人. 红球菌-R04生物降解多卤代联苯的影响因素研究. 中国环境科学, 2010, 30(5): 694–698. |
| [17] | Kitagawa W, Miyauchi K, Masai E, Fukuda M. Cloning and characterization of benzoate catabolic genes in the gram-positive polychlorinated biphenyl degrader Rhodococcus sp. strain RHA1. Journal of Bacteriology, 2001, 183(22): 6598–6606 DOI:10.1128/JB.183.22.6598-6606.2001 . |
| [18] | Seah SYK, Labbé G, Nerdinger S, Johnson MR, Snieckus V, Eltis LD. Identification of a serine hydrolase as a key determinant in the microbial degradation of polychlorinated biphenyls. The Journal of Biological Chemistry, 2000, 275(21): 15701–15708 DOI:10.1074/jbc.275.21.15701 . |
| [19] | Ma WH, Zhao Z, Wang Y, Zhang YZ, Ding JY. Effect of aromatic amino acid transport gene knock-out on L-tryptophan accumulation in Corynebacterium pekinense PD-67. Acta Microbiologica Sinica, 2012, 52(11): 1344–1351 (in Chinese). 马温华, 赵智, 王宇, 张英姿, 丁久元. 北京棒杆菌芳香族氨基酸转运蛋白基因敲除对L-色氨酸积累的影响. 微生物学报, 2012, 52(11): 1344–1351. |
| [20] | Michalska K, Chang C, Mack JC, Zerbs S, Joachimiak A, Collart FR. Characterization of transport proteins for aromatic compounds derived from lignin: benzoate derivative binding proteins. Journal of Molecular Biology, 2012, 423(4): 555–575 DOI:10.1016/j.jmb.2012.08.017 . |
| [21] | Harayama S, Rekik M, Bairoch A, Neidle EL, Ornston LN. Potential DNA slippage structures acquired during evolutionary divergence of Acinetobacter calcoaceticus chromosomal benABC and Pseudomonas putida TOL pWW0 plasmid xylXYZ, genes encoding benzoate dioxygenases. Journal of Bacteriology, 1991, 173(23): 7540–7548 DOI:10.1128/jb.173.23.7540-7548.1991 . |
| [22] | Youn JW, Jolkver E, Krämer R, Marin K, Wendisch VF. Characterization of the dicarboxylate transporter DctA in Corynebacterium glutamicum. Journal of Bacteriology, 2009, 191(17): 5480–5488 DOI:10.1128/JB.00640-09 . |
| [23] | Li KW, Gu WP, Liang JR, Xiao YC, Qiu HY, Yang HS, Wang X, Jing HQ. Gene polymorphism analysis of Yersinia enterocolitica outer membrane protein A and putative outer membrane protein A family protein. BMC Genomics, 2014, 15(1): 201 DOI:10.1186/1471-2164-15-201 . |
| [24] | Emmerstorfer A, Wriessnegger T, Hirz M, Pichler H. Overexpression of membrane proteins from higher eukaryotes in yeasts. Applied Microbiology and Biotechnology, 2014, 98(18): 7671–7698 DOI:10.1007/s00253-014-5948-4 . |
| [25] | Cho W, Stahelin RV. Membrane-protein interactions in cell signaling and membrane trafficking. Annual Review of Biophysics and Biomolecular Structure, 2005, 34(1): 119–151 DOI:10.1146/annurev.biophys.33.110502.133337 . |
| [26] | Lino B, Chagolla A, de la Vara LEG. Membrane proteins involved in transport, vesicle traffic and Ca2+ signaling increase in beetroots grown in saline soils. Planta, 2016, 244(1): 87–101 DOI:10.1007/s00425-016-2488-4 . |
| [27] | Nishikawa Y, Yasumi Y, Noguchi YS, Sakamoto H, Nikawa JI. Functional analyses of Pseudomonas putida benzoate transporters expressed in the yeast Saccharomyces cerevisiae. Bioscience, Biotechnology, and Biochemistry, 2008, 72(8): 2034–2038 DOI:10.1271/bbb.80156 . |
| [28] | Chen DW, Zhang Y, Jiang CY, Liu SJ. Benzoate metabolism intermediate benzoyl coenzyme A affects gentisate pathway regulation in Comamonas testosteroni. Applied and Environmental Microbiology, 2014, 80(13): 4051–4062 DOI:10.1128/AEM.01146-14 . |
| [29] | Cao B, Geng AL, Loh KC. Induction of ortho-and meta-cleavage pathways in Pseudomonas in biodegradation of high benzoate concentration: MS identification of catabolic enzymes. Applied Microbiology and Biotechnology, 2008, 81(1): 99–107 DOI:10.1007/s00253-008-1728-3 . |
| [30] | Altenschmidt U, Oswald B, Steiner E, Herrmann H, Fuchs G. New aerobic benzoate oxidation pathway via benzoyl-coenzyme A and 3-hydroxybenzoyl-coenzyme A in a denitrifying Pseudomonas sp.. Journal of Bacteriology, 1993, 175(15): 4851–4858 DOI:10.1128/jb.175.15.4851-4858.1993 . |
| [31] | 王松鹤. 谷氨酸棒杆菌中苯甲酸转运蛋白研究. 中国科学院研究生院博士学位论文, 2011. |
 2017, Vol. 57
2017, Vol. 57