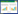中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 吕伟强, 刘聪, 黄丽丽, 颜霞. 2017
- Weiqiang Lü, Cong Liu, Lili Huang, Xia Yan. 2017
- 内生菌KM-1-2全基因组ORFs信号肽和分泌蛋白预测及功能分析
- Genome-wide prediction and analysis of the secretory proteins and ORFs signal peptide of ginkgo endophyte KM-1-2
- 微生物学报, 2017, 57(3): 411-421
-
文章历史
- 收稿日期:2016-08-03
- 修回日期:2016-09-28
- 网络出版日期:2016-12-02
2. 国家旱区作物逆境生物学国家重点实验室, 陕西 杨凌 712100;
3. 西北农林科技大学植物保护学院, 陕西 杨凌 712100
2. State Key Laboratory of Crop Stress Biology for Arid Areas, Yangling 712100, Shaanxi Province, China;
3. College of Plant Protection, Northwest A & F University, Yangling 712100, Shaanxi Province, China
Xia Yan, Tel:+86-29-87092262, E-mail:luckyx@126.com
分泌蛋白通常是由N端的信号肽指导通过细胞膜被运输到胞外的正确作用位点。根据信号肽学说,当内质网结合核糖体仍处于游离状态时,即已开始翻译多肽链,并在分泌蛋白N端形成一个长15-30个氨基酸的信号肽[1]。分泌蛋白应该具有以下特征:(1) 在其N-端均具有用于分泌至胞外的信号肽序列;(2) 无跨膜结构域;(3) 无GPI锚定位点;(4) 没有将蛋白输送至线粒体或其它胞内细胞器的预测定位信号[2]。满足以上4个标准的通常可定义为分泌蛋白编码基因。尽管不同的蛋白信号肽存在差异,但信号肽的基本结构是相似的。信号肽一般有3个明显的结构域,即N结构域、H结构域、C结构域。随着对信号肽研究的深入,人们根据信号肽的氨基酸组成及其所在位置将它划分为5种类型[3-7]:(1) 分泌信号肽;(2) RR-motif信号肽;(3) 脂蛋白信号肽;(4) Prepilin-like信号肽;(5) 细菌素和信息素信号肽。
国外学者利用生物信息学分析软件对杨叶锈菌 (Melampsora spp.)[8]和胚芽乳杆菌 (Lactobacillus plantarum)[9]中所含的分泌蛋白进行预测;国内学者则基于全基因组数据对多杀性巴氏杆菌[10]、禾谷炭疽菌[2]、西瓜果斑病菌[11]、香蕉细菌性软腐病菌[12]、玉米弯孢叶斑病菌[13]、大丽轮枝菌[14]、菜豆根瘤菌[15]、葡萄灰链霉菌[16]等分泌蛋白进行了预测。然而对植物内生放线菌分泌蛋白的研究尚未见报道,随着银杏叶内生菌KM-1-2全基因组序列的测定,对开展该菌分泌蛋白的预测提供了便利条件。外泌蛋白的分析,可以为研究微生物与植物之间、生防菌与病原菌之间互作提供参考。
本研究以银杏叶内生菌KM-1-2 5299条序列为基础,基于分泌蛋白所具有的主要特征,利用SignalP、PSORT、TMHMM、TargetP、Big-PI等生物信息学分析程序对分泌蛋白进行预测,探究分泌蛋白在内生过程中发挥的作用,以期为后续该菌与宿主的互作机制研究奠定基础。
1 材料和方法 1.1 数据获取本实验室前期分离并测序得到的KM-1-2 ORFs注释及功能信息,DDBJ/ENA/GenBank序列号MAUN00000000。其中KM-1-2共包含6906条编号以 > KM-1-2为起始的蛋白质编码ORFs;将其蛋白组信息保存在FASTA格式。
1.2 分泌蛋白确定方法 (图 1)
|
| 图 1. 分泌组预测流程 Figure 1. Flowchart of strategy used to identify secretome. |
1.2.1 N-端信号肽预测: 利用SignalP 4.1 (http://www.cbs.dtu.dk/services/SignalP/) 对蛋白质组是否有信号肽进行预测分析[17-20]。通过神经网络法进行N端信号肽及其剪切位点预测,包含5个得分参数,即C值 (剪切位点原始分值)、S值 (信号肽序列分值)、Y值 (C值几何均值与S值的联合得分值)、mean S (S值的算术均值)、D值 (mean S值与最大Y值的权重均值)。其中D值用于区分信号肽 (SP=‘YES’) 与非信号肽 (SP=‘NO’)。本研究使用参数D值“YES”,预测蛋白含有N端信号肽序列。SignalP是应用最广泛的预测软件之一,测试发现其对分泌蛋白的预测准确性达89%,因此可单独用于蛋白分泌组预测。: 1.2.2 跨膜区预测: TMHMM Server V.2.0 (http://www.cbs.dtu.dk/services/TMHMM/) 软件预测蛋白的跨膜螺旋结构,排除具有跨膜螺旋结构的蛋白[21]。预测蛋白跨膜螺旋的数量 (Number of predicted TMHs,PredHel) 和跨膜区氨基酸长度 (Exp number of AAs in TMHs,ExpAA),同时给出查询蛋白长度值 (len)、前60个氨基酸所预测跨膜螺旋数 (First60)、及拓扑结构 (Topology) 等参数。ExpAA长度大于18则证明有跨膜结构或者有信号肽[22]。: 1.2.3 胞外定位预测: PSORT[23] (http://psort.nibb.ac.ip/form.html) 软件确定这些分泌型信号肽的准确性以及该信号肽切割位点是否可被识别并切割。可预测蛋白定位,并对可能的定位情况进行打分,分值越高定位于该处的可能性越高。本研究使用extr值 > 17,预测蛋白定位于胞外。: 1.2.4 亚细胞定位预测: 利用TargetP 1.1 Server[24-26] (http://www.cbs.dtu.dk/services/TargetP/) 预测分泌蛋白进入植物后在植物细胞内的定位,明确哪些蛋白定位于胞外。通过预测的最高值与第二高值之间的差异分值 (diff) 进行可信度分区,1代表diff > 0.8,2代表0.8 > diff > 0.6,3代表0.6 > diff > 0.4,4代表0.4 > diff > 0.2,5代表diff < 0.2。本研究使用参数Loc值“S”,预测蛋白含信号肽序列并定位于胞外。: 1.2.5 GPI锚定位点预测: 利用big-PI Predictor (http://mendel.imp.ac.at/sat/gpi/gpi_server.html) 在线分析实现蛋白脂质锚定修饰的预测,去除锚定蛋白序列。本研究使用结果为None potential GPI-modification site,即无膜质结合位点的蛋白序列。: 1.3 分泌蛋白的功能预测
将获得的分泌蛋白通过NCBI protein BLAST (http://blas.tncb.inml.nih.gov/Blas.tcgi) 与数据库中已知蛋白进行相似性比对,预测分泌蛋白的可能功能,参数默认使用。使用Blast2go[27-28]程序分析酶学分类,GO注释。
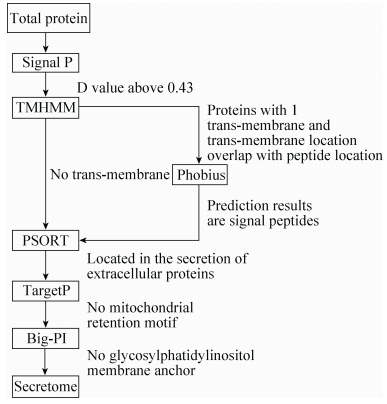
2 结果和分析 2.1 银杏叶内生菌KM-1-2分泌蛋白预测结果银杏叶内生菌KM-1-2共有5299条蛋白质序列,首先采用SignalP V4.1对全基因ORFs序列进行信号肽预测,结果表明338个序列在N端含有典型的信号肽序列,所占比例为7.32%。通过TMHMM V2.0在线分析上述蛋白的跨膜结构域,结果 (图 2) 显示有226个蛋白不含有跨膜结构域,所占比例为58.25%;162个蛋白含有不同数量的跨膜结构域,其中,含有1个跨膜结构域的蛋白有120条,所占比例为74.10%;含有2个跨膜结构域的为24条,所占比例为14.80%;其余含有多个跨膜结构域的序列共占比例11.10%。
|
| 图 2. 388个定位于胞外的蛋白跨膜区预测情况 Figure 2. The transmembrane prediction of 388 extracellular proteins. |
对于上述不含跨膜结构域的226条蛋白序列进行深入分析,排除跨膜结构域定位于膜内的5条序列,剩余的221条跨膜结构域均定位于膜外。对于只含1个跨膜域的蛋白质,其所具有的跨膜结构域位置均位于N端,而该区域可能为前期所预测的信号肽序列。由于TMHMM V2.0程序并不能完全对信号肽序列和所属跨膜区序列进行区分,可能误将N-端信号肽预测为跨膜区,造成预测的假阴性。鉴于此,此次研究将含有1次跨膜结构的120条序列提交于Phobius软件进行深入分析,将其预测含有信号肽的119条蛋白序列重新保留而进入后续的分析研究,降低了预测中假阴性的出现。
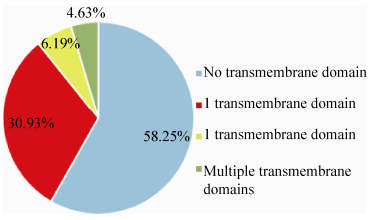
后续采用PSORT软件将上述得到的345条序列预测分析,结果分为细胞质型、细胞质膜型、胞外型3类。其中胞外型共149条,所占比例为43.20%。同时,使用Target PV1.1预测蛋白是否含有转运肽,从而明确蛋白质的细胞定位情况,可归属为线粒体目标肽、叶绿体转运肽以及分泌途径信号肽等,从而有助于将分泌目标为胞内细胞器 (线粒体、叶绿体等) 的蛋白进行排除。上述蛋白中,含有胞外定位信号、线粒体目标肽、其他定位信号所占比例分别为85.58%、12.75%、1.67% (图 3)。
|
| 图 3. 345个具有典型分泌特征的分泌蛋白的定位 Figure 3. Location of 345 secretory proteins with typical characteristics. |
利用big-PI Predictor对上述129条蛋白序列进行GPI锚定蛋白预测,由于GPI软件预测所需最短序列数为55,为保证分泌蛋白预测的准确性,所以排除1条少于55个aa的ORFs。对剩余的128条序列进行预测分析,结果显示128个蛋白序列均不具有GPI锚定位点。
通过综合SignalP和TargetP等诸多生物信息学在线预测分析菌株KM-1-2中5299条蛋白序列,最终获得128条具有4个典型特征的分泌蛋白。对上述分泌蛋白氨基酸长度进行分析,结果显示见图 4,这些分泌蛋白多集中于100-700 aa,所占比例高达90.60%。该结果表明KM-1-2分泌蛋白多属于小型蛋白,所含的氨基酸长度一般较小。
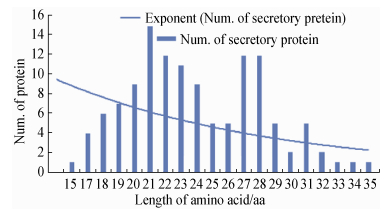
|
| 图 4. 分泌蛋白氨基酸长度分析 Figure 4. Analysis of secretory protein with different length of amino acid. |
2.2 银杏叶内生菌KM-1-2分泌蛋白信号肽特征分析
现已明确大多数物种的信号肽主要是通过5种类型的信号肽识别位点从而被信号肽酶所识别并被切割,从而使成熟蛋白穿过膜而转运至细胞的不同部位。本研究对128个分泌蛋白所含信号肽的氨基酸长度进行分析,结果显示 (图 5),含有信号肽长度为20-28个aa的蛋白序列数量最多,所占比例为70.80%。另外,采用LipoP V1.0对上述分泌蛋白进行信号肽酶识别位点的预测分析,结果显示,121个蛋白序列含有SPI型信号肽识别位点,4个含有细菌素-信息素信号肽 (CYT型),3个被预测为SPII。总体分析说明银杏叶内生菌中的分泌蛋白大部分是由SPI型信号肽酶进行识别,从而切除信号肽。
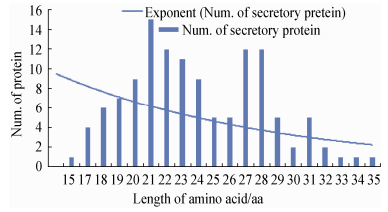
|
| 图 5. 分泌蛋白信号肽长度分析 Figure 5. Analysis of signal peptide length of secretory protein. |
通过对银杏叶内生菌分泌蛋白信号肽的切割位点-3位到+3位进行统计分析,结果显示 (表 1),位于-3、-2、-1、1、2、3位最多的氨基酸分别为A、A、A、A、A、A,所占比例分别为32.28%、40.94%、29.13%、29.13%、25.98%、36.22%。位于信号肽切割位点之前的-3位、-2位、-1位的氨基酸组成为A-A-A,属A-X-A类型,为SPI型信号肽识别位点,这与通过LipoP预测结果一致。
| Types of amino acids | Amino acid distribution in cleavage sites | |||||
| -3 | -2 | -1 | 1 | 2 | 3 | |
| A G L T V P S F Q R D E H I N M W C K Y |
32.28 14.17 13.39 11.02 10.24 7.87 2.36 1.57 1.57 1.57 0.79 0.79 0.79 0.79 0.79 0.00 0.00 0.00 0.00 0.00 |
40.94 11.81 9.45 7.87 7.09 4.72 9.45 0.79 3.15 0.00 1.57 0.79 0.00 0.00 0.79 0.79 0.79 0.00 0.00 0.00 |
29.13 10.24 16.54 9.45 7.09 7.09 3.94 0.79 3.15 0.00 3.15 1.57 0.79 0.79 0.00 2.36 0.00 1.57 1.57 0.79 |
29.13 12.60 11.02 8.66 5.51 9.45 4.72 0.79 2.36 1.57 3.15 1.57 3.15 1.57 1.57 0.79 0.79 0.00 0.79 0.79 |
25.98 11.02 8.66 11.81 5.51 12.60 6.30 0.79 1.57 2.36 1.57 0.79 2.36 2.36 0.00 1.57 1.57 0.00 1.57 1.57 |
36.22 6.30 11.02 8.66 5.51 8.66 2.36 1.57 1.57 2.36 2.36 4.72 3.15 0.00 1.57 0.00 1.57 0.79 0.79 0.79 |
同时,对组成蛋白质的20种氨基酸在信号肽中的分布情况进行统计分析见图 6,结果显示A所占比例最大,为25.81%,其次为L,所占比例为15.28%,T、V、G、R、S,所比例分别为10.04%、9.94%、7.63%、6.81%、5.28%。
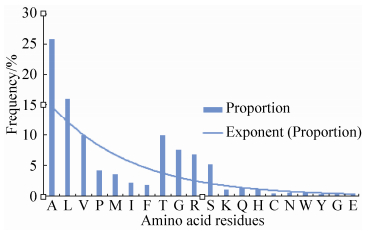
|
| 图 6. 20种氨基酸在分泌蛋白信号肽中所占比例情况 Figure 6. The frequency of 20 amino acid residues of signal peptides in secretory proteins. |
2.3 功能注释
在NCBI中通过Protein-BLAST将最后得到的128个分泌蛋白序列与已知蛋白进行同源比对。有2个蛋白未BLAST到任何蛋白 (1.0E-3),占所有蛋白数量的1.56%。比对的E-value数值分布在1.9E-179和3.2E-8之间。BLAST相似度分布在57.20%-100%,其中大于70%相似度的序列占87.50%,所以本研究中对蛋白质的功能鉴定有较高的可靠性。使用Blast2Go分析这些分泌蛋白的物种分布,其中所属物种数量较多的分别为Streptomyces sp. (51.60%)、Streptomyces xiamenensis(8.72%)、Streptomyces avicenniae(2.66%)、Streptomyces specialis(2.41%)。此外Blast2Go得到的Mapping和Annotation的蛋白分别为66和59条,占蛋白数量的51.50%和46.10%。
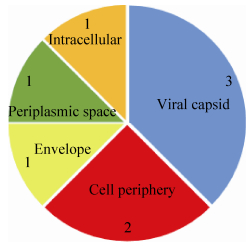
通过Blast2GO对蛋白序列进行分析,共有66个蛋白有GO注释,统计第4层GO数据,共得到294条GO术语信息,其中包含生物途径 (图 7)(biologic process) 104条,分子功能67条 (molecular function),细胞组件8条 (cellular component)。生物途径中最多的是大分子化合物代谢 (30条)、碳水化合物代谢 (20条)、有机物分解代谢 (13条)。分子功能 (图 8) 中最多的是水解肽酶活性 (14条)、丝氨酸蛋白酶活性 (14条)、蛋白酶活性 (8条)。在细胞组件 (图 9) 中最多的是病毒衣壳 (3条)、细胞外周 (2条)。
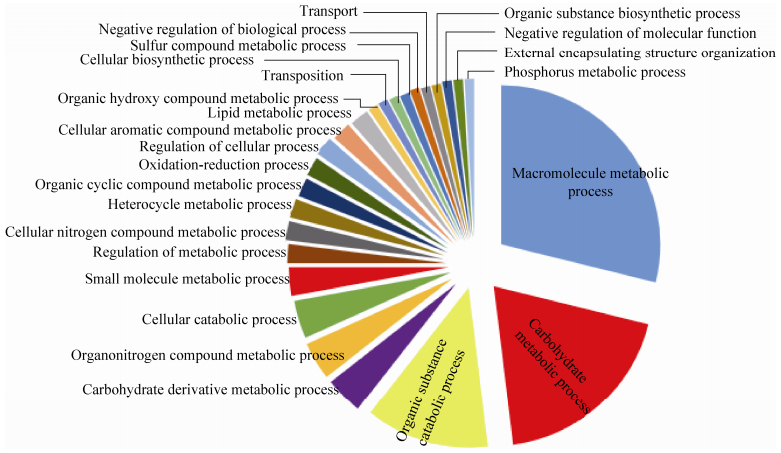
|
| 图 7. KM-1-2分泌蛋白组生物途径分析 Figure 7. Biological pathway analysis of KM-1-2 secretome. |

|
| 图 8. KM-1-2分泌蛋白组分子功能分析 Figure 8. Molecular function analysis of KM-1-2 scretome. |
|
| 图 9. KM-1-2分泌蛋白组细胞组件分析 Figure 9. Cells component analysis of KM-1-2 scretome. |
2.4 胞外酶的种类
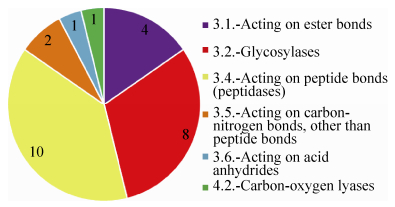
除重复与假定蛋白外,共得到26个蛋白属于酶类。这些酶类仅占所有预测的分泌蛋白总数的20.50%,它们的功能分布见图 10。
|
| 图 10. KM-1-2分泌蛋白组酶学分类分析 Figure 10. Scretome enzymology classification analysis. |
银杏叶内生菌KM-1-2胞外分泌酶类分布较为广泛,其中属于水解酶类 (EC 3.) 的比例最高,达到96%;氧化还原酶类和转移酶类分别占4.0%。未发现氧化还原酶类 (EC 1.)、转移酶类 (EC 2.)、异构酶类 (EC 3.) 和连接酶类 (EC 4.)。在水解酶类中EC 3.1.、EC 3.2.、EC 3.4.、EC 3.5.和EC 3.6.分别作用于酯键、糖苷键、碳氮键、羧酸酐键。
3 讨论分泌蛋白是指由细胞合成并分泌到胞外的蛋白质,可以分为两大类。一类是经典分泌蛋白 (classical secreted protein),另一类为非经典分泌蛋白 (non-classical secreted protein)。非经典途径分泌蛋白缺少常规的信号肽,不依赖内质网-高尔基体的膜分泌系统,目前对这类分泌蛋白进行预测的软件较少,常用的预测软件有SecretomeP 2.0,SOSUI-GramN,NClassG+可以分析非经典分泌蛋白[15]。但这类分泌蛋白只占少数,本研究主要考虑的是经典途径分泌的蛋白,通过蛋白N端信号肽来预测分泌蛋白。
对分泌蛋白信号肽分析和跨膜预测的软件众多,不同软件的算法不一致,这就导致了不同软件预测的分泌蛋白有一定的差别,采用多个软件对基因组蛋白同时分析,可以有效地提高预测的准确性。将个软件按照预测的结果来分类,可分为如下几类:(1) 蛋白信号肽及其切割位点预测,常见的软件如SignalP,其他功能类似的软件有Phobius,SigCleave,PrediSI等;(2) 跨膜蛋白预测,常见的软件有TMHMM,TMpred;(3) 蛋白亚细胞定位预测,常见的如ProtComp,TargetP,PSORT等。综合已有文献报道的分泌蛋白软件预测使用情况,SignalP对信号肽预测准确度为96%,TMHMM对跨膜蛋白预测的准确度高达97%,而TargetP对蛋白质亚细胞定位预测的准确度也高达90% (表 2)。本研究结合分泌蛋白的特征,采用6种生物信息软件结合预测,提高了预测的准确度,使得预测结果更为可靠。
| Prediction algorithms | Objects predicted | Accuracy/% | Applicable organisms |
| SignalP V4.1 | N-terminal signal peptides | 96 | Gram-positive bacteria Gram-negative bacteria Eukaryotes |
| TMHMM V2.0 | Transmembrane domains | 97-98 | Non-plant |
| PSORT | Prediction of protein localization sites | 86 | Gram-positive bacteria Gram-negative bacteria Yeast, Animal, Plant |
| Big-PI predictor | GPI-anchor site | > 80 | Metazoa, Protozoa |
| TargetP V1.1 | Mitochondrial or other localization sequence | 90 | Non-plant Plant |
采取组合法对KM-1-2的全基因组序列进行预测分析,共得到128条分泌蛋白序列,占全基因组的2.4%。通过对蛋白功能的预测进行分类,结果发现,KM-1-2胞外分泌蛋白含有丰富的糖苷水解酶、酯酶、蛋白酶等胞外水解酶系。分析KM-1-2不同类型的信号肽结构和数量,并将这些数据与其他细菌和真菌的分析结果进行比较 (表 3),在这4种菌中,均未发现Preplilin-like信号肽,而其它类型的信号肽含量均有明显差异。说明不同细菌或真菌的信号肽酶组成、蛋白分泌途径确实存在差异。
单纯地以试验手段去捕获生物中重要蛋白并进一步深入研究是十分有限的。通过生物信息学的手段可以高通量地获悉生物中某一类型的重要蛋白,并在此基础上进一步分选以获得感兴趣的蛋白,是一种快速而有效的方法。众多的软件已经被用于最初的对所有预测蛋白的功能分析,并已证实十分有效。
目前,已经有多个物种开展了分泌蛋白功能预测分析。唐雯等[29]发现里氏木霉分泌蛋白中64%是水解酶,包括糖苷酶 (38.78%)、蛋白酶 (14.29%) 和脂类水解酶 (3.74%) 等,其中糖苷水解酶占很大比例,包括很多与纤维素、几丁质等多糖降解相关的酶。王海霖等[17]发现灰葡萄孢菌367个蛋白质有功能描述,其中包括214个酶类,这些酶主要包括各种糖苷水解酶、酯酶、蛋白酶、多糖聚合酶、氧化还原酶、角质酶等。韩长志等[3]发现禾谷炭疽菌具有预测功能的蛋白为330个,其功能较多的集中于酶类,包括α-半乳糖苷酶、β-木聚糖酶、β-木糖苷酶等。本研究中,通过对生防放线菌KM-1-2分泌蛋白组功能预测,发现它们主要集中在糖苷水解酶、酯酶、蛋白酶等水解酶类,这与里氏木霉分泌蛋白的功能较为相似。内生菌分泌蛋白组中水解酶的存在是否与它的内生性有关还有待进一步实验的证明。
| [1] | Von Heijne G. Life and death of a signal peptide. Nature, 1998, 396(6707): 111–113 DOI:10.1038/24036 . |
| [2] | Han CZ. Prediction for secreted proteins from Colletotrichum graminicola genome. Biotechnology, 2014, 24(2): 36–41 (in Chinese). 韩长志. 全基因组预测禾谷炭疽菌的分泌蛋白. 生物技术, 2014, 24(2): 36–41. |
| [3] | Yan SM, Wu G. Signal peptide of cellulase. Applied Microbiology and Biotechnology, 2014, 98(12): 5329–5362 DOI:10.1007/s00253-014-5742-3 . |
| [4] | Zhang Y, Yang J, Liu L, Su Y, Xu L, Zhu YY, Li CY. Analysis of secretary proteins in the genome of the plant pathogenic fungus Botrytis Cinerea//Li DL, Liu YD, Chen YY. Computer and Computing Technologies in Agriculture Ⅳ:IFIP Advances in Information and Communication Technology. Berlin Heidelberg:Springer, 2010, 344:227-237. |
| [5] | Zhang SW, Zhang TH, Zhang JN, Huang YF. Prediction of signal peptide cleavage sites with subsite-coupled and template matching fusion algorithm. Molecular Informatics, 2014, 33(3): 230–239 DOI:10.1002/minf.v33.3 . |
| [6] | Ng SYM, VanDyke DJ, Chaban B, Wu J, Nosaka Y, Aizawa SI, Jarrell KF. Different minimal signal peptide lengths recognized by the archaeal prepilin-like peptidases FlaK and PibD. Journal of Bacteriology, 2009, 191(21): 6732–6740 DOI:10.1128/JB.00673-09 . |
| [7] | Tjalsma H, Bolhuis A, Jongbloed JDH, Bron S, Van Dijl JM. Signal peptide-dependent protein transport in Bacillus subtilis:a genome-based survey of the secretome. Microbiology and Molecular Biology Reviews, 2000, 64(3): 515–547 DOI:10.1128/MMBR.64.3.515-547.2000 . |
| [8] | Joly DL, Feau N, Tanguay P, Hamelin RC. Comparative analysis of secreted protein evolution using expressed sequence tags from four poplar leaf rusts (Melampsora spp.). BMC Genomics, 2010, 11: 422 DOI:10.1186/1471-2164-11-422 . |
| [9] | Mathiesen G, Sveen A, Brurberg MB, Fredriksen L, Axelsson L, Eijsink VGH. Genome-wide analysis of signal peptide functionality in Lactobacillus plantarum WCFS1. BMC Genomics, 2009, 10: 425 DOI:10.1186/1471-2164-10-425 . |
| [10] | Peng Z, Liang W, Liu WJ, Xu ZF, Tan C, Wu B, Zhou R, Chen HC. Prediction and analysis of secreted proteins encoding genes in genome of Pasteurella Multocida HN06. Progress in Veterinary Medicine, 2015, 36(12): 6–10 (in Chinese). 彭忠, 梁婉, 刘文静, 徐卓菲, 谭臣, 吴斌, 周锐, 陈焕春. 多杀性巴氏杆菌HN06基因组分泌蛋白的预测与分析. 动物医学进展, 2015, 36(12): 6–10. |
| [11] | Pan H, Gao TY, Wu MD, Yang L, Zhang J, Li GQ. Screening and functional prediction of type Ⅱ secretory proteins in Acidovorax citrulli AAC00-1. Journal of Huazhong Agricultural University, 2015, 34(3): 27–35 (in Chinese). 潘宏, 高天一, 吴明德, 杨龙, 张静, 李国庆. 西瓜果斑病菌T2SS分泌蛋白的筛选和功能预测. 华中农业大学学报, 2015, 34(3): 27–35. |
| [12] | Xiao WC, Fan HY, Bai TT, Wang W, Li HP. Prediction and analysis of the signal peptide and secreted proteins in soft rot bacteria XJ8-3-3 genome ORFs of banana. Journal of Fruit Trees, 2014, 31(6): 1057–1064 (in Chinese). 肖文超, 范会云, 白亭亭, 王婉, 李华平. 香蕉细菌性软腐病菌XJ8-3-3基因组中ORFs的信号肽及分泌蛋白功能预测分析. 果树学报, 2014, 31(6): 1057–1064. |
| [13] | Gao JX, Gao SG, Li YQ, Cheng J. Genome-wide prediction and analysis of the classical secreted proteins of Curvularia lunata. Journal of Plant Protection, 2015, 42(6): 869–876 (in Chinese). 高金欣, 高士刚, 李雅乾, 陈捷. 玉米弯孢叶斑病菌全基因组分泌蛋白的预测与分析. 植物保护学报, 2015, 42(6): 869–876. |
| [14] | Tian L, Chen JY, Chen XY, Wang JN, Dai XF. Prediction and analysis of Verticillium dahliae VdLs.17 secretome. Scientia Agricultura Sinica, 2011, 44(15): 3142–3153 (in Chinese). 田李, 陈捷胤, 陈相永, 汪佳妮, 戴小枫. 大丽轮枝菌 (Verticillium dahliae VdLs.17) 分泌组预测及分析. 中国农业科学, 2011, 44(15): 3142–3153. |
| [15] | Zhang W, Pan W, Ma JT, Guo T. Genome-wide prediction and analysis of the non-classical secreted proteins of Rhizobium etli CFN42. Journal of Dali University, 2015, 14(6): 45–48 (in Chinese). 张武, 潘伟, 马金田, 郭涛. 菜豆根瘤菌CFN42全基因组非经典分泌蛋白的预测与分析. 大理学院学报, 2015, 14(6): 45–48. |
| [16] | Wang HL, Ge WQ, Guo R, Suo YP, Cheng FS. Prediction and analysis of secretome proteins in Botrytis cinerea. Journal of Qingdao Agricultural University (Natural Science), 2015, 32(3): 174–179 (in Chinese). 王海霖, 葛文谦, 郭瑞, 索一平, 程凡升. 葡萄灰霉病菌胞外分泌蛋白质组的功能预测分析. 青岛农业大学学报 (自然科学版), 2015, 32(3): 174–179. |
| [17] | Petersen TN, Brunak S, Von Heijne G, Nielsen H. SignalP 4.0:discriminating signal peptides from transmembrane regions. Nature Methods, 2011, 8(10): 785–786 DOI:10.1038/nmeth.1701 . |
| [18] | Emanuelsson O, Brunak S, Von Heijne G, Nielsen H. Locating proteins in the cell using TargetP, SignalP and related tools. Nature Protocols, 2007, 2(4): 953–971 DOI:10.1038/nprot.2007.131 . |
| [19] | Melhem H, Min XJ, Butler G. The impact of SignalP 4.0 on the prediction of secreted proteins//Proceedings of the 2013 IEEE Symposium on Computational Intelligence in Bioinformatics and Computational Biology. Singapore:IEEE, 2013:16-22. |
| [20] | Mak MW, Wang W, Kung SY. Fusion of conditional random field and signalP for protein cleavage site prediction[C]//Proceedings of 2009 APSIPA Summit and Conference. Sapporo, Japan:APSIPA, 2009:716-721. |
| [21] | Chen YJ, Yu P, Luo JC, Jiang Y. Secreted protein prediction system combining CJ-SPHMM, TMHMM, and PSORT. Mammalian Genome, 2003, 14(12): 859–865 DOI:10.1007/s00335-003-2296-6 . |
| [22] | Xu C, Chen H, Gleason ML, Xu JR, Liu HQ, Zhang R, Sun GY. Peltaster fructicola genome reveals evolution from an invasive phytopathogen to an ectophytic parasite. Scientific Reports, 2016, 6: 22926 DOI:10.1038/srep22926 . |
| [23] | Nakai K, Horton P. PSORT:a program for detecting sorting signals in proteins and predicting their subcellular localization. Trends in Biochemical Sciences, 1999, 24(1): 34–35 DOI:10.1016/S0968-0004(98)01336-X . |
| [24] | Jaramillo VDA, Sukno SA, Thon MR. Identification of horizontally transferred genes in the genus Colletotrichum, reveals a steady tempo of bacterial to fungal gene transfer. BMC Genomics, 2015, 16: 2 DOI:10.1186/1471-2164-16-2 . |
| [25] | Klee EW, Ellis LBM. Evaluating eukaryotic secreted protein prediction. BMC Bioinformatics, 2005, 6: 256 DOI:10.1186/1471-2105-6-256 . |
| [26] | Emanuelsson O, Nielsen H, Brunak S, Von Heijne G. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence. Journal of Molecular Biology, 2000, 300(4): 1005–1016 DOI:10.1006/jmbi.2000.3903 . |
| [27] | Conesa A, Götz S, García-Gómez JM, Terol J, Talón M, Robles M. Blast2GO:a universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics, 2005, 21(18): 3674–3676 DOI:10.1093/bioinformatics/bti610 . |
| [28] | Conesa A, Götz S. Blast2GO:a comprehensive suite for functional analysis in plant genomics. International Journal of Plant Genomics, 2008, 2008: 619832 . |
| [29] | Tang W, Yan M. Prediction and analysis of the secreteomic in Trichoderma reesei. Acta Microbiologica Sinica, 2008, 48(4): 473–479 (in Chinese). 唐雯, 严明. 里氏木霉 (Trichoderma reesei) 分泌组的预测及分析. 微生物学报, 2008, 48(4): 473–479. |
| [30] | Liu YT, Li ZY, Zhu YY, Li CY, Li YZ. Analysis of the coding region for signal peptide-containing proteins in Pseudomonas syringae pv. tomato Genome. Hereditas (Beijing), 2005, 27(6): 959–964 (in Chinese). 刘雅婷, 李正跃, 朱有勇, 李成云, 李永忠. 植物病原细菌Pseudomonas syringae pv. tomato基因组中的信号肽分析. 遗传, 2005, 27(6): 959–964. |
| [31] | Huang JL, Wu JZ, Xiao CG, Li CJ, Wang GX. Analysis of signal peptides of the secreted proteins in Ralstonia solanacearum GMI1000. Hereditas (Beijing), 2007, 29(11): 1409–1416 (in Chinese). 黄俊丽, 吴金钟, 肖崇刚, 李常军, 王贵学. 植物病原细菌Ralstonia solanacearum GMI1000中分泌蛋白信号肽分析. 遗传, 2007, 29(11): 1409–1416. DOI:10.1360/yc-007-1409 |
 2017, Vol. 57
2017, Vol. 57