中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 姜雄健, 王前, 刘桂明, 赵大贺, 刘景芳, 向华. 2017
- Xiongjian Jiang, Qian Wang, Guiming Liu, Dahe Zhao, Jingfang Liu, Hua Xiang. 2017
- 地中海富盐菌PhaE蛋白乙酰化修饰对其功能的影响
- Acetylation regulates the function of PhaE in Haloferax mediterranei
- 微生物学报, 57(11): 1665-1675
- Acta Microbiologica Sinica, 57(11): 1665-1675
-
文章历史
- 收稿日期:2017-01-23
- 修回日期:2017-03-01
- 网络出版日期:2017-03-29
2. 中国科学院大学生命科学学院, 北京 100049
2. College of Life Sciences, University of Chinese Academy of Sciences, Beijing 100049, China
Hua Xiang, Tel/Fax: +86-10-64807472, E-mail:xiangh@im.ac.cn
蛋白质翻译后修饰,是指mRNA被翻译成蛋白质后,蛋白质上个别氨基酸残基被共价修饰的过程。它使蛋白质的结构更为复杂,功能更为完善,调节更为精细,作用更为专一[1]。迄今为止,已有400多种蛋白质翻译后修饰被发现(http://www.uniprot.org/docs/ptmlist),随着技术进步,越来越多的修饰种类被发现。其中研究较多的包括磷酸化、甲基化、泛素化、糖基化、乙酰化、琥珀酰化、亚硝基化等。早在20世纪60年代,真核生物组蛋白N末端赖氨酸乙酰化现象已被发现[2]。但直到21世纪初,这种修饰才引起人们的广泛关注。乙酰化主要发生在赖氨酸残基上,根据修饰位点的不同,赖氨酸乙酰化主要分成两类,一类是N末端乙酰化,乙酰基团位于赖氨酸α位碳原子的氨基上;另一类是内部赖氨酸乙酰化,乙酰基团位于内部赖氨酸ε位碳原子的氨基上。前者是一种不可逆修饰,与蛋白质的稳定性密切相关;后者是一种可逆修饰,在调节蛋白的结构和功能方面发挥重要作用[3]。
已有的乙酰化修饰组学研究表明,乙酰化修饰在真核和原核生物中均广泛存在,且在细胞的生命活动中发挥着重要的作用。在真核生物中,组蛋白的乙酰化能够促进DNA与组蛋白八聚体的解离,从而促进基因的转录与翻译过程[4]。在稳定期的大肠杆菌中,复制起始蛋白DnaA的乙酰化能够阻止其与ATP或者oriC的结合,抑制DNA的复制起始,调节复制起始的频率[5]。已有的研究表明,乙酰化修饰不仅参与DNA复制起始和基因转录的调控,在代谢过程中也发挥着重要的调控作用。在沙门氏菌中,人们发现,在不同的碳源中,乙酰化修饰通过调节中心代谢途径中代谢酶乙酰化水平的变化,调节细胞碳代谢流的方向[6]。相较于真核生物和细菌,人们对乙酰化修饰在古菌中的作用却知之甚少。
地中海富盐菌(Haloferax mediterranei)是一株分离自西班牙海水晒盐场的极端嗜盐古菌,它能够利用多种碳源合成生物可降解塑料PHA (Polyhydroxyalkanoate,聚羟基脂肪酸酯)。PHA是一种生物聚酯,主要作为胞内碳源、能源及还原力的贮藏性物质,在胞内以颗粒形式存在[7]。在PHA颗粒上结合着很多与PHA代谢相关的蛋白,本实验室前期研究表明,地中海富盐菌PHA颗粒共结合5种蛋白,它们的编码基因组成了pha基因簇(phaJ-phaR-phaP-phaE-phaC)。其中,PhaP是PHA颗粒的主要结构蛋白,PhaR是调控蛋白,它们的基因有8 bp的重叠且phaP与phaR共转录[8-9];PhaJ是与PHA降解相关的蛋白[10];PhaEC是PHA合成过程中关键的合酶,其中PhaC是合酶的大亚基,具有活性催化中心;PhaE则为小亚基,是该合酶不可或缺的组分,一般被认为可调节活性,但其调节机制尚不清楚[11]。
本研究利用高分辨率质谱对地中海富盐菌PHA颗粒结合蛋白的乙酰化修饰进行了鉴定,发现PHA合酶PhaEC的小亚基PhaE具有乙酰化现象,通过对其乙酰化位点的突变研究,初步探讨了PhaE乙酰化修饰对菌体生长、葡萄糖消耗以及PHA合成能力的影响,为深入研究乙酰化修饰调控PHA代谢的分子机制奠定了基础。
1 材料和方法 1.1 材料 1.1.1 引物设计合成及测序:根据GenBank登录的地中海富盐菌基因组序列,利用Primer引物设计软件设计特异性引物(表 1),由英潍捷基(上海)贸易有限公司合成。所构建突变敲除载体的测序由睿博兴科生物技术(北京)有限公司完成。| Primer | Sequence (5′→3′) |
| phaEC-DF3 | CAGGTCGACTCTAGAGCGATGGGTGACTTCC |
| phaEC-DR3 | AACCAGTACCGGTAAGGATAGCACAGCGAAA |
| phaEC-K105R-F1 | GCGAACGAGGCATTCCGCGAGGTGATGGGTACC |
| phaEC-K105R-R1 | ggtacccatcacctcgcggaatgcctcgttcgc |
| phaEC-K105Q-F1 | GCGAACGAGGCATTCCAGGAGGTGATGGGTACC |
| phaEC-K105Q-R1 | ggtacccatcacctcCTGgaatgcctcgttcgc |
| phaEC-K170R-F1 | CACGCCGTCGAGACGCGCCTCGACCGGCTCCTC |
| phaEC-K170R-R1 | Gaggagccggtcgaggcgcgtctcgacggcgtg |
| phaEC-K170Q-F1 | CACGCCGTCGAGACGCAGCTCGACCGGCTCCTC |
| phaEC-K170Q-R1 | gaggagccggtcgagctgcgtctcgacggcgtg |
| phaEC-VF2 | acgcagttcttccagttaca |
| phaEC-VR2 | cgttgtgaatgtacccgaag |
1.1.2 质粒与菌株:本实验所用质粒和菌株如表 2和表 3所示,大肠杆菌(Escherichia coli) JM109、110,地中海富盐菌(Haloferax mediterranei)野生型、部分敲除株,以及敲除质粒pHFX (Ampr)均由本实验室保存。其他phaE突变敲入载体及phaE突变株均为本研究中构建。
| Plasmid | Characteristics | Source or reference |
| pHFX | 4.0 kb, integration vector containing pyrF and its native promoter, Ampr | Liu, et al. 2011[12] |
| pDEC | 5.3 kb, integration vector of pHFX for knock-out of phaEC | Cai, et al. 2014[9] |
| pHFX-phaEC | 7.3 kb, integration vector of pHFX for cloning with phaC and phaE | This study |
| pK105R | 7.3 kb, pHFX-phaEC derived integration vector for knock-in of phaC and phaE (with the K105R mutation) into DFS-01 to generate E (K105R) | This study |
| pK105Q | 7.3 kb, pHFX-phaEC derived integration vector for knock-in of phaC and phaE (with the K105Q mutation) into DFS-01 to generate E (K105Q) | This study |
| pK170R | 7.3 kb, pHFX-phaEC derived integration vector for knock-in of phaC and phaE (with the K170R mutation) into DFS-01 to generate E (K170R) | This study |
| pK170Q | 7.3 kb, pHFX-phaEC derived integration vector for knock-in of phaC and phaE (with the K170Q mutation) into DFS-01 to generate E (K170Q) | This study |
| pK105/170R | 7.3 kb, pHFX-phaEC derived integration vector for knock-in of phaC and phaE (with the K105R/K170R mutation) into DFS-01 to generate E (K105/170R) | This study |
| pK105/170Q | 7.3 kb, pHFX-phaEC derived integration vector for knock-in of phaC and phaE (with the K105Q/K170Q mutation) into DFS-01 to generate E (K105/170Q) | This study |
| pTA03-5220 | 8.5 kb, expression vector of PhaE | This study |
| Strain | Characteristics | Source or reference |
| E. coli JM109 | Host for plasmid construction | Invitrogen |
| E. coli JM110 | Host for plasmid construction | Invitrogen |
| Haloferax mediterranei | Wild-type strain=ATCC 33500 | CGMCC |
| H. mediterranei DF50 | pyrF-deleted mutant of H. mediterranei | Liu, et al. 2011[12] |
| EPS | eps-deleted mutant of H. mediterranei DF50 | Liu, et al. 2015[10] |
| DFS-01 | phaE-deleted and phaC-deleted mutant of EPS | This study |
| E (K105R) | EPS strain with PhaE carrying a K105R mutation | This study |
| E (K105Q) | EPS strain with PhaE carrying a K105Q mutation | This study |
| E (K170R) | EPS strain with PhaE carrying a K170R mutation | This study |
| E (K170Q) | EPS strain with PhaE carrying a K170Q mutation | This study |
| E (K105/170R) | EPS strain with PhaE carrying a K105R/K170R mutation | This study |
| E (K105/170Q) | EPS strain with PhaE carrying a K105Q/K170Q mutation | This study |
| Haloferax volcanii H1424 | Haloferax host strain for protein expression | Stroud, et al. 2012[13] |
1.1.3 酶和试剂:用于PCR扩增的Taq DNA聚合酶购自生工生物工程(上海)股份有限公司,KOD plus高保真聚合酶购自TOYOBO公司(上海);DNA限制性内切酶、T4 DNA连接酶、T4 DNA聚合酶购自New England Biolabs公司;DNA marker (100 bp和1 kb)购自博迈德科技(北京)发展有限公司;质粒提取和DNA凝胶PCR产物回收试剂盒购自Axygen公司。
HisTrap HP 1 mL镍柱购自GE公司;超滤离心管(10 kDa)购自生工生物工程(上海)股份有限公司;2-D蛋白定量试剂盒购自GE公司,预染蛋白marker购自Thermo公司;碘乙酰胺购自GE公司;胰酶trypsin购自Roche公司。
1.1.4 培养基和溶液:嗜盐古菌常用培养基AS-168;特殊培养基YE–培养基(AS-168不加Yeast extract);PA培养基(g/L):NaCl 110.00,MgSO4·7H2O 29.52,MgCl2·6H2O 20.51,KCl 5.00,CaCl2 1.00,NH4Cl 2.00,微量元素溶液SL-6[14] 1 mL,柠檬酸铁铵8 mg,1, 4-piperazinediethanesulfonic acid (PIPES) 15.00,Glucose 10.00,pH值为7.2;YPC培养基(g/L):NaCl 144.00,MgCl2·6H2O 30.00,MgSO4·7H2O 33.00,KCl 4.20,CaCl2 0.33,Yeast extract 5.00,Peptone (soya) 1.00,Casamino acids 5.00,1 mol/L Tris-Cl (pH 7.5) 12 mL,pH值为7.5。蛋白纯化缓冲液A、B;以及PHA酯化液等按相关文献配制[10]。 1.2 菌体的培养将DF50在AS-168平板上划线培养后,挑取单克隆至10 mL AS-168液体培养基中培养,待培养2–3 d至稳定前期后以1:25的比例转移至150 mL PA培养基中,37、200 r/min培养18 h后收集菌体[15]。
1.3 PHA颗粒的提取采用蔗糖密度梯度离心法提取PHA颗粒[16]。收集生长至稳定期的菌体,用TBSL (g/L,NaCl 150,KCl 20,MgSO4·7H2O 5,Tris-Cl 100 mmol/L)冲洗2次后加入适量TBSL溶液重悬菌体,然后超声破碎菌体(250 W,3 s on/5 s off,30 min)。破碎后的菌液加入超离管中,依次在底部缓慢注入浓度为1.0、1.3、1.6 mol/L的蔗糖溶液(采用100 mmol/L pH 7.1的Tris-Cl缓冲溶液配制)。31000 r/min离心2.5 h后取出位于1.3 mol/L和1.6 mol/L蔗糖梯度之间的PHA颗粒层。
1.4 颗粒蛋白的分离将利用蔗糖密度梯度离心纯化的PHA颗粒,重悬于溶解缓冲液(6 mol/L尿素,2 mol/L硫脲),于4摇匀过夜,使蛋白充分溶解于缓冲液中。经11000 r/min离心10 min,转移上清至新EP管中并多次重复离心,以尽量除去不溶物。
1.5 PhaE蛋白乙酰化位点鉴定将洗脱的颗粒蛋白用10 K Nano Sep离心柱(Pall Life Sciences,Ann Arbor,MI,USA)离心浓缩,用2-D蛋白定量试剂盒进行定量。取300 μg颗粒蛋白利用10 K Nano Sep离心柱,13000 g离心脱盐,在该离心柱超滤膜[Ultra filtration (UF)]上37酶解过夜,酶量按50:1 (蛋白:酶)的比例添加,胰酶溶于20 mmol/L三乙基铵碳酸氢钠(Triethylammonium bicarbonate,TEAB)中。按100:1补加酶继续酶解3–4 h。离心收集酶解肽段。分别用50 μL 50 mmol/L TEAB和20 mmol/L TEAB洗涤滤膜2次,将3次离心所得酶解肽段合并,SpeedVac真空干燥。将样品溶于0.1%的甲酸(Formic acid)后于C18柱子(75 μm ID × 150 mm,2 μm,100 Å)进行分离,随后利用Thermo Orbitrap Fusion mass spectrometer (Thermo Fisher Scientific,Waltham,MA)进行MS和MS/MS分析[参数:MS分辨率(@ m/z 200) 120000;扫描范围(m/z) 350-1550;最大注射速率(ms) 50;AGC Target 2.00E+05。MS/MS:Fragmentation HCD:NCE (%) 35;Detector Type:Ion Orbitrap;AGC Target 1.00E+04;Max Injection (ms) 100;Dynamic Exclusion (sec) 60]。所得质谱数据利用Mascot (Matrix Science,London,UK;version 2.5) 软件对地中海富盐菌Uniprot蛋白质组数据库进行搜库,搜库参数:碎片离子分子量容忍度0.020 Da;母离子容忍度15.0 PPM。赖氨酸乙酰化、甲硫氨酸氧化、谷氨酰胺和天冬酰胺脱酰胺设定为可变修饰,肽段得分值设定为30。
1.6 PhaE蛋白乙酰化位点突变株的构建在pHFX-phaEC野生型敲入载体的基础上,设计突变引物(表 1)环式PCR扩增整个质粒。PCR产物经DpnⅠ酶消化后转化大肠杆菌JM109,挑取单克隆提取质粒后进行测序验证,验证成功后的突变型质粒转化大肠杆菌JM110,挑取阳性克隆培养后提取质粒(去甲基化,提高转化效率),然后将质粒转化DFS-01菌株,利用同源交换原理[12],首先通过同源臂(上游或下游)将整个质粒敲入基因组中,利用YE–培养基(尿嘧啶营养缺陷型筛选压力)筛选单交换菌株。将筛选得到的单交换菌株单克隆接入双交换筛选培养基即含尿嘧啶(50 μg/mL)及5-氟乳清酸(5-Fluoroorotic acid,FOA,250 μg/mL)的AS-168中,在反向筛选压力下同源臂(下游或上游)进行第2次交换,用验证引物(表 1)筛选双交换阳性克隆,得到PhaE乙酰化位点原位突变的目的菌株(表 3)。
1.7 PhaE蛋白突变株的生长测定挑取实验菌株单克隆至10 mL AS-168液体培养基中培养,2–3 d后以1:25的比例转移至150 mL PA发酵培养基中,在第0、12、24、36、48、72、96、120 h取1 mL菌液测定其OD600值。取生长至72 h的菌体,测定其OD600值,将野生型和突变株稀释至相同的OD600值后,对菌液进行连续梯度稀释,每个梯度取6 μL点在固体PA培养平板上,拍照监测菌体的生长变化。
1.8 PhaE蛋白突变株的PHA产量检测利用将PHA甲酯化的方法分析细胞积累PHA的含量及组分[10]。步骤如下:收集菌体,–70℃冻存3–4 h,迅速转移进行冷冻真空干燥,称取约80 mg左右的样品于酯化管中,加入2 mL酯化液(970 mL甲醇,30 mL浓硫酸,1 g苯甲酸)和2 mL氯仿,95℃烘箱处理4 h。冷却至室温后,加入1 mL ddH2O,振荡混匀后静置分层。取1 μL氯仿相进行气相色谱分析(GC-6820,Agilent)。PHBV标样购自Sigma公司。
1.9 PhaE蛋白突变株的葡萄糖消耗情况检测在上述培养过程中,在24、48、72、96、120 h取样,于12000 r/min离心5 min后,取上清用ddH2O稀释20倍。然后取25 μL稀释液进行葡萄糖浓度测定(SBA-40D型生物传感分析仪,山东省生物传感器重点实验室),1 g/L葡萄糖作为标样使用。
1.10 PhaE蛋白的异源表达与纯化将带有6*His Tag的PhaE蛋白表达载体PTA03-5220转入表达宿主Haloferax volcanii H1424[13]中。转化成功后在42、200 r/min的条件下在YPC液体培养基中培养至稳定期收集菌体。用适量裂解缓冲液重悬菌体,超声破碎(3 s on/5 s off,200 W,30 min),离心取上清,0.22 μm膜过滤。采用镍重力柱对目的蛋白进行挂柱回收,10倍体积清洗缓冲液(咪唑浓度20 mmol/L)清洗柱子,适量洗脱缓冲液(咪唑浓度1 mol/L)洗脱蛋白。将样品经SDS-PAGE分离后利用质谱鉴定目的蛋白[10]。
1.11 Western blot我们利用Western blot方法对颗粒蛋白上的PhaP与PhaE蛋白进行相对定量。PhaP、PhaE抗体均由遗传与发育研究所实验动物中心制作。取适量的PHA颗粒样品水浴煮沸,离心后取上清经SDS-PAGE分离后进行Western blot检测[15]。运用ImageJ软件对显影条带进行定量计算,以PhaP蛋白含量作为内参,对PhaE蛋白的含量进行相对定量。
2 结果和分析 2.1 PHA颗粒蛋白的分离及PhaE乙酰化位点的鉴定PHA颗粒蛋白经蔗糖密度梯度离心后,如图 1-A所示,在1.3 mol/L和1.6 mol/L浓度蔗糖溶液之间存在一个明显的白色颗粒层,即为PHA颗粒。将其收集后经6 mol/L尿素和2 mol/L硫脲洗脱收集颗粒蛋白。该蛋白混合物的酶解肽段经Nano-LC-MS/MS分析和Mascot搜库后,我们在PhaE蛋白上共鉴定到2个高度可信的乙酰化肽段,分别是DIWLNSANEAFK[Ac]EVMGTSAFAAATG QTVEDALEMQR和RQHAVETK[Ac]LDR。前一个肽段上的乙酰化位点为105位的赖氨酸(K[Ac]),后一个肽段上的乙酰化位点为170位赖氨酸(K[Ac])。图 1-B所示即为乙酰化肽段RQHAVETK[Ac]LDR的MS/MS鉴定图谱(含K170乙酰化位点)。

|
| 图 1 蔗糖密度梯度离心收集PHA颗粒和PhaE乙酰化肽段MS/MS鉴定图谱 Figure 1 MS/MS analysis of the acetylated peptide of the PHA-granule protein PhaE. A: PHA granules collected by sucrose density gradient centrifugation; B: MS/MS spectra of the acetylated peptide RQHAVETK[Ac]LDR of the PhaE. |
2.2 phaE基因突变株的构建
为了检测PHA合酶PhaEC中PhaE亚基K105及K170位点乙酰化修饰对该菌生理代谢的影响,我们对上述2个位点分别进行了单突变和双突变。利用表 1中的突变引物,以野生型的pHFX-phaEC质粒为模板,采用环式PCR的方法扩增,PCR产物经DpnⅠ消化转化大肠杆菌JM109,挑取转化子提取质粒进行测序验证,突变成功的转化子约占1/5,双突变型质粒在单突变型质粒基础上构建。得到质粒后,将质粒转化大肠杆菌JM110,得到阳性克隆后再提取质粒转化地中海富盐菌DFS-01,对转化子进行单、双交换筛选,用检测引物phaEC-VF2、phaEC-VR2 (表 1)进行验证,共得到6种PhaE突变株。其中,E (K105R)、E (K170R)、E (K105/170R) (表 3) 3个突变株将乙酰化的赖氨酸突变为精氨酸,模拟去乙酰化状态;E (K105Q)、E (K170Q)、E (K105/170Q) (表 3) 3个突变株将乙酰化的赖氨酸突变为谷氨酰胺,模拟乙酰化状态。用上述分别模拟去乙酰化和乙酰化状态的突变株,在后续的研究中,检测乙酰化修饰对菌体生理代谢的影响。
2.3 PhaE乙酰化位点突变影响菌体的生理代谢 2.3.1 PhaE乙酰化位点突变抑制菌体的生长:PhaE是PHA合成途径中的关键酶,它的突变可能会引起PHA合成情况的改变,而PHA又与细胞的物质和能量代谢密切相关,因此推测突变可能会影响菌体的生长和PHA的合成。本研究采用发酵培养基PA培养实验菌株,首先通过测定发酵液OD600的值来反映菌体的生长和PHA的合成情况。实验结果表明,K105和K170两个乙酰化位点的任何一种单点突变都不能对菌体OD600值造成明显影响(数据未展示),但将2个乙酰化的赖氨酸都突变成精氨酸时,如图 2-A所示,与野生型相比,从48 h开始,精氨酸双突变菌株E (K105/170R) OD600值增加明显减慢,在96 h时,E (K105/170R)突变株OD600值不足6,野生型OD600值已大于10。而谷氨酰胺双突变株E (K105/170Q) OD600值自24 h之后,已表现出显著的增加,且较野生型略高。说明PhaE去乙酰化显著抑制了菌体OD600值的增加。

|
| 图 2 野生型与突变型菌株生长情况测定 Figure 2 Cell growth comparison of the wild-type (strain EPS), E (K105/170R) and E (K105/170R) strains. A: the growth curves of the strains detected by OD600. The value of OD600 of the WT is more than 1.8-fold to the E (K105/170R) strain at 96 h (P < 0.01, n=3); B: growth detection by serial dilution spotting assay. Compared to the WT and E (K105/170Q), the growth of E (K105/170R) is a little bit slower. |
为了进一步验证双突变菌株是否影响菌体的生长,我们利用梯度稀释平板法检测了不同突变株的生长情况。如图 2-B所示,在固体培养基(PA)平板上,相同起始数量的菌株细胞(均约107),从稀释度10–4开始可以看出E (K105/170R)双突变株的生长慢于野生型和谷氨酰胺双突变株E (K105/170Q),特别是在稀释度10–5中,精氨酸双突变株E (K105/170R)的生长明显慢于野生型和谷氨酰胺双突变株E (K105/170Q)。说明PhaE去乙酰化确实抑制了菌体的生长。
2.3.2 PhaE乙酰化位点突变影响PHA的合成:由于菌体中PHA颗粒积累情况也会影响OD600值,我们推测精氨酸双突变株OD600值增加减缓(图 2-A)可能也与PHA合成有关,且PhaE是PHA合成的关键酶,因此我们进一步分析了突变株在培养过程中PHA含量的变化,以了解菌体中PHA的积累是否也是OD600值增加明显减慢的重要原因。如图 3所示,在发酵培养基PA中,与OD600值的变化趋势类似,48 h后,突变株E (K105/170R) PHA合成能力受到明显影响,在72 h时其PHA产量明显低于野生型菌株。96 h时,突变株E (K105/170R)积累的PHA仅为1.75 g/L,而野生型合成的PHA为4.0 g/L,约2.3倍于突变株E (K105/170R)。而谷氨酰胺双突变株E (K105/170Q)自48 h之后,其PHA合成呈显著增加的趋势,在96 h时也高达3.5 g/L,与野生型接近。由此可见,PhaE去乙酰化直接影响了PHA的合成能力(图 3),进而可能间接地影响了菌株的生长(图 2-B),因此应是二者的叠加效应减缓了突变株E (K105/170R) OD600值的增加(图 2-A)。

|
| 图 3 野生型与突变型菌株产PHA能力测定 Figure 3 Time courses of PHA accumulation in different strains. The PHA concentration of the WT (EPS strain) is more than 2.1-fold to the E (K105/170R) strain at 96 h (P < 0.01, n=3). |
2.3.3 PhaE乙酰化位点突变降低菌体葡萄糖消耗能力:在发酵培养基PA中,葡萄糖作为唯一的碳源,其消耗程度能够从侧面反映菌体生长及代谢的情况,且糖酵解的中间产物乙酰辅酶A是PHA合成的重要前体物质,因此通过检测菌体葡萄糖的消耗能力,不仅可以判断乙酰化修饰与菌体生长的相关性,也可进一步验证其对PHA合成的影响。由图 4可以看出,自48 h开始,突变菌株E (K105/170R)的葡萄糖利用速率明显低于野生型及另外一个突变株E (K105/170Q)。96 h时,野生型和谷氨酰胺双突变株E (K105/170Q)发酵液中葡萄糖含量已趋近于0,而突变株E (K105/170R)发酵液中葡萄糖含量仍有3 g/L,其发酵液中葡萄糖完全消耗殆尽,则需要7 d时间(数据未展示)。显然,突变株E (K105/170R)葡萄糖消耗速率远远低于野生型和谷氨酰胺双突变株。该结果进一步证明,PhaE去乙酰化影响了PHA合成,导致菌体生长减慢,同时其葡萄糖利用速度明显降低。

|
| 图 4 野生型与突变型菌株发酵液葡萄糖含量测定 Figure 4 Time courses of glucose consumption during 120-h fermentation. The WT (strain EPS) had almost used up the glucose while the E (K105/170R) still had about 3 g/L of glucose in the medium at 96 h (n=3). |
2.4 PhaE乙酰化位点突变影响PHA颗粒上PhaE含量
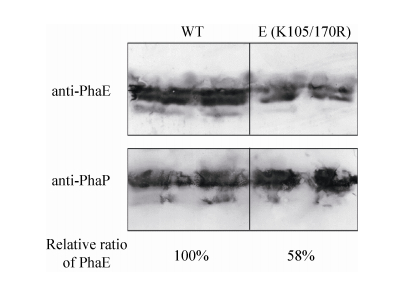
上述结果表明,PhaE去乙酰化影响了菌体对葡萄糖的利用以及PHA的合成,而PhaE是PHA合成中的关键酶,我们推测,正是因为PhaE乙酰化位点的突变,影响了PHA合酶PhaEC的活性,导致葡萄糖的代谢中间产物不能很好地转化为碳源贮藏物质PHA,从而减缓了葡萄糖的消耗速率。已有的研究表明,PhaEC合酶通过结合在PHA颗粒上发挥其PHA合成活性[11]。因此我们分别检测了野生型与突变株PHA颗粒上PhaE蛋白含量的变化。分别将野生型与突变株E (K105/170R)进行发酵培养,在48 h收集其PHA颗粒,取等量的PHA颗粒,利用Western blot分别检测PHA结构蛋白PhaP和PHA合成关键蛋白PhaE在PHA颗粒中的含量。如图 5所示(野生型和突变株均为2个泳道重复),在野生型和突变株中,在结构蛋白PhaP含量相当的情况下,突变株PHA颗粒上的PhaE蛋白显著降低,约为野生型PHA颗粒上所结合PhaE的58%。已有研究表明,赖氨酸的乙酰化修饰可中和赖氨酸所带的正电荷[16],因此我们推测去乙酰化(突变为精氨酸R)恢复了赖氨酸带正电的特性,从而影响了其与PHA颗粒的结合,而突变成谷氨酰胺(模拟乙酰化)对菌体的生理代谢无明显影响也从反面证实了这一推测。
|
| 图 5 野生型与突变型菌株PhaE与颗粒结合Western blot分析 Figure 5 Analysis of PhaE abundance on PHA granules by Western blot. WT: The EPS strain. |
3 讨论
地中海富盐菌全基因组测序表明,该菌中共有11个乙酰化酶基因和2个去乙酰化酶基因[17],远高于大多数原核生物中乙酰化酶和去乙酰化酶的数目,暗示乙酰化修饰在该菌中具有重要的生理功能。如前所述,PHA是在碳源过量的条件下,由菌体合成的一种作为胞内碳源、能源及还原力的贮藏性物质。地中海富盐菌可以从乙酰辅酶A和丙酰辅酶A前体物质出发,特异地积累聚羟基丁酸-羟基戊酸共聚酯[poly(3-hydroxybutyrate-co-3-hydroxyvalerate),PHBV][17]。乙酰化修饰也是以乙酰辅酶A为乙酰基供体,在乙酰化酶的催化下完成对底物的修饰[3]。众所周知,乙酰辅酶A是生物体内重要的物质和能量代谢的中间产物,在地中海富盐菌中,乙酰辅酶A既是PHA合成的前体物质,又是乙酰化修饰的乙酰基供体,因此,我们推测乙酰化修饰参与了PHA合成的调控。PhaEC合酶是PHA合成过程中的关键酶,PhaE是该合酶不可或缺的非催化亚基,推测PhaE对该合酶活性具有重要的调节作用,其赖氨酸位点的乙酰化修饰对该菌PHA合成的影响佐证了这一推测。
在沙门氏菌中,人们发现当以葡萄糖为唯一碳源时,中心代谢途径中的很多酶乙酰化水平显著提高,在葡萄糖浓度相同的情况下,敲除去乙酰化酶的突变株(乙酰化水平较野生型高)表现出更快的生长速度[6]。说明乙酰化修饰提高了中心代谢相关酶类的活性,促进了菌体利用葡萄糖的能力,从而加快了菌体的生长。在本研究中,PhaE两个乙酰化位点突变之后,菌体也表现出葡萄糖消耗能力降低、生长变慢、PHA合成能力下降的现象。该结果表明,当胞内碳源过剩(如乙酰-CoA含量高)时,PhaE的乙酰化修饰提高了PhaEC合酶的PHA合成活性,从而加速了菌体利用葡萄糖将过多碳源(如乙酰-CoA)转化为PHA的贮存过程,反之,去乙酰化则抑制了PHA的合成。
本研究Western blot结果表明,在等量的PHA颗粒上,乙酰化位点突变株E (K105/170R) PHA颗粒上PhaE蛋白的含量显著降低,仅为野生型结合PhaE的58% (图 5)。由于PhaEC合酶是通过结合在PHA颗粒上发挥活性,因此,我们推测PhaE蛋白的去乙酰化改变了其带电性,影响了PhaEC合酶与PHA颗粒或PhaC的结合,从而降低了其PHA合成活性,导致PHA产量降低。综上,本研究表明,PhaE的乙酰化修饰是PhaEC合酶活性调节的一种分子机制。
与细菌和真核生物相比,古菌域中乙酰化修饰的研究相对较少。本研究以嗜盐古菌中的模式菌株地中海富盐菌为研究对象,利用高分辨率质谱首次鉴定了PHA合成关键酶PhaEC中PhaE亚基的乙酰化修饰,并通过乙酰化位点突变,发现该蛋白的乙酰化修饰可调节PHA的合成。本研究首次从代谢方面解析了乙酰化修饰在古菌细胞活动中可能的生理功能,为古菌中乙酰化修饰的研究奠定了基础。
| [1] | Hart GW, Ball LE. Post-translational modifications:a major focus for the future of proteomics. Molecular & Cellular Proteomics, 2013, 12(12): 3443. |
| [2] | Phillips DMP. The presence of acetyl groups in histones. Biochemical Journal, 1963, 87(2): 258-263. DOI:10.1042/bj0870258 |
| [3] | Hentchel KL, Escalante-Semerena JC. Acylation of biomolecules in prokaryotes:a widespread strategy for the control of biological function and metabolic stress. Microbiology and Molecular Biology Reviews, 2015, 79(3): 321-346. DOI:10.1128/MMBR.00020-15 |
| [4] | Sterner DE, Berger SL. Acetylation of histones and transcription-related factors. Microbiology and Molecular Biology Reviews, 2000, 64(2): 435-459. DOI:10.1128/MMBR.64.2.435-459.2000 |
| [5] | Zhang QF, Zhou AP, Li SX, Ni JJ, Tao J, Lu J, Wan BS, Li S, Zhang J, Zhao SM, Zhao GP, Shao F, Yao YF. Reversible lysine acetylation is involved in DNA replication initiation by regulating activities of initiator DnaA in Escherichia coli. Scientific Reports, 2016, 6: 30837. DOI:10.1038/srep30837 |
| [6] | Wang QJ, Zhang YK, Yang C, Xiong H, Lin Y, Yao J, Li H, Xie L, Zhao W, Yao YF, Ning ZB, Zeng R, Xiong Y, Guan KL, Zhao SM, Zhao GP. Acetylation of metabolic enzymes coordinates carbon source utilization and metabolic flux. Science, 2010, 327(5968): 1004-1007. DOI:10.1126/science.1179687 |
| [7] | Anderson AJ, Dawes EA. Occurrence, metabolism, metabolic role, and industrial uses of bacterial polyhydroxyalkanoates. Microbiology and Molecular Biology Reviews, 1990, 54(4): 450-472. |
| [8] | Cai SF, Cai L, Liu HL, Liu XQ, Han J, Zhou J, Xiang H. Identification of the haloarchaeal phasin (PhaP) that functions in polyhydroxyalkanoate accumulation and granule formation in Haloferax mediterranei. Applied and Environmental Microbiology, 2012, 78(6): 1946-1952. DOI:10.1128/AEM.07114-11 |
| [9] | Cai SF, Cai L, Zhao DH, Liu GM, Han J, Zhou J, Xiang H. A novel DNA-binding protein, PhaR, plays a central role in the regulation of polyhydroxyalkanoate accumulation and granule formation in the haloarchaeon Haloferax mediterranei. Applied and Environmental Microbiology, 2015, 81(1): 373-385. DOI:10.1128/AEM.02878-14 |
| [10] | Liu GM, Hou J, Cai SF, Zhao DH, Cai L, Han J, Zhou J, Xiang H. A patatin-like protein associated with the polyhydroxyalkanoate (PHA) granules of Haloferax mediterranei acts as an efficient depolymerase in the degradation of native PHA. Applied and Environmental Microbiology, 2015, 81(9): 3029-3038. DOI:10.1128/AEM.04269-14 |
| [11] | Lu QH, Han J, Zhou LG, Zhou J, Xiang H. Genetic and biochemical characterization of the poly(3-Hydroxybutyrate-co-3-Hydroxyvalerate) synthase in Haloferax mediterranei. Journal of Bacteriology, 2008, 190(12): 4173-4180. DOI:10.1128/JB.00134-08 |
| [12] | Liu HL, Han J, Liu XQ, Zhou J, Xiang H. Development of pyrF-based gene knockout systems for genome-wide manipulation of the archaea Haloferax mediterranei and Haloarcula hispanica. Journal of Genetics and Genomics, 2011, 38(6): 261-269. DOI:10.1016/j.jgg.2011.05.003 |
| [13] | Stroud A, Liddell S, Allers T. Genetic and biochemical identification of a novel single-stranded DNA-binding complex in Haloferax volcanii. Frontiers in Microbiology, 2012, 3: 224-238. |
| [14] | Zhao DH, Cai L, Wu JH, Li M, Liu HL, Han J, Zhou J, Xiang H. Improving polyhydroxyalkanoate production by knocking out the genes involved in exopolysaccharide biosynthesis in Haloferax mediterranei. Applied Microbiology and Biotechnology, 2013, 97(7): 3027-3036. DOI:10.1007/s00253-012-4415-3 |
| [15] | Han J, Lu QH, Zhou LG, Zhou J, Xiang H. Molecular characterization of the phaECHm genes, required for biosynthesis of poly(3-hydroxybutyrate) in the extremely halophilic archaeon Haloarcula marismortui. Applied and Environmental Microbiology, 2007, 73(19): 6058-6065. DOI:10.1128/AEM.00953-07 |
| [16] | Wieczorek R, Pries A, Steinbüchel A, Mayer F. Analysis of a 24-kilodalton protein associated with the polyhydroxyalkanoic acid granules in Alcaligenes eutrophus. Journal of Bacteriology, 1995, 177(9): 2425-2435. DOI:10.1128/jb.177.9.2425-2435.1995 |
| [17] | Han J, Hou J, Zhang F, Ai GM, Li M, Cai SF, Liu HL, Wang L, Wang ZJ, Zhang SL, Cai L, Zhao DH, Zhou J, Xiang H. Multiple propionyl coenzyme A-supplying pathways for production of the bioplastic poly(3-hydroxybutyrate-co-3-hydroxyvalerate) in Haloferax mediterranei. Applied and Environmental Microbiology, 2013, 79(9): 2922-2931. DOI:10.1128/AEM.03915-12 |
 2017, Vol. 57
2017, Vol. 57




