中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 程妙文, 罗玮, 杜瑶. 2017
- Miaowen Cheng, Wei Luo, Yao Du. 2017
- CRISPR-CAS系统介导的新一代基因靶向修饰技术及其在工业微生物中的应用
- A new generation of targeted gene modification technology guided by CRISPR/CAS system and the application in industrial microorganism
- 微生物学报, 57(11): 1621-1633
- Acta Microbiologica Sinica, 57(11): 1621-1633
-
文章历史
- 收稿日期:2017-01-24
- 修回日期:2017-05-27
- 网络出版日期:2017-06-29
随着新一代基因测序技术的出现和发展,越来越多物种的全基因组序列将会被快速解析。面对海量的基因组数据,如何高效进行基因功能的确定和快速进化成为功能基因组学的重要研究内容。基因定点编辑技术是研究基因功能的一种基本策略,研究人员可以直接编辑或修饰DNA序列,迅速获得基因功能和相关信息,并使这些生物基因信息得以充分利用[1]。自1987年Thompsson[2]构建ES细胞基因敲除小鼠模型至今经过近30年的发展,基因组编辑技术日趋成熟从而得以广泛应用,并在2012年被Science评为十大重要科学进展之一。在基因组靶向修饰技术中,锌指核酸酶(Zinc-finger nucleases,ZFN)以及类转录激活因子效应物核酸酶(Transcription activator-like effector nucleases,TALEN)技术的应用较为广泛,取得了令人瞩目的成就。然而,两者在结合结构域的改造筛选以及组装都存在着较高的技术难度,并且筛选的工作量很大,成为限制其发展的瓶颈[3]。
近年来,广泛存在于细菌和古菌等原核生物中的一种获得性免疫系统CRISPR系统受到广泛关注,该系统能够特异性识别并结合噬菌体DNA,通过转录产物crRNA介导Cas蛋白识别并抵御外源性DNA[4]。根据这一原理构建的基因组靶向修饰技术被称为第三代基因组编辑技术,同ZFNs和TALEs技术相比,CRISPR/Cas9系统具有高效性、普适性、低成本,易操作等多种优点,将基因靶向修饰技术提高到了新的高度。
1 CRISPR/Cas 系统的结构和分类1987年,科学家在Escherichia coli K-12的碱性磷酸酶基因附近区域首次发现了成簇的规律间隔的短回文重复[5],2002年将其正式命名为Clustered regularly interspaced short palindromic repeats (CRISPR)。CRISPR/Cas系统是细菌针对病毒和噬菌体DNA入侵进化形成的一种获得性免疫系统。在人类已知的物种中,有40%细菌和将近90%古细菌的基因组或质粒中存在至少一个CRISPR位点[6]。CRISPR由一系列短的高度保守的正向重复序列(Repeats)与长度相似的间隔序列(Spacers)间隔排列组成。而CRISPR位点的数量存在物种差异性,CRISPR中重复序列的数量也随着不同物种而有所变化[7]。CRISPR位点大多定位在染色体上,只有个别出现在质粒中[8]。
一个典型的CRISPR/Cas基因座由一个编码Cas蛋白的操纵子以及一个重复-间隔(repeat-spacer)序列组成。如图 1所示,CRISPR位点为CRISPR基因座(CRISPR locus),基因座中包含有重复序列(Repeats)和间隔序列(Spacers),CRISPR位点与上游的前导序列(Leader sequence,LS),CRISPR功能相关的基因(CRISPR-associated genes,Cas genes)以及位于Cas蛋白编码基因上游tracrRNA编码基因共同构成了CRISPR系统。
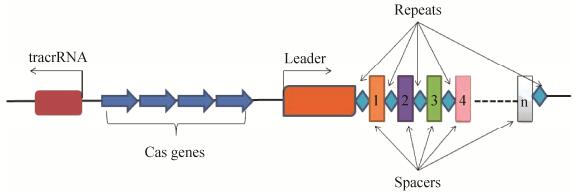
|
| 图 1 典型的CRISPR/Cas基因座结构 Figure 1 The structure of a typical CRISPR locus. |
重复序列(Repeats)和间隔序列(Spacers)是CRISPR序列中的主体,由简短稳定的同向重复序列(Direct repect,DR)和长度相近的非重复的间隔序列(Spacer)间隔排列而成,称为R-S结构。DR长度为21–48个碱基[6],在3′末端存在GAAA(C/G)的保守序列,能够形成稳定的茎环结构。间隔序列的长度在26–72 bp,不同CRISPR位点的间区序列数目变化很大,部分间区序列与噬菌体、大质粒等外源基因组以及同一细菌基因组的其他部分具有高度同源性[9]。CRISPR序列的形成来源于外源噬菌体的入侵,细菌会将一段外源核酸中的特殊片段整合成一段间隔序列,并在后续的免疫过程中发挥重要作用。除此以外,CRISPR/Cas系统中另外一种组分Cas基因是一类较大的多态性家族,编码的蛋白质具有与核酸结合的功能以及与核酸酶、聚合酶、解旋酶等结合的活性,Cas蛋白通过位点特异性切割将入侵的DNA切断。CRISPR位点的第一个重复序列上游有CRISPR前导序列(Leader sequence),该前导序列可以作为启动子启动CRISPR[10],转录产生的非编码RNA被命名为CRISPR RNAs (crRNAs)[8]。另外,在CRISPR基因座上游有一段保守序列转录生成的tracrRNA与crRNA的部分双链互补形成复合物,引导Cas蛋白到正确的位点进行靶序列切割,并且tracrRNA可协助Cas蛋白复合物剪切pre-crRNA形成成熟crRNA。
在噬菌体侵入的起始阶段,Cas蛋白复合物靶向裂解噬菌体基因组中短的原型间隔序列,这些原型间隔序列整合到宿主基因组中CRISPR位点的5′端,成为宿主基因组中间区序列的一部分,然后被插入的短间隔序列被转录形成crRNA。当噬菌体再次感染宿主时,crRNA和tracrRNA形成二元复合体指导Cas蛋白剪切靶向基因的特异位点。
根据参与CRISPR/Cas系统的Cas蛋白差异,该系统可分为三大类型:Ⅰ型、Ⅱ型和Ⅲ型。Ⅰ型系统广泛分布于细菌和古细菌中[11],是CRISPR/Cas系统中Cas蛋白最多和最复杂的系统,需要6个Cas蛋白参与,其中Cas3蛋白对CRISPR功能的发挥至关重要,该蛋白具有解旋酶活性及DNA酶活性,是干扰阶段的主要作用酶[12]。Ⅱ型系统的组成相对较简单,且只存在于细菌中[13],以Cas9蛋白为核心。RNAseⅢ目前已鉴定出2个Ⅲ型系统(Ⅲ-A型和Ⅲ-B型)[14],在Cas9蛋白存在的情况下发挥作用,Cas9蛋白协助crRNA (CRISPR RNA)的成熟,并参与外源DNA的剪切[15]。在一些Ⅲ型系统中,Cas6也参与crRNA的成熟,加工过后的crRNA被运送至特殊的Cas剪切复合体中发挥导向作用[16]。Cas6是CRISPR特异性的核糖核酸内切酶,而Cas10则与目标序列的干扰相关[15]。
2 CRISPR/Cas 系统的作用机制及应用于基因编辑CRISPR/Cas是一种防御系统,用以保护细菌和古细菌不受外来物质的侵害。由于Ⅱ型CRISPR/Cas免疫系统只需要1个核酸内切酶来切割DNA双链,因此作用机制和过程相对简单。目前只有类型Ⅱ的CRISPR/Cas系统应用于基因编辑或基因沉默。如图 2所示,Ⅱ型CRISPR/Cas系统发挥作用的流程大致分为3个阶段:获得阶段、表达阶段和干扰阶段。其中获得阶段指的是来源于病毒或质粒的小段DNA整合到细菌的CRISPR阵列中,产生一个新的间隔序列-重复序列单元,整合到CRISPR的5′端的2个重复序列之间[4],重复单元的获得是产生免疫机制的基础。在新间隔序列的获得过程中,细菌的Cas蛋白复合物识别位于DNA序列下游被称为PAM (Proto-spacer adjacent motifs)位点的特殊保守序列[17],将临近PAM的序列作为候选protospacer,然后在CRISPR基因座的5′端合成重复序列,最后新的间隔序列整合到2个重复序列之间。不同生物体中PAM元件是有差异的,在哺乳动物细胞中一般为NGG。PAM位点并不会被复制插入到间隔序列中,因此PAM位点的存在可能是CRISPR系统区分自身DNA与外源DNA而避免发生自身免疫的机制之一[18],同时也是基因编辑时靶序列选择的重要要求[19]。在表达阶段,CRISPR位点在前导序列的驱动下转录出前体CRISPR RNA (pre CRISPR RNA),在核酸内切酶作用下加工成包含不同的间隔序列和部分重复序列短的CRISPR RNA (crRNA)。干扰过程是CRISPR/Cas发挥抵御外源遗传物质入侵最关键的步骤,成熟的crRNA以及tracrRNA与特异的Cas9蛋白形成核糖核蛋白复合物,再与外源DNA结合并扫描外源DNA,寻找其上的靶序列,crRNA的间隔序列与靶序列互补配对,外源DNA在配对的特定位置被核糖核蛋白复合物切割。体外实验证明Cas9基因是参与CRISPR免疫系统的唯一必需基因[20],Cas9是由1409个氨基酸组成的多结构域蛋白,包括氨基端的RuvC-like结构域及位于蛋白中间位置的HNH核酸酶结构域[21]。HNH核酸酶结构域可以切割与crRNA互补配对的模板链,切割位点位于原型间隔序列毗邻基序(Protospacer adjacent motif,PAM)上游3 nt处,RuvC-like结构域可以对另一条链进行切割,切割位点位于PAM上游3–8 nt处[22]。在crRNA与tracrRNA形成的双链RNA的指导下,Cas9蛋白对靶位点进行切割。

|
| 图 2 CRISPR/Cas的作用机制[23] Figure 2 Action mechanism of CRISPR/Cas[23]. A: DNA double strand breaks (DSB) are repaired by homologous recombination (HR) or nonhomologous end-joining (NHEJ); B: The establishment of CRISPR/Cas9 immune system and three stages of which plays a role, that Cas9 protein shear target gene to produce DSB is the basis for gene editing. |
在免疫机制中,crRNA与tracrRNA形成二元复合物指导Cas9蛋白结合到靶位点然后进行特异性切割是细菌发挥免疫作用的关键。研究人员根据crRNA和tracrRNA的结构特征,设计出模拟二者结合后形成的二聚体结构的单链引导RNA (single guide RNA,sgRNA),sgRNA具备了crRNA-tracrRNA复合物的功能,能够与Cas9核酸内切酶结合并将后者引导至基因组上的靶位点处进行结合与切割。经过改造后的Cas9-sgRNA基因靶向修饰系统只需要将Cas9和sgRNA两种成分导入细胞即可实现基因组的靶向修饰作用[18]。
2012年,Jennifer Doudna和Emmanuelle Char-pentier以化脓性链球菌(Streptococcus thermophilus)的Ⅱ型CRISPR系统为基础进行的研究工作为促使CRISPR/Cas从细菌的天然免疫系统发展成为DNA编辑工具奠定了重要基础。Cas9蛋白在sgRNA的引导下导致目标DNA双链断裂(DSB),在同源重组(Homologous recombination,HR)和非同源末端连接(Non-homologous end joining,NHEJ)修复过程中实现对基因组的改造[24]。在进行同源重组修复时,细胞以含有同源臂的靶基因片段为基础与断裂双链进行结合,断裂处以靶基因上的DNA序列作为修复模板来恢复断裂前的序列,因此含有同源臂的外源基因就能整合到靶位点的DNA序列上。如果将人工合成的供体DNA转入细胞,那么将以该DNA为模板,可以提高定点修饰基因的效率。而非同源末端连接修复的保真性较弱,该机制往往导致DNA断裂处碱基的突变,多数情况下发生碱基缺失,这种错误的修复如果发生在基因的外显子上,能够引起该基因阅读框的改变,达到DNA定点突变而丧失基因功能的目的[25]。除了基因的插入和删除,利用CRISPR/Cas9系统还可对靶基因进行激活和抑制。CRISPR/Cas系统中Cas9具有2个核酸酶结构域(Ruv和HLH),如若在其中D10A和H840A位点发生突变,Cas9蛋白将会变成失去DNA切割活性的dCas9,但它依旧保留着与DNA结合的能力。在靶向基因gRNA同dCas9在细胞中共表达时,则sgRNA可以介导二者的结合。如果dCas9结合到靶基因的阅读框内,可阻断RNA聚合酶(RNA polymerase,RNAP)的延伸作用;如果dCas9结合到靶基因的启动子区域则可阻止基因转录的起始[26]。除基因抑制,dCas9蛋白还可以用来进行靶基因的激活,将dCas蛋白与具有转录激活的蛋白质功能域融合则可构建具有转录激活活性的CRISPR-on系统。在CRISPR转录激活系统中,靶序列位置对激活效率有重要影响。当靶序列与启动子的距离合适时,激活效率较高;靶序列处于启动子上游较远位置时,激活效率相对下降;当靶序列与启动子距离过近,或处于开放阅读框内时,则会产生抑制效应。
虽然CRISPR/Cas系统在近几年才开始被应用,但其发展却十分迅速,已经广泛运用于各种生物体中,它在基因编辑中也将有着更加广阔的发展空间。
3 CRISPR/Cas与ZFNs、TALENs的比较锌指核酸酶(Zinc-finger nucleases,ZFNs)、转录激活因子效应物核酸酶(Trans-cription activator like effector nucleases,TALENs)以及CRISPR/Cas系统都是目前能够进行基因编辑的工具。ZFNs和TALENs均是通过蛋白质的DNA结合域和FokI切割结构域形成二聚体来切割靶基因。ZFNs是将多个锌指串联形成的ZFP结构域与Ⅱs型限制性内切酶FokI[27]的切割结构域相连接来实现对靶序列的切割。增加串连锌指的数目可识别更长的靶序列,同时也就增强了DNA靶向修饰的特异性。但由于锌指结构α螺旋中碱基存在相互作用,ZFn也存在上下文依赖效应,且大多数研究者并不公布其ZF序列,导致对ZF识别的核苷酸序列研究不是完全明了,所以ZFn在构建、组装以及筛选方面难度很大。TALE核酸酶(TALEN)是由TALE代替了ZF作为DNA结合域与FokI切割结构域连接成核酸酶。通过TALE识别特异的DNA序列,FokI二聚化产生核酸内切酶活性,与ZFn一样在特异的靶DNA序列上产生双链断裂以实现精确的基因编辑[28]。TALE核酸酶比ZFNs切割的效率高,但其蛋白质的分子量较大,在模型的构建过程中存在不必要结构以及序列长度的影响,且分子操作的难度比较大,因此模型构建比较繁杂,可操控性较低,实验成本较昂贵。与前两种基因编辑工具相比,CRISPR/Cas系统只需设计针对靶基因序列互补的RNA以及向宿主细胞中导入编码Cas9蛋白的基因即可。CRISPR/Cas系统对靶序列的识别是RNA与DNA的碱基配对过程,对靶基因序列的识别更为精确,在切割时间以及空间的调节上也更为便利,并且识别位点多样化,不需形成二元复合物。而Cas9蛋白即可完成与FokI酶相似的功能,而且多个位点可一次性断裂,成功率高,对细胞毒性小[6, 11],这大大提高了基因操作的效率及简便性。因此,CRISPR/Cas系统与ZFNs和TALENs相比更为高效、简单和廉价,普通的实验室也可以自行完成构建,使得CRISPR/Cas系统具有更好的应用前景。
4 CRISPR/Cas 基因编辑技术在工业微生物中的应用基于CRISPR/Cas系统的基因编辑技术在最近几年日益成熟,这也促进了各国科学工作者将该技术应用于从原核生物到真核生物等各种生物基因组的改造之中。在哺乳动物细胞中,CRISPR/Cas技术和生物医学结合在一起,在人类遗传疾病、病毒感染和癌症治疗方面取得了重大突破。如在艾滋病治疗方面,将LTR-targeting CRISPR/Cas9复合物导入HIV病毒或者已经诱导的T细胞中,可以有效剪切或者突变LTR位点,从而导致HIV无法致病;更重要的是,LTR-targeting CRISPR/Cas9甚至能够剪切已经插入宿主染色体上的病毒基因。为了抑制HIV-1前病毒表达,TAR位点是最理想的靶基因,CRISPR/Cas系统的特异性很强,靶基因的1个碱基发生突变都有可能导致无法识别,而TAR位点相对比较保守而且在不同的HIV病毒亚类中也基本一致[29-31]。在植物以及光合微生物中,Myat T[32]利用CRISPR/Cas技术将烟草(Nicotiana tabacum)中原有的RuBisCo用细长集球藻(Synechococcus elongates)中的1, 5-二磷酸核酮糖羧化酶(RuBisCo)进行替换,为进一步在植物体内引入蓝细菌的二氧化碳富集机制,提高作物产量打下了基础。而在工业微生物领域,CRISPR/Cas系统已经广泛应用于微生物代谢工程,如改造菌株、解析基因功能或者改变代谢流。表 1列出了CRISPR/Cas9系统在各种模式生物中的应用。
| Species | Materials | Target genes | Repair approaches | References |
| Escherichia coli | DH5α, JM109 | rpsL, aroA | NHEJ, HR | [33] |
| Yeast | Saccharomyces cerevisiae | CAN1, LYP1 | NHEJ, HR | [34-35] |
| Yeast | Pichia pastoris | PMTs | NHEJ, HR | [36] |
| Flamentous fungi | Trichoderma reesei | URA5 | HR | [37] |
| Flamentous fungi | Neurospora crassa | clr‑2 promoter | HR | [38] |
| Fungus | Ustilago maydis | bE1, bW2 | NHEJ | [39] |
4.1 CRISPR/Cas技术在大肠杆菌中的应用
大肠杆菌是工业生物技术中的主要工程菌之一,并常常用于生产各种有价值的化学品、药物和生物燃料。有研究曾将内源性大肠杆菌DNA修复组分RecA、RecT和RecBCD应用于基因组工程,然而此内源性系统效率低下,需要较长的同源区并且不适用于对多位点进行重组[40]。在CRISPR/Cas系统[41]发现之后,将插入了可诱导的λ-Red重组酶基因的移动质粒与含有Cas蛋白编码基因以及供体模板基因的质粒导入大肠杆菌,可实现靶向多重基因组工程。随后对质粒进行不断构建筛选和优化,Zhao等通过一个质粒的构建和一步转化实现了对多基因位点的高效编辑[42]。夏军等利用二元载体构建CRISPR/Cas偶联λ-Red重组酶的系统,敲除大肠杆菌中的ppc和fad D基因,分别阻断了丙酮酸流向蛋白质合成的方向以及阻遏脂肪酸进入降解途径,使得大肠杆菌在脂肪酸工业生产中得到了更好的应用,并为构建其他工程菌提供了一定的理论基础。Li等将CRISPR/Cas技术作为基因编辑的工具运用到大肠杆菌中,通过插入β-胡萝卜素合成途径的通路相关基因以及优化2-C-甲基-赤藓糖醇-4-磷酸(MEP)途径和中心代谢途径实现了β-胡萝卜素的高量生产。与构建质粒介导过表达相比,这种基因整合方法稳定性更高且减少了代谢途径中的负担[43]。除在体外进行基因组装配外,Qi等[44]在大肠杆菌中对Cas9两个核酸酶结构域进行定点突变,得到仅保留有与DNA结合功能的dCas9蛋白,在sgRNA的引导下,使得dCas9结合在开放阅读框内,阻止了RNA聚合酶与DNA的结合,干扰转录的正常进行,导致基因沉默,调节基因表达。
在大肠杆菌中,余深翼[33]等利用CRISPR/Cas系统技术构建aroA基因的敲除系统,并分析其对不同大肠杆菌aroA基因敲除、修复的差异。构建靶向aroA基因且含有人工设计同源修复供体基因序列的CRISPR/Cas9载体,将该系统分别应用到大肠杆菌DH10B、DH5α和JM109细胞中,结果表明该系统能有效敲除3种大肠杆菌的aroA基因,敲除效率达到46%–58%。致病菌aroA基因缺失后,无法正常合成芳香类化合物,当其感染哺乳动物细胞后,生长受到限制且毒力下降。大肠杆菌aroA基因CRISPR/Cas9敲除系统的成功构建,不仅能让我们了解aroA基因的功能,还能为开发减毒大肠杆菌疫苗提供新型、有效的基因敲除工具。
4.2 CRISPR/Cas技术在酵母中的应用酿酒酵母、毕赤酵母是工业微生物代谢工程中使用较为密集的模式生物体之一,且因为与细胞周期、功能基因组学、蛋白质相互作用、癌症和其他重大疾病等研究密切相关从而成为优选模式生物体。DiCarlo等[45]首次在酿酒酵母中采用CRISPR-Cas9系统,他们在CAN1位点(编码精氨酸透过酶)重组90 bp寡肽DNA (包含上下游同源臂及终止密码子)及1.4 kb包含同源臂的Kan MX敲除盒,从而实现高效率的定点基因敲除(约100%),开启了CRISPR-Cas9系统在酵母领域应用新时代。随后,Zhang等[35]运用相同的系统在多倍体工业酵母ATCC 4124中逐步敲除LEU2、TRP1、URA3和HIS3,且效率达到15%–60%,最终成功构建四重缺陷菌株(Δura3,Δtrp1,Δleu2,Δhis3)。前期的研究为CRISPR-Cas9系统在酿酒酵母中的应用奠定了基础,但随着研究的发现,当把CRISPR-Cas9系统运用在独立单个基因编辑时,无法展现其独特优势。因此,利用该系统提高酿酒酵母多位点的同源重组效率开始得到广泛研究;同时,针对多个位点进行基因敲除的CRISPR-Cas9系统改造也应运而生。
在酿酒酵母中,Bao等[34]借助原始CRISPR阵列结构并整合供体DNA序列,构建了一步实现多基因位点敲除的CRISPR-Cas系统——HI-CRISPR (Homology-Integrated CRISPR——Cas),并成功在4天时间里同时敲除CAN1、ADE2及LYP1基因,敲除效率达到27%–87%,由此验证了此系统的有效性。CRISPR/Cas9系统在酿酒酵母中除了用于基因敲除外,还可以用于基因整合。Jakočiunas等[46]利用CRISPR-Cas9系统构建gRNA载体及重组元件(靶基因上下游同源区、启动子、终止子及待整合基因),在酿酒酵母的ADE2、HIS3、URA3位点分别整合了crtYB、crtI、crtE,利用新的Cas EMBLR的同源重组方法重新构建类胡萝卜素合成途径,且重组效率达30.6%,这种方法随后也应用于细菌中来高量生产芳香族类氨基酸。
2015年,毛银平[36]在毕赤酵母表达系统糖基化修饰改造的研究中,为解决酵母过度O-糖基化修饰的问题引入了dCas9蛋白。常规的基因敲除方式虽然可以阻断O-糖基转移酶基因PMTs的表达,但该基因对酵母生长是必不可少的,敲除致死将对酵母的生长产生影响。根据CRISPR/Cas9系统可对靶基因进行激活和抑制的优点,将Cas9蛋白突变成失去DNA切割活性却依旧保留着与DNA结合能力的dCas9,并由诱导型启动子D6控制诱导表达,结果表明该方式融合的CRISPR-dCas9鞭向阻遏系统无抑制O-糖基化的作用,但在一定程度上抑制了毕赤酵母的过度O-糖基化修饰。
CRISPR/Cas9系统在非传统酵母中也有一定的应用,解脂耶氏酵母(Yarrowia lipolytica)是研究最广泛的非常规酵母之一,它能够将糖分子转化为人工难以合成制造的脂质和碳氢化合物,但如何高效改造其基因组受到有限的基因编辑工具的制约。而CRISPR-Cas9基因编辑系统的开发为挖掘其生物制造潜力提供了一种新的工具。Schwartz等[47]对CRISPR-Cas9加以改造,并利用这个改造的系统成功地在解脂耶氏酵母中进行基因敲除以及将新的基因导入此酵母体内。CRISPR-Cas9系统的应用使得研究人员成功走出了在酵母体内制造长链碳氢化合物的第一步,为利用活的有机体生产生物燃料分子前体和专用聚合物奠定了基础。
4.3 CRISPR/Cas技术在丝状真菌中的应用除了酵母,CRISPR/Cas9技术在霉菌中也有一定的应用。Liu等[37]首次针对丝状真菌里氏木霉(Trichoderma reesei)改造了CRISPR-Cas9系统,与酿酒酵母不同,人类细胞的密码子优化的Cas9基因在组成型启动子Ppdc (pdc的启动子,编码丙酮酸脱羧酶的基因,参与葡萄糖的基因[48])或诱导型启动子Pcbh1 (cbh1的启动子,编码纤维二糖水解酶I的基因,受葡萄糖抑制,由一系列低聚糖或纤维素诱导[49])的控制下在里氏木霉Qm6a或Rut-C30中不起代谢作用。所以要采用Cas9密码子优化、体外转录gRNA及改造Cas9的组成型启动子为诱导性启动子,实现CRISPR-Cas9系统在丝状真菌中的可调控基因编辑。2015年,Liu等[37]将带有SV40核定位信号序列的酿脓链球菌(Streptococcus pyogenes) Cas9基因经过密码子优化,通过农杆菌介导的真菌转化,得到表达Cas9的菌株,再将体外合成gRNA装配到Cas9表达菌株中,同时转化携带与靶基因5′和3′侧翼序列同源的供体DNA,通过同源重组,达到基因置换的目的。研究中已经能够同时转化3个供体DNA,虽然同源重组率仅4.2%,但是能够说明CRISPR/Cas9同时定点插入多基因的可行性。2017年,Liu等[50]将CRISPR/Cas技术首次应用于可生产纤维素酶的真菌嗜热毁丝菌中,该研究首先利用此技术将amdS基因插入到野生型菌株中并利用筛选培养基来验证该体系在嗜热真菌中的有效性。随后,以嗜热毁丝菌中纤维素酶生产途径基因(cre-1、res-1、gh1-1和alp-1)作为靶向目标分别进行单基因以及多基因的敲除,得到高效生产纤维素酶的多种菌株,与亲本菌株相比,突变菌株细胞外分泌的蛋白质和木质纤维素酶活性显著增加(分别高达5和13倍)。此研究中CRISPR/Cas9系统的开发可优化嗜热真菌菌丝体物种的全基因组学代谢工程来高效生产木质纤维素酶和生物基燃料以及相关化学产品。
随后,利用CRISPR-Cas9系统在丝状真菌链孢霉(Neurospora crassa)中进行启动子替换及基因插入也得到广泛研究。在Matsu-Ura[38]等的研究中,运用CRISPR/Cas9系统在模式生物脉孢菌中进行有效基因的替换,利用Cas9核酸内切酶和单crRNA:tracrRNA嵌合指导RNA (gRNA),用β-微管蛋白启动子代替内源性clr-2启动子,显著增加了多种纤维素酶的表达。Nødvig等在进行野生棘孢曲霉(Aspergillus aculeatus)的基因编辑中,应用体外组装的办法对6个曲霉种(A. nidulans、A. aculeatus、A. niger、A. carbonarius、A. luchuensis、A. brasiliensis)进行了改造,验证了利用CRISPR/Cas系统进行基因编辑的高效性。
在探索丝状真菌的类胡萝卜素光诱导合成的分子遗传学机制时,本课题组利用split-marker策略敲除该基因,但与CRISPR/Cas9技术相比,该策略效率较低且分离纯合子较为繁琐,通过利用CRISPR/Cas9技术对该基因及光受体蛋白基因进行编辑将能有效解决上述问题。
除应用于工业微生物外,CRISPR/Cas系统还可用于构建模式生物以及进行生物信息学检索。在CRISPR/Cas系统中,细菌从入侵者那里捕获的DNA序列插入到自身重复序列之间形成间隔序列时,总是被整合到宿主基因组CRISPR位点的5′端。因此通过分析CRISPR位点中间隔序列的组成和排列顺序,可以用来对细菌分型。细菌的进化历程也可以反映在CRISPR位点间隔序列的多态性中,方便研究不同细菌之间的亲缘关系。此外,CRISPR/Cas系统还可以和现代分子学生物技术如高通量DNA测序结合来展开更为广阔的研究。
5 问题及展望基于CRISPR/Cas9系统的新型基因编辑技术的飞速发展为分子生物学的研究带来了无限的可能,然而其脱靶效应依旧是一个不容忽视的问题。CRISPR/Cas系统的特异性是由PAM前20 bp的RNA-DNA相互作用决定的,理论上CRISPR/Cas系统脱靶的概率会较高。虽然在细菌中CRISPR/Cas系统特异性很高,但研究表明利用CRISPR/Cas9系统对水稻的OsMPK2进行基因定点编辑时,发现了较多碱基的删除,经分析发现是sgRNA介导的CRISPR/Cas9系统分别对靶位点(On-target)以及与靶位点高度相似的序列(Off-target)分别进行了切割导致的[51]。因此,提高CRISPR/Cas系统的特异性来降低脱靶效应是大规模应用该技术的前提。脱靶效应主要与核酸酶结构以及靶基因的复杂性有关,因此通过改造其系统元件或采用不同策略能增加基因组编辑的特异性[52]。例如,Cas9-D10A切口酶配合一对sgRNA使用,可以减少意外的脱靶基因修饰,它分别在相对的两条DNA链上产生切口,从而形成功能性的双链断裂。这种设计能最大限度降低脱靶效应,同时保持高效而又特异的基因修饰。同时在设计sgRNA时,对于靶向基因特异性起决定性作用的12 bp可以含有2个碱基的错配[53]。此外,Cas9蛋白活性的时效性控制以及Cas9与sgRNA的比例也需要进一步的研究。在体外组装的质粒导入宿主菌株后,需平衡Cas9蛋白的活性和表达Cas9质粒的拷贝数。Cas9蛋白的高表达不仅将对细胞造成负担,并可能引起不希望看到的DSBs的脱靶效应,不论是在基因敲除还是基因插入中Cas9蛋白的高表达会影响转化后宿主菌的生长和恢复速率,在大多数情况下将对细胞造成致命的危害。另一方面,sgRNA的表达调节也会影响基因编辑的效率,因为Ryan等[54]曾报道了工业酵母菌株中启动子依赖性对基因编辑效率的影响。因此,我们在追求基因编辑的最优化时需要考虑宿主菌株的倍性水平,单倍体中获得的效率可能偏离多倍体菌株中的高水平表达。除脱靶效应以及表达量的控制外,CRISPR/Cas系统的许多分子机制尚不明确,如Cas蛋白是通过何种机制将获取的外源基因序列整合到自身基因座的5′端。且CRISPR/Cas9来源于细菌,对其研究依旧处于细胞阶段,且大部分都是应用于微生物中,那么当它应用于哺乳动物细胞时,是否会产生毒性或是否会诱发哺乳动物细胞或个体的免疫反应?
总之,CRISPR-Cas系统研究时间虽短,但是发展速度迅猛。相信在不久的将来,能发展成为一种操作方便、特异性强、切割效率高并能应用于各种生物的基因组定点修饰技术。
| [1] |
Pu Q, Luo J, Shen LY, Li Q, Zhang Y, Zhang SH, Zhu L. The advance and application of CRISPR/Cas9 mediated genome editing technique. China Biotechnology, 2015, 35(11): 77-84.
(in Chinese) 蒲强, 罗嘉, 沈林園, 李强, 张谊, 张顺华, 朱砺. CRISPR/Cas9基因组编辑技术的研究进展及其应用. 中国生物工程杂志, 2015, 35(11): 77-84. |
| [2] | Thomas KR, Capecchi MR. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell, 1987, 51(3): 503-512. DOI:10.1016/0092-8674(87)90646-5 |
| [3] | Gaj T, Gersbach CA, Barbas Ⅲ CF. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology, 2013, 31(7): 397-405. DOI:10.1016/j.tibtech.2013.04.004 |
| [4] | Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140 |
| [5] | Mahfouz MM, Li L, Shamimuzzaman M, Wibowo A, Fang X, Zhu JK. De novo-engineered transcription activator-like effector (TALE) hybrid nuclease with novel DNA binding specificity creates double-strand breaks. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(6): 2623-2628. DOI:10.1073/pnas.1019533108 |
| [6] | Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats. BMC Bioinformatics, 2007, 8: 172. DOI:10.1186/1471-2105-8-172 |
| [7] | Wei CX, Liu JY, Yu ZS, Zhang B, Gao GJ, Jiao RJ. TALEN or Cas9-rapid, efficient and specific choices for genome modifications. Journal of Genetics and Genomics, 2013, 40(6): 281-289. DOI:10.1016/j.jgg.2013.03.013 |
| [8] | Sorek R, Kunin V, Hugenholtz P. CRISPR-a widespread system that provides acquired resistance against phages in bacteria and archaea. Nature Reviews Microbiology, 2008, 6(3): 181-186. DOI:10.1038/nrmicro1793 |
| [9] | Mojica FJM, Díez-Villaseñor C, García-Martínez J, Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. Journal of Molecular Evolution, 2005, 60(2): 174-182. DOI:10.1007/s00239-004-0046-3 |
| [10] |
Li TM, Du B. CRISPR-Cas system and coevolution of bacteria and phages. Hereditas, 2011, 33(3): 213-218.
(in Chinese) 李铁民, 杜波. CRISPR-Cas系统与细菌和噬菌体的共进化. 遗传, 2011, 33(3): 213-218. |
| [11] | Gesner EM, Schellenberg MJ, Garside EL, George MM, Macmillan AM. Recognition and maturation of effector RNAs in a CRISPR interference pathway. Nature Structural & Molecular Biology, 2011, 18(6): 688-692. |
| [12] | Sinkunas T, Gasiunas G, Fremaux C, Barrangou R, Horvath P, Siksnys V. Cas3 is a single-stranded DNA nuclease and ATP-dependent helicase in the CRISPR/Cas immune system. The EMBO Journal, 2011, 30(7): 1335-1342. DOI:10.1038/emboj.2011.41 |
| [13] | Wiedenheft B, Lander GC, Zhou KH, Jore MM, Brouns SJJ, van der Oost J, Doudna JA, Nogales E. Structures of the RNA-guided surveillance complex from a bacterial immune system. Nature, 2011, 477(7365): 486-489. DOI:10.1038/nature10402 |
| [14] | Makarova KS, Haft DH, Barrangou R, Brouns SJJ, Charpentier E, Horvath P, Moineau S, Mojica FJM, Wolf YI, Yakunin AF, van der Oost J, Koonin EV. Evolution and classification of the CRISPR-Cas systems. Nature Reviews Microbiology, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [15] | Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao YJ, Pirzada ZA, Eckert MR, Vogel J, Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886 |
| [16] | Wang RY, Preamplume G, Terns MP, Terns RM, Li H. Interaction of the Cas6 riboendonuclease with CRISPR RNAs:recognition and cleavage. Structure, 2011, 19(2): 257-264. DOI:10.1016/j.str.2010.11.014 |
| [17] | Heidelberg JF, Nelson WC, Schoenfeld T, Bhaya D. Germ warfare in a microbial mat community:CRISPRs provide insights into the co-evolution of host and viral genomes. PLoS One, 2009, 4(1): e4169. DOI:10.1371/journal.pone.0004169 |
| [18] | Sternberg SH, Redding S, Jinek M, Greene EC, Doudna JA. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature, 2014, 507(7490): 62-67. DOI:10.1038/nature13011 |
| [19] | Semenova E, Nagornykh M, Pyatnitskiy M, Artamonova Ⅱ, Severinov K. Analysis of CRISPR system function in plant pathogen Xanthomonas oryzae. FEMS Microbiology Letters, 2009, 296(1): 110-116. DOI:10.1111/fml.2009.296.issue-1 |
| [20] | Sapranauskas R, Gasiunas G, Fremaux C, Barrangou R, Horvath P, Siksnys V. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli. Nucleic Acids Research, 2011, 39(21): 9275-9282. DOI:10.1093/nar/gkr606 |
| [21] | Westra ER, van Erp PBG, Künne T, Wong SP, Staals RHJ, Seegers CLC, Bollen S, Jore MM, Semenova E, Severinov K, de Vos WM, Dame RT, de Vries R, Brouns SJJ, an der Oost J. CRISPR immunity relies on the consecutive binding and degradation of negatively supercoiled invader DNA by Cascade and Cas3. Molecular Cell, 2012, 46(5): 595-605. DOI:10.1016/j.molcel.2012.03.018 |
| [22] | Friedland AE, Tzur YB, Esvelt KM, Colaiácovo MP, Church GM, Calarco JA. Heritable genome editing in C. elegans via a CRISPR-Cas9 system. Nature Methods, 2013, 10(8): 741-743. DOI:10.1038/nmeth.2532 |
| [23] | Jakociunas T, Jensen MK, Keasling JD. CRISPR/Cas9 advances engineering of microbial cell factories. Metab Eng, 2016, 34: 44-59. DOI:10.1016/j.ymben.2015.12.003 |
| [24] | Barrangou R. RNA-mediated programmable DNA cleavage. Nature Biotechnology, 2012, 30(9): 836-838. DOI:10.1038/nbt.2357 |
| [25] |
Wang X, Zhang ZQ, Zhang ZY. Genome targeting modification technology based on TALE nucleases engineering. Chinese Journal of Biochemistry and Molecular Biology, 2012, 28(3): 211-216.
(in Chinese) 王昕, 张志强, 张智英. TALE核酸酶介导的基因组定点修饰技术. 中国生物化学与分子生物学报, 2012, 28(3): 211-216. |
| [26] | Jinek M, East A, Cheng A, Lin S, Ma E, Doudna J. RNA-programmed genome editing in human cells. Elife, 2012, 2(2): e00471. |
| [27] | Bitinaite J, Wah DA, Aggarwal AK, Schildkraut I. FokI dimerization is required for DNA cleavage. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(18): 10570-10575. DOI:10.1073/pnas.95.18.10570 |
| [28] | Li T, Huang S, Zhao XF, Wright DA, Carpenter S, Spalding MH, Weeks DP, Yang B. Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes. Nucleic Acids Research, 2011, 39(14): 6315-6325. DOI:10.1093/nar/gkr188 |
| [29] | Ebina H, Misawa N, Kanemura Y, Koyanagi Y. Harnessing the CRISPR/Cas9 system to disrupt latent HIV-1 provirus. Scientific Reports, 2013, 3: 2510. DOI:10.1038/srep02510 |
| [30] | Goren M, Yosef I, Edgar R, Qimron U. The bacterial CRISPR/Cas system as analog of the mammalian adaptive immune system. RNA Biology, 2012, 9(5): 549-554. DOI:10.4161/rna.20177 |
| [31] | Mali P, Yang LH, Esvelt KM, Aach J, Guell M, Dicarlo JE, Norville JE, Church GM. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [32] | Lin MT, Occhialini A, Andralojc PJ, Parry MAJ, Hanson MR. A faster Rubisco with potential to increase photosynthesis in crops. Nature, 2014, 513(7519): 547-550. DOI:10.1038/nature13776 |
| [33] |
Yu SY, Zhao JR, Zheng LH, Zhu EP, Zhou WD, Wu BC. The application of CRISPR/Cas9 technology for aroA gene knockout in Escherichia coli. Acta Veterinaria et Zootechnica Sinica, 2016, 47(4): 762-770.
(in Chinese) 余深翼, 赵金荣, 郑玲红, 朱二鹏, 周五朵, 吴宝成. 利用CRISPR/Cas9技术构建大肠杆菌aroA基因的敲除系统及其初步应用. 畜牧兽医学报, 2016, 47(4): 762-770. |
| [34] | Bao ZH, Xiao H, Liang J, Zhang L, Xiong X, Sun N, Si T, Zhao HM. Homology-integrated CRISPR-Cas (HI-CRISPR) system for one-step multigene disruption in Saccharomyces cerevisiae. ACS Synthetic Biology, 2015, 4(5): 585-594. DOI:10.1021/sb500255k |
| [35] | Zhang GC, Kong Ⅱ, Kim H, Liu JJ, Cate JHD, Jin YS. Construction of a quadruple auxotrophic mutant of an industrial polyploid Saccharomyces cerevisiae strain by using RNA-guided Cas9 nuclease. Applied and Environmental Microbiology, 2014, 80(24): 7694-7701. DOI:10.1128/AEM.02310-14 |
| [36] |
Mao YP. Research of the glycosylation modifaction in Pichia pastoris expression system. Master Dissertation of Anhui University, 2015.
(in Chinese) 毛银平. 毕赤酵母表达系统糖基化修饰改造研究. 安徽大学硕士学位论文, 2015. |
| [37] | Liu R, Chen L, Jiang YP, Zhou ZH, Zou G. Efficient genome editing in filamentous fungus Trichoderma reesei using the CRISPR/Cas9 system. Cell Discovery, 2015, 1: 15007. DOI:10.1038/celldisc.2015.7 |
| [38] | Matsu-Ura T, Baek M, Kwon J, Hong C. Efficient gene editing in Neurospora crassa with CRISPR technology. Fungal Biology and Biotechnology, 2015, 2(1): 4. DOI:10.1186/s40694-015-0015-1 |
| [39] | Schuster M, Schweizer G, Reissmann S, Kahmann R. Genome editing in Ustilago maydis using the CRISPR-Cas system. Fungal Genetics and Biology, 2015, 89: 3-9. |
| [40] | He AS, Rohatgi PR, Hersh MN, Rosenberg SM. Roles of E. coli double-strand-break-repair proteins in stress-induced mutation. DNA Repair, 2006, 5(2): 258-273. DOI:10.1016/j.dnarep.2005.10.006 |
| [41] | Jiang Y, Chen B, Duan CL, Sun BB, Yang JJ, Yang S. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system. Applied and Environmental Microbiology, 2015, 81(7): 2506-2514. DOI:10.1128/AEM.04023-14 |
| [42] | Zhao DD, Yuan SL, Xiong B, Sun HN, Ye LJ, Li J, Zhang XL, Bi CH. Development of a fast and easy method for Escherichia coli genome editing with CRISPR/Cas9. Microbial Cell Factories, 2016, 15: 205. DOI:10.1186/s12934-016-0605-5 |
| [43] | Li YF, Lin ZQ, Huang C, Zhang Y, Wang ZW, Tang YJ, Chen T, Zhao XM. Metabolic engineering of Escherichia coli using CRISPR-Cas9 meditated genome editing. Metabolic Engineering, 2015, 31: 13-21. DOI:10.1016/j.ymben.2015.06.006 |
| [44] | Qi LS, Larson MH, Gilbert LA, Doudna JA, Weissman JS, Arkin AP, Lim WA. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [45] | DiCarlo JE, Norville JE, Mali P, Rios X, Aach J, Church GM. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research, 2013, 41(7): 4336-4343. DOI:10.1093/nar/gkt135 |
| [46] | Jakočiūnas T, Rajkumar AS, Zhang J, Arsovska D, Rodriguez A, Jendresen CB, Skjødt ML, Nielsen AT, Borodina I, Jensen MK, Keasling JD. CasEMBLR:Cas9-facilitated multiloci genomic integration of in vivo assembled DNA parts in Saccharomyces cerevisiae. ACS Synthetic Biology, 2015, 4(11): 1226-1234. DOI:10.1021/acssynbio.5b00007 |
| [47] | Schwartz CM, Hussain MS, Blenner M, Wheeldon I. Synthetic RNA polymerase Ⅲ promoters facilitate high-efficiency CRISPR-Cas9-mediated genome editing in Yarrowia lipolytica. ACS Synthetic Biology, 2016, 5(4): 356-359. DOI:10.1021/acssynbio.5b00162 |
| [48] | Li JX, Wang J, Wang SW, Xing M, Yu SW, Liu G. Achieving efficient protein expression in Trichoderma reesei by using strong constitutive promoters. Microbial Cell Factories, 2012, 11: 84. DOI:10.1186/1475-2859-11-84 |
| [49] | Ilmén M, Onnela ML, Klemsdal S, Keränen S, Pentilä M. Functional analysis of the cellobiohydrolase I promoter of the filamentous fungus Trichoderma reesei. Molecular and General Genetics MGG, 1996, 253(3): 303-314. |
| [50] | Liu Q, Gao RR, Li JG, Lin LC, Zhao JQ, Sun WL, Tian CG. Development of a genome-editing CRISPR/Cas9 system in thermophilic fungal Myceliophthora species and its application to hyper-cellulase production strain engineering. Biotechnology for Biofuels, 2017, 10: 165. DOI:10.1186/s13068-017-0852-7 |
| [51] | Shan QW, Wang YP, Li J, Zhang Y, Chen KL, Liang Z, Zhang K, Liu JX, Xi JJ, Qiu JL, Gao CX. Targeted genome modification of crop plants using a CRISPR-Cas system. Nature Biotechnology, 2013, 31(8): 686-688. DOI:10.1038/nbt.2650 |
| [52] | Shen B, Zhang WS, Zhang J, Zhou JK, Wang JY, Chen L, Wang L, Hodgkins A, Iyer V, Huang XX, Skarnes WS. Efficient genome modification by CRISPR-Cas9 nickase with minimal off-target effects. Nature Methods, 2014, 11(4): 399-402. DOI:10.1038/nmeth.2857 |
| [53] |
Wang MY, Yang YN, Bian HW. New genome targeting modification technology using a CRISPR-Cas system. Chinese Journal of Biochemistry and Molecular Biology, 2014, 30(5): 426-433.
(in Chinese) 王梦瑶, 杨亦农, 边红武. 基于CRISPR-Cas系统的基因组定点修饰新技术. 中国生物化学与分子生物学报, 2014, 30(5): 426-433. |
| [54] | Ryan OW, Skerker JM, Maurer MJ, Li X, Tsai JC, Poddar S, Lee ME, Deloache W, Dueber JE, Arkin AP, Cate JH. Selection of chromosomal DNA libraries using a multiplex CRISPR system. eLife Sciences, 2014, 3: e03703. |
 2017, Vol. 57
2017, Vol. 57




