中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 吴玉珍, 徐海津, 白艳玲, 张秀明, 乔明强. 2017
- Yuzhen Wu, Haijin Xu, Yanling Bai, Xiuming Zhang, Mingqiang Qiao. 2017
- CRISPR-Cas9系统与mazF介导的大片段删减法在酿酒酵母染色体大片段删减中的比较
- Comparison of CRISRP-Cas9 system and mazF-mediated method for large deletions in Saccharomyces cerevisiae
- 微生物学报, 57(11): 1604-1611
- Acta Microbiologica Sinica, 57(11): 1604-1611
-
文章历史
- 收稿日期:2016-11-15
- 修回日期:2017-02-22
- 网络出版日期:2017-02-27
2. 南开大学分子微生物学与技术教育部重点实验室, 天津 300071
2. Key Laboratory of Molecular Microbiology and Technology, Ministry of Education, Nankai University, Tianjin 300071, China
酿酒酵母作为真核模式生物,由于具有繁殖快、无致病性、遗传背景清晰等优势,多年来被广泛应用于工业生产及生物学研究[1]。基于合成生物学中底盘细胞的概念,在酿酒酵母基因组精简的过程中[2],染色体大片段删减成为一种必要的手段。在酿酒酵母中删减染色体大片段,染色体裂分技术[3]、Cre-loxP系统[4]以及Latour法[5]都是比较成熟的常用手段。然而这几种方法在打靶效率、同源区残留问题以及操作简便性等方面都有待提高。近年来,利用CRISPR-Cas系统进行基因编辑已成为研究热点,在大肠杆菌[6]、酿酒酵母[7-9]、大米[10]、小鼠[11]等生物中均有报道。
CRISPR-Cas系统是细菌和古生菌利用RNA引导的核酸酶来抵御病毒、质粒等外源入侵核酸的免疫系统,CRISPR全名为成簇的规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats),Cas为CRISPR相关蛋白(CRISPR -associated proteins)。CRISPR-Cas系统分为3个主要类型及11个亚型[12]。其中Ⅱ型CRISPR-Cas系统中以Cas9为标志蛋白。CRISPR-Cas系统发挥作用靠RNA复合物的引导作用以及Cas9核酸酶的剪切作用。其中RNA复合物由CRISPR RNA (crRNA)和反式激活CRISPR RNA (tracrRNA)组成,crRNA包含了20–30 bp的靶序列以及能与tracrRNA结合的序列,二者结合成能够被Cas9核酸酶识别的RNA二聚体复合物,进而引导Cas9核酸酶切割邻近PAMs的靶序列。目前广泛应用的是简化的Ⅱ型CRISPR-Cas系统,该系统中的gRNA由crRNA以及确定的tracrRNA融合而成,应用时只需更换gRNA 5′端20 bp的靶序列即可[13]。需要注意的是,选择的靶序列一定要紧挨着PAM序列(一般为NGG形式)的5′端。
Cas9核酸酶介导的DNA双链断裂可以通过非同源末端连接(non-homologous end joining,NHEJ)和同源重组方式(homology-directed repair,HDR)两种手段进行修复。NHEJ方式可能会导致双链断裂位置不同长度的插入或缺失突变;HDR方式则可以通过基因组DNA与DNA修复模板的同源重组进行精确的点突变或外源基因插入。
mazF基因是来源于大肠杆菌中编码核酸内切酶的毒力基因,MazF能够特异性切割游离mRNA的ACA位点,因此MazF蛋白的表达能够导致原核及真核细胞的程序性死亡[14]。本实验中即利用mazF该特点,将其作为负筛标记进行大片段删减及筛选标记回收。
本文通过利用CRISPR-Cas系统以及mazF介导的大片段删减方法同时对酿酒酵母中的染色体大片段进行删减,将两种方法的转化效率、删除成功率以及操作简便性等方面进行了比较,为以后在酿酒酵母中进行大片段删减的方法选择提供了依据。
1 材料和方法 1.1 材料 1.1.1 菌种、质粒和引物:本研究中用到的菌株和质粒如表 1所示,本研究中用到的引物由金唯智生物科技(北京)有限公司合成,信息如表 2所示。| Strains or plasmids | Characteristics | Source |
| BY4741 | MATa; his3Δ1; leu2Δ0; ura3Δ0; met15Δ0 | EUROSCARF |
| IMX581 | MATa; ura3-52 can1_::cas9-natNT2; TRP1; LEU2; HIS3 | EUROSCARF |
| 41-cas9 | MATa; his3Δ1; leu2Δ0; ura3Δ0; met15Δ0 can1_::cas9-LEU2 | This study |
| pUG73 | Template for LEU2 | EUROSCARF |
| pROS10 | 2 μm ampR URA3 gRNA-CAN1.Y gRNA-ADE2.Y | EUROSCARF |
| pROS10-11L | 2 μm ampR URA3 gRNA-STB6 gRNA-BLI1 | This study |
| pGREG506-mazF | Template for mazF | This study |
| Primers | Sequences (5′→3′) | Target genes |
| HRA-F | ATGCTTCTACTCCGTCTGCT | Upstream region of CAN1 |
| HRA-R | ATTTTGAAGCTATGAGCTCCAGCTTCGGCCACATTTATGACGATC | Upstreamregion of CAN1 |
| Cas9-F | ATAATGATCGTCATAAATGTGGCCGAAGCTGGAGCTCATAGCTTC | Cas9 |
| Cas9-R | CCTGCAGCGTACGAAGCTTCAGCTGCCGCAAATTAAAGCCTTCGAG | Cas9 |
| Leu2-F | GACGCTCGAAGGCTTTAATTTGCGGCAGCTGAAGCTTCGTACGC | LEU2 |
| Leu2-R | AGAAGAGTGGTTGCGAACAGAGTAAGCATAGGCCACTAGTGGATCTG | LEU2 |
| HRB-F | TATCAGATCCACTAGTGGCCTATGCTTACTCTGTTCGCAACCACT | Downstream region ofCAN1 |
| HRB-R | CTTGTCCCTTATTAGCCTTG | Downstream region ofCAN1 |
| 6005 | GATCATTTATCTTTCACTGCGGAGAAG | Backbone of pROS10 |
| YKL072W-F | TGCGCATGTTTCGGCGTTCGAAACTTCTCCGCAGTGAAAGATAAATGATCTCAA ATTCAAATTTATCACAGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAG TCCGTTATCAAC |
2 μm fragment |
| YKL061W-F | TGCGCATGTTTCGGCGTTCGAAACTTCTCCGCAGTGAAAGATAAATGATCTGAC ATGAAGTTAAATATGTGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAG TCCGTTATCAAC |
2 μm fragment |
| 11L-det-F | GACAAGACCACCGAGTCGAAT | Detection of deletant strain |
| 11L-det-R | TTTGTACGCTGGTGACATCG | Detection of deletant strain |
| 11L repair-F1 | GTGGATATACGTAATGCACATT | Donor DNA |
| 11L repair-R1 | TCGATCTATCGTTTTTGATAGCTTTTTCCCCCAGCAAATATATT | Donor DNA |
| 11L repair-F2 | AATATATTTGCTGGGGGAAAAAGCTATCAA AAACGATAGATCGA | Donor DNA |
| 11L repair-R2 | AGTTTTTTAATGAGTATTGAATCTG | Donor DNA |
| B29-F | CACCGAGTCGAATCCAAGTT | HR1 |
| B29-R | TAATCGATCTATCGTTTTTGATAGCTTTTTCCCCCAGCAAATATAT | HR1 |
| B30-F | AATAATATATTTGCTGGGGGAAAAAGCTATCAAAAACGATAGATCGAT | HR3 |
| B30-R | GCTTTTGTTCCCTTTAGTGAGGGTTCCAGAAATCTTGGCTGAACAC | HR3 |
| mazF-F | TTTGGTGTTCAGCCAAGATTTCTGGAACCCTCACTAAAGGGAACA | mazF |
| mazF-R | CCTGCAGCGTACGAAGCTTCAGCTGTGCTGGTCGCTATACTGCT | mazF |
| B31-F | ATCGACAGCAGTATAGCGACCAGCACAGCTGAAGCTTCGTACGC | LEU2 |
| B31-R | TCTGATGAGTGGCTTCCAGTTGCATGCATAGGCCACTAGTGGATCTG | LEU2 |
| B32-F | TATCAGATCCACTAGTGGCCTATGCATGCAACTGGAAGCCACTC | HR2 |
| B32-R | GTTGGAAATCCGACGTTATT | HR2 |
1.1.2 培养基和生长条件:YPD培养基(g/L):20 g葡萄糖,20 g蛋白胨,10 g酵母粉,1 L ddH2O。合成培养基SC (g/L):20.0 g葡萄糖,5.0 g硫酸铵,1.7 g无氨基酵母氮源,1.3 g氨基酸混合物,1 L ddH2O。碳源更换为同等比例半乳糖的合成培养基作为mazF负筛培养基。营养缺陷培养基则为添加了相应氨基酸缺陷的氨基酸混合物的合成培养基。LB培养基(g/L):10 g胰蛋白胨,5 g酵母粉,10 g氯化钠,1 L ddH2O。氨苄青霉素的使用浓度为100 μg/mL。固体培养基加入20 g/L琼脂粉。BY4741的培养条件为30 ℃,200 r/min;大肠杆菌培养条件为37 ℃,200 r/min。 1.1.3 主要试剂:SORB:0.1 mol/L醋酸锂,0.01 mol/L Tris-HCl (pH 8,用1 mol/L储液稀释),0.001 mol/L EDTA/NaOH (pH 8,用0.5 mol/L储液稀释),1 mol/L山梨醇,调节pH=8,灭菌后室温保存。40% PEG:0.1 mol/L醋酸锂,0.01 mol/L Tris-HCl (pH 8,用1 mol/L储液稀释),0.001 mol/L EDTA/NaOH (pH 8,用0.5 mol/L储液稀释),40% PEG3350,过滤除菌,4 ℃保存。实验所用氨基酸、抗生素购于鼎国科技生物有限公司;质粒快速小提试剂盒购于天根生物科技公司;PCR试剂,DNA marker购于宝生物工程有限公司;大肠杆菌感受态DH5α购于康为世纪生物科技有限公司。 1.2 利用CRISPR-Cas9系统进行基因组大片段删减
CRISPR-Cas9系统主要通过介导染色体双链断裂(DSB),进而以修复片段为模板进行DNA修复,从而达到大片段删减的目的,工作原理简图如图 1所示。为了提高大片段删减的成功率,本文在目的片段的两端选取了2个靶点,如图 1中箭头所指。修复片段如图所示由目的基因上下游片段融合而成,通过共转化带有2个靶序列的gRNA表达质粒与修复片段发生染色体双链断裂与定向修复。最终通过菌落PCR与测序确认目的大片段的删除。
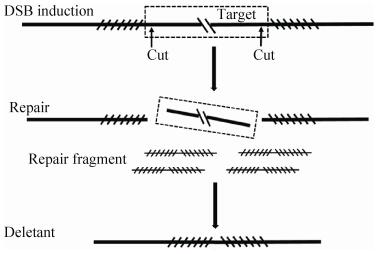
|
| 图 1 CRISPR-Cas9系统删减方法示意图 Figure 1 Schematic representation of the CRISPR-Cas9 system deletion method. |
1.2.1 Cas9蛋白表达菌株的构建:通过酵母基因组数据库(Saccharomyces Genome Database,SGD)查询基因can1序列,设计引物HRA-F/R、HRB-F/R,以酿酒酵母基因组为模板分别扩增can1基因的上下游片段作为同源重组的同源臂。以质粒pUG73为模板扩增LEU2标记,以菌株IMX581基因组为模板扩增cas9基因。将含有搭叠序列的片段共转化导入细胞,利用酿酒酵母的同源重组系统获得cas9与LEU2整合到酿酒酵母染色体上can1基因位置的Cas9蛋白表达菌株。 1.2.2 gRNA表达质粒的构建[8]:以质粒pROS10作为gRNA表达质粒,可搭载2个靶序列gRNA。首先PCR扩增得到pROS10质粒骨架以及带有2个靶点gRNA序列的2 μm片段,将纯化回收的2个片段转化至酿酒酵母BY4741,利用质粒pROS10所带的URA3筛选标记进行筛选。挑选单菌落接种至液体培养基,提取质粒回转至大肠杆菌DH5α扩增质粒,进行测序比对后,选取带有两个正确gRNA靶序列的质粒,并保存菌种。 1.3 mazF作为负筛标记的大片段删减方法[14-15]
利用mazF作为负筛标记的大片段删减方法主要有两步,如图 2所示,首先利用同源重组将同源臂、LEU2标记以及mazF表达盒整合到目的大片段的上游,得到中间态菌株。第二步,通过诱导表达毒力基因mazF发生第2次同源重组,将目的大片段以及筛选标记弹出,得到大片段无痕删减的最终缺失态菌株。
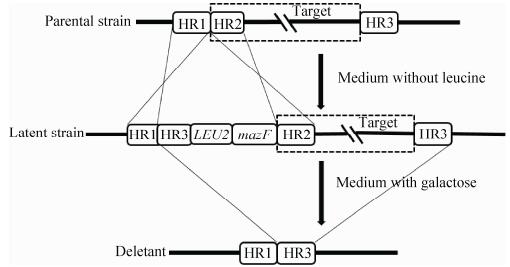
|
| 图 2 mazF介导的删减方法示意图 Figure 2 Schematic representation of the mazF-mediated deletion method. |
1.4 酿酒酵母感受态的制备及转化[16]
制备酿酒酵母感受态细胞:1.5 mL离心管室温收集酵母菌体,加入1 mL无菌水漂洗,再加入1 mL SORB进行漂洗。最后用50 μL SORB悬浮,加入4 μL鲑精DNA后用于转化或–80 ℃保存。
酿酒酵母转化是基于LiOAc法。取80 μL酵母感受态细胞,加入1/5体积质粒DNA或PCR片段产物充分混匀。加入6倍体积40% PEG,混匀后室温放置30 min。加入1/9体积DMSO混匀后42 ℃热击15 min。离心去上清后加入100 μL无菌水涂布平板。
1.5 基因组大片段删减菌株的表型检测为了检测大片段缺失菌株的生长表型,本文测定了出发菌株及大片段删减菌株的生长曲线以及在不同压力条件下菌株的生长情况。菌株起始OD调为0.1,接菌至含20 mL YPD培养基摇瓶中,间隔8 h利用分光光度计测定OD600吸光值。为了检测大片段缺失菌株的抗逆性有无变化,将10倍梯度稀释的菌液点至YPD平板以及分别添加了8%乙醇、1.5 mol/L NaCl、1 mol/L山梨醇的平板上。为了检测菌株的耐热性,将菌液于50 ℃水浴30 min后稀释点板。通过添加0.1 mol/L Tris-HCl缓冲液配置pH 8.5的碱性平板,检测耐碱性。
2 结果和分析 2.1 Cas9蛋白表达菌株的构建为了避免每次删减时都转化cas9基因表达质粒,本实验首先将cas9基因整合到酿酒酵母BY4741染色体上can1基因座,构建了1株Cas9蛋白表达菌株,命名为41-cas9菌株。PCR扩增cas9基因、同源臂HRA、HRB以及LEU2筛选标记。共转化4个片段,通过菌落PCR[17]初步检测整合成功的转化子。转接初步验证正确的转化子,转化靶点为ade2基因的质粒pROS10,将转化所得转化子点接到YPD平板,如图 3所示,可以观察到有变红的菌落出现,证明cas9基因正确整合到染色体并表达出具有生物活性的Cas9蛋白,在ade2基因位置进行切割导致其失活。

|
| 图 3 ade2基因突变菌落表型 Figure 3 Phenotype of colonies with ade2 gene mutated. |
2.2 构建gRNA表达质粒进行酿酒酵母大片段删减
本文选取质粒pROS10作为gRNA表达质粒,可搭载2个靶序列gRNA。以大片段YKL072W-YKL061W作为删减对象,该片段共包括14个ORF,全长约为26.5 kb,如表 3所示。分别选取大片段两端基因STB6和BLI1作为Cas9切割位点。PCR克隆pROS10质粒骨架和两侧分别带有含STB6和BLI1靶序列gRNA的2 μm片段后转化至酿酒酵母进行组装,提取质粒回转大肠杆菌扩增,经测序正确的质粒命名为pROS10-11L。供体DNA由删减片段上下游片段融合而成,大小为500 bp左右。将质粒pROS10-11L与修复片段一起转化至菌株41-cas9,涂布SC-ura-leu平板。通过6次平行实验,转化子个数平均为5个(表 4),以引物对11L-det-F/R进行菌落PCR检测,条带大小为1200 bp,符合预期,如图 4所示。测序验证正确敲除大片段。验证正确的YKL072W-YKL061W片段缺失菌命名为W4。未加修复片段的对照平板平均得到2个转化子,均为假阳性。转化子的数目偏少可能是由于同时进行2个位点的双链断裂而对菌体有损伤。
| Systematic name | Standard name | Length/ bp |
| YKL061W | BLI1 | 342 |
| YKL062W | MSN4 | 1893 |
| YKL063C | 504 | |
| YKL064W | MNR2 | 2910 |
| YKL065C | YET1 | 621 |
| YKL065C-A | 222 | |
| YKL066W | 444 | |
| YKL067W | YNK1 | 462 |
| YKL068W | NUP100 | 2880 |
| YKL068W-A | 237 | |
| YKL069W | 543 | |
| YKL070W | 510 | |
| YKL071W | 771 | |
| YKL072W | STB6 | 2301 |
| Methods | Clone No. a | Clones with expected deletion/% |
| CRISPR-Cas9 | 5 | 100 (30/30) |
| mazF | 100 | 93 (28/30) |
| aValues represent results of six independent operations. | ||

|
| 图 4 大片段删除PCR检测电泳图 Figure 4 Identify of large deletion. M: DL2000 marker; lane 1–8: PCR amplication with 11L-det-F/R. |
2.3 mazF作为负筛标记的大片段删减
该方法在Latour法的基础上将负筛标记URA3更换为mazF标记。通过半乳糖诱导mazF表达进行第2次同源重组,得到100个转化子(表 4)。随机选取30个转化子同样进行菌落PCR,正确率达93%。检测结果如图 5。

|
| 图 5 大片段删除PCR检测电泳图 Figure 5 Identify of large deletion. M: DL2000 marker; lane 1–9: PCR amplication with 11L-det-F/R; lane 10: wild type BY4741. |
2.4 大片段删减菌株的表型检测
出发菌株及大片段YKL072W-YKL061W缺失菌W4生长曲线如图 6所示。可见缺失菌株W4生长并未受到影响,与出发菌株几乎没有区别。抗逆性点板实验结果如图 7所示,在YPD、8%乙醇、1.5 mol/L NaCl、1.0 mol/L山梨醇、pH 8.5平板以及热处理后,W4菌株生长均未受影响,说明大片段YKL072W-YKL061W的删减对菌株耐热性、耐碱性、乙醇耐受性以及高盐、高渗透压条件下的生长无明显影响。
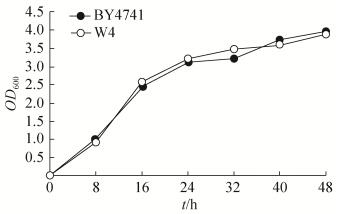
|
| 图 6 基因组大片段缺失菌株生长曲线 Figure 6 Growth curve of the strain with large deletion. |

|
| 图 7 大片段缺失菌株的表性检测 Figure 7 Phenotypes of the deletion mutants. |
3 讨论
CRISPR-Cas系统是一项新的用于定向基因编辑的重要技术,通过带有特异性靶序列gRNA引导Cas9核酸酶,在目的序列进行切割产生双链断裂,通过供体DNA与基因组DNA的同源重组进而实现对基因组的定向改造。本实验选取了26.5 kb的染色体大片段YKL072W-YKL061W,在大片段区域的上下游端部基因位置选取了2个靶序列,构建双gRNA表达质粒,与修复DNA一起转化至已构建好的Cas9蛋白表达菌株41-cas9。平均得到5个转化子,经检测全部为正确删减了大片段的缺失菌株,正确率100%。
mazF介导的大片段删减方法首先转化3个同源臂及正负筛选标记构建中间态菌株,然后利用诱导表达mazF毒力基因进行大片段的删减以及筛选标记的回收,得到无碱基残留的大片段缺失菌株。本文中在大片段YKL072W-YKL061W的删减中得到约100个转化子,正确率高达93%。
本文也对大片段YKL072W-YKL061W删减菌株W4进行了初步的表性检测,生长曲线表明缺失菌株的生长状态未受到影响;在8%乙醇、1.5 mol/L NaCl、1.0 mol/L山梨醇、pH 8.5平板以及热处理条件下W4生长状况也与野生菌株BY4741无异。
CRISPR-Cas系统以其操作的简便性在大片段删减应用中的优势明显,尤其体现在大规模基因删减中,仅需改变20 bp的gRNA序列即可,在实验设计及操作上都极为简便。且在添加了修复模板DNA同源重组修复后正确率几乎为100%,为合成生物学中最小基因组细胞的构建创造了可能。mazF介导的大片段删减方法需要克隆多个片段,且需要诱导表达mazF,在更换目的片段后需要重新设计大批引物,相比之下操作相对繁复。但是根据文献报道,CRISPR-Cas系统存在脱靶现象,导致得到非预期的突变。也就是说,CRISPR-Cas系统在针对不同的删除目标时作用效果并不稳定。而mazF法的优势在于不需要PAM序列,相对稳定,具有普适性。
总之,本文中两种方法均可以高效删减酿酒酵母染色体大片段。CRISPR-Cas系统适用于大批量删减基因,操作简便省时;mazF介导的大片段删减法适用于无合适gRNA序列等基因。
| [1] | Runguphan W, Keasling JD. Metabolic engineering of Saccharomyces cerevisiae for production of fatty acid-derived biofuels and chemicals. Metabolic Engineering, 2014, 21: 103-113. DOI:10.1016/j.ymben.2013.07.003 |
| [2] | Fujio T. Minimum genome factory:innovation in bioprocesses through genome science. Biotechnology and Applied Biochemistry, 2007, 46(3): 145-146. DOI:10.1042/BA20060117 |
| [3] | Sugiyama M, Nakazawa T, Murakami K, Sumiya T, Nakamura A, Kaneko Y, Nishizawa M, Harashima S. PCR-mediated one-step deletion of targeted chromosomal regions in haploid Saccharomyces cerevisiae. Applied Microbiology and Biotechnology, 2008, 80(3): 545-553. DOI:10.1007/s00253-008-1609-9 |
| [4] | Gueldener U, Heinisch J, Koehler GJ, Voss D, Hegemann JH. A second set of loxP marker cassettes for Cre-mediated multiple gene knockouts in budding yeast. Nucleic Acids Research, 2002, 30(6): e23. DOI:10.1093/nar/30.6.e23 |
| [5] | Hirashima K, Iwaki T, Takegawa K, Giga-Hama Y, Tohda H. A simple and effective chromosome modification method for large-scale deletion of genome sequences and identification of essential genes in fission yeast. Nucleic Acids Research, 2006, 34(2): e11. DOI:10.1093/nar/gnj011 |
| [6] | Jiang WY, Bikard D, Cox D, Zhang F, Marraffini LA. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508 |
| [7] | DiCarlo JE, Norville JE, Mali P, Rios X, Aach J, Church GM. Genome engineering in saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research, 2013, 41(7): 4336-4343. DOI:10.1093/nar/gkt135 |
| [8] | Mans R, van Rossum HM, Wijsman M, Backx A, Kuijpers NGA, van den Broek M, Daran-Lapujade P, Pronk JT, van Maris AJ, Daran JMG. Crispr/Cas9:a molecular swiss army knife for simultaneous introduction of multiple genetic modifications in Saccharomyces cerevisiae. FEMS Yeast Research, 2015, 15(2): fov004. |
| [9] | Hao HH, Wang XF, Jia HY, Yu M, Zhang XY, Tang H, Zhang LP. Large fragment deletion using a CRISPR/Cas9 system in Saccharomyces cerevisiae. Analytical Biochemistry, 2016, 509: 118-123. DOI:10.1016/j.ab.2016.07.008 |
| [10] | Zhou H, Liu B, Weeks DP, Spalding MH, Yang B. Large chromosomal deletions and heritable small genetic changes induced by CRISPR/Cas9 in rice. Nucleic Acids Research, 2014, 42(17): 10903-10914. DOI:10.1093/nar/gku806 |
| [11] | Zhang LQ, Jia RR, Palange NJ, Satheka AC, Togo J, An Y, Humphrey M, Ban LY, Ji Y, Jin HH, Feng XC, Zheng YW. Large genomic fragment deletions and insertions in mouse using CRISPR/Cas9. PLoS One, 2015, 10(3): e120396. |
| [12] |
Fu Q, Sun JH, Yan YX. The functional aspects of bacterial CRISPR-Cas systems and interactions between phages and its bacterial hosts——a review. Acta Microbiologica Sinica, 2015, 55(3): 251-257.
(in Chinese) 傅强, 孙建和, 严亚贤. 细菌CRISPR-Cas系统功能及其与噬菌体相互作用. 微生物学报, 2015, 55(3): 251-257. |
| [13] | Sander JD, Joung JK. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology, 2014, 32(4): 347-355. DOI:10.1038/nbt.2842 |
| [14] | Liu QL, Liu HJ, Yang YY, Zhang XM, Bai YL, Qiao MQ, Xu HJ. Scarless gene deletion using mazF as a new counter-selection marker and an improved deletion cassette assembly method in Saccharomyces cerevisiae. The Journal of General and Applied Microbiology, 2014, 60(2): 89-93. DOI:10.2323/jgam.60.89 |
| [15] | Liu QL, Wu YZ, Yang P, Zhang XM, Bai YL, Xu HJ, Qiao MQ. MazF-mediated deletion system for large-scale genome engineering in Saccharomyces cerevisiae. Research in Microbiology, 2014, 165(10): 836-840. DOI:10.1016/j.resmic.2014.10.005 |
| [16] | Knop M, Siegers K, Pereira G, Zachariae W, Winsor B, Nasmyth K, Schiebel E. Epitope tagging of yeast genes using a PCR-based strategy:more tags and improved practical routines. Yeast, 1999, 15(10B): 963-972. DOI:10.1002/(SICI)1097-0061(199907)15:10B<>1.0.CO;2-M |
| [17] | Wang H, Kohalmi SE, Cutler AJ. An improved method for polymerase chain reaction using whole yeast cells. Analytical Biochemistry, 1996, 237(1): 145-146. DOI:10.1006/abio.1996.0213 |
 2017, Vol. 57
2017, Vol. 57




