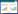中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 王晓庆, 王晓政, 陈亮宇, 赵心清, 黄婷婷, 林双君
- Xiaoqing Wang, Xiaozheng Wang, Liangyu Chen, Xinqing Zhao, Tingting Huang, Shuangjun Lin
- 基因组信息指导的星海链霉菌中2-甲硫基-N6-(4-羟基异戊烯基)-腺苷的分离鉴定
- Genome guided identification of 2-methylthio-N6-(4-hydroxy-isopentenyl)-adenosine from Streptomyces xinghaiensis NRRL B24674T
- 微生物学报, 2016, 56(5): 778-786
- Acta Microbiologica Sinica, 2016, 56(5): 778-786
-
文章历史
- 收稿日期: 2015-07-07
- 修回日期: 2015-08-28
- 网络出版日期: 2015-09-29
2. 大连理工大学生命科学技术学院, 辽宁 大连 116024
2. School of Life Science and Biotechnology, Dalian University of Technology, Dalian 116024, Liaoning Province, China
tRNA的转录后修饰几乎存在于包括古生菌、细菌和真核生物在内的所有生物当中[1]。有趣的是,生物体内存在大量用于tRNA核苷酸的修饰酶[2],从而形成多样的tRNA结构。目前为止,从tRNA中鉴定了100多个被修饰的核苷酸结构(http://medstat.med.utah.edu/RNAmods),例如4-硫尿核苷、5-甲基胺甲基-2-硫尿核苷、2-硫代胞苷等[3]。目前研究较多的是位于tRNA反密码子环附近的修饰,这种修饰大多数在其34位(摇摆位点)或37位(反密码子的3'端附近)进行甲基化修饰或者甲硫基化修饰[4],例如2-甲硫基-N6-异戊烯基-腺苷(ms2i6A)修饰和2-甲硫基-N6-(4-羟基异戊烯基)-腺苷(ms2io6A)修饰。
tRNA核苷酸修饰的生物学作用目前还不是很清晰。最初认为这些修饰在维持tRNA独特的二级和三级结构稳定性上发挥作用。在一些情况下,tRNA中被修饰核苷酸的出现能够影响中心代谢,如硫胺素的生物合成以及细菌的毒性作用等[4]。深入研究表明,在tRNA反密码子环区域和摆动位点的特殊修饰能够提高翻译的保真度和翻译效率,同时降低翻译中移码突变的概率,影响细胞生长和细胞对胁迫环境的应激反应[5, 6]。
研究发现,在大肠杆菌tRNA的37位碱基的修饰为2-甲硫基-N6-异戊烯基-腺苷[7];在鼠伤寒沙门氏菌中,存在以2-甲硫基-N6-(4-羟基异戊烯基)-腺苷修饰的tRNA[7];在红球菌中存在2-甲硫基-异戊烯基腺嘌呤、2-甲硫基-异戊烯基腺苷、2-甲硫基-N6-(4-羟基异戊烯基)-腺苷等[8];但在链霉菌中目前还没有相关的报道。然而,一些放线菌,尤以链霉菌为主,能在次级代谢过程中产生一些重要的核苷类次级代谢物,它们都是由核苷或核苷酸衍生而成的天然产物,其结构多样并且很多具有广谱的抗菌活性[9, 10]。因此,我们推测在链霉菌中可能也存在类似的核苷修饰方式。
ms2io6A在鼠伤寒沙门氏菌中的生物合成途径已经有报道,其推测的合成途径如图 1,MiaA、B和E是参与其中的相关蛋白[11]。首先二甲基烯丙基二磷酸-tRNA转移酶(MiaA)催化DMAPP[二甲基烯丙基二磷酸(Δ2-异戊烯焦磷酸)]的二甲基烯丙基基团转移到tRNA37位腺苷(A37)的氨基N6上形成N6-异戊烯基腺苷(i6A37)[12];然后甲硫基-N6-异戊烯基腺苷合成酶(MiaB)利用S-腺苷甲硫氨酸(SAM)、铁硫蛋白催化i6A37甲硫基化,形成2-甲硫基-N6-异戊烯基腺苷(ms2i6 A37)[13, 14];最后2-甲硫基-N6-异戊烯基腺苷-tRNA单加氧酶(MiaE)催化ms2i6A37氧化形成2-甲硫基-N6-(4-羟基异戊烯基)腺苷(ms2io6A)[15]。

|
| 图 1. ms2io6A可能的修饰途径 Figure 1. The hypothetic modification pathway for ms2io6A. |
本研究中使用的菌种星海链霉菌S.xinghaiensis NRRL B24674T是从大连星海湾分离鉴定的一株海洋来源链霉菌。经初步检测,该菌发酵液具有很好的广谱抗菌活性,不仅对鲍曼不动杆菌、铜绿假单胞菌、大肠杆菌等革兰氏阴性菌具有很好的抑制作用,而且对葡萄球菌、枯草芽胞杆菌等革兰氏阳性菌均具有很好的抑制作用[16]。星海链霉菌S.xinghaiensis NRRL B24674T基因组测序已经完成[17],本文利用该菌的基因组序列信息,通过基因组挖掘的方式,寻找上述甲硫基或异戊烯基修饰的核苷的相关合成基因,然后通过一系列分离纯化手段对该菌的发酵粗提物进行分析,以探究该菌次级代谢产物中是否存在2-甲硫基或N6-异戊烯基修饰的核苷,或者以这些结构单元为母体形成的核苷类次级代谢产物。
1 材料和方法 1.1 菌株与培养基星海链霉菌S.xinghaiensis NRRL B24674T分离自大连星海湾。固体平板培养基(g/L):玉米淀粉30.0,黄豆饼粉7.0,KNO3 1.5,K2HPO4 0.5,琼脂20,蒸馏水1 L,pH 7.5。种子培养基(g/L):胰蛋白胨17.0,大豆蛋白胨3.0,葡萄糖2.5,K2HPO4 2.5,蒸馏水1 L,pH 7.5。发酵培养基(g/L):可溶性淀粉20.0,黄豆饼粉25.0,(NH4)2SO4 2.0,NaCl 2.0,K2HPO4 0.5,CaCO3 5.0,蒸馏水1 L,pH 7.2。
1.2 主要试剂与仪器试剂:石油醚、氯仿、二氯甲烷、乙酸乙酯、甲醇、乙酸、氨水,购自上海凌峰试剂公司;氘代甲醇,购自美国Cambridge Isotope Laboratory公司;色谱级乙腈(Acetonitrile,ACN),购自Sigma-Aldrich公司;HP20大孔树脂,购自日本三菱株式会社;正相柱层析硅胶,购自上海盛亚化工有限公司;C18反相硅胶柱层析硅胶(50 μm),购自日本YMC公司;Sephedex TM LH-20,购自美国GE医疗。
仪器:旋转蒸发仪,购自荷兰Buchi公司;Innova 44R摇床,购自美国New Brunswick Scientific公司;恒温生化培养箱,购自韶关市泰宏医疗器械有限公司;高效液相色谱仪(High Performance Liquid Chromatography,HPLC),液-质联用仪(Liquid Chromatography-Mass Spectrometer,LC-MS),购自安捷伦科技有限公司;600 M核磁共振仪,购自德国Bruker公司。
1.3 序列分析利用BioEdit 7.1.11建立星海链霉菌基因组序列(GenBank登录号:AFRPxb20150299000)[17]的本地数据库用于BLAST比对。以Salmonella enterica中MiaA(WP_001000734),MiaB(WP_001519200)和MiaE(WP_000218459)的氨基酸序列在该基因组数据库进行比对,查找同源蛋白。利用PSI-BLAST在NCBI的GenBank中进行同源蛋白查找。氨基酸序列比对用BioEdit进行,系统进化树构建用MEGA 5.0完成,系统进化树中所用MiaA同源蛋白来自于GenBank。
1.4 菌株的培养及发酵将菌种划线接种于固体平板,30 ℃恒温培养箱培养6 d,用接种环刮取1 cm2的孢子,接种于50 mL种子培养基,30 ℃、200 r/min振荡培养48 h。取种子液,按10%接种量接种到500 mL发酵培养基,30 ℃、200 r/min振荡培养96 h,加入12%(质量比)的灭菌的HP20大孔树脂,继续振荡培养72 h。
1.5 次级代谢产物的提取对星海链霉菌进行大量发酵,共85 L。将发酵所得混合物取出后,6000 r/min离心30 min,取得上层的大孔树脂。随后用1.5倍发酵液体积的乙酸乙酯对大孔树脂进行反复萃取3次。再将萃取后的乙酸乙酯于30 ℃减压蒸馏,去除有机溶剂,获得发酵粗提物。
1.6 次级代谢产物的分离纯化使用正向硅胶色谱柱对发酵粗提物进行分离,分离条件为石油醚,石油醚:二氯甲烷=3:1,二氯甲烷,二氯甲烷:甲醇=20:1、5:1和1:1,甲醇。收集洗脱液,利用高效液相色谱仪对其进行分析,将样品合并为FR1-FR6共6个组分。HPLC中以水和乙腈作为流动相,HPLC分离柱为安捷伦XDB C18柱(5 µm,4.6 mm×250 mm),梯度洗脱条件为:0-15 min,10%-100% ACN(乙腈);15-25 min,100% ACN;25-27 min,100%-10% ACN;27-33 min,10% ACN;流速为0.6 mL/min,检测波长设定为210 nm,在没有特别说明情况下,HPLC的检测波长均为210 nm。
用长度与直径比较大的反相硅胶柱色谱柱对FR4进行粗分,用甲醇和水的混合液作为流动相进行梯度洗脱,甲醇的浓度依次为30%、50%、75%、100%。收集洗脱液,利用HPLC对样品进行分析,合并为FR4-7。然后,用长度与直径比较小的反相硅胶柱色谱柱对FR4-7进行细分,用甲醇和水的混合液作为流动相进行梯度洗脱,甲醇的浓度依次为40%、50%、100%。收集洗脱液,利用HPLC对样品进行分析,合并为FR4-7-8。对组分FR4-7-8用Sephedex LH-20进行纯化,甲醇作为洗脱剂,HPLC分析洗脱液,合并样品得到化合物1,纯度为97%。
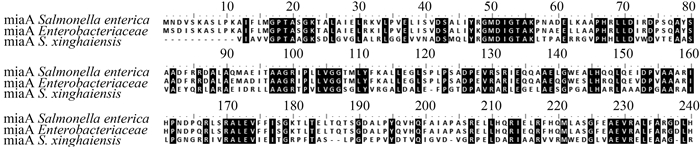
2 结果和分析 2.1 星海链霉菌基因组中MiaA和MiaB的同源蛋白我们以对Salmonella enterica中tRNA的37位腺苷进行2-甲硫基-N6-(4-羟基异戊烯基)(ms2io6A)修饰的关键蛋白MiaA(WP_001000734),MiaB(WP_001519200)和MiaE(WP_000218459)为出发蛋白,通过同源比对,在星海链霉菌基因组中找到MiaA和MiaB的同源蛋白,与Salmonella enterica中相应蛋白的一致性和相似性分别为39%/56%和40%/59%(图 2,图 3)。随后,我们通过同源比对搜索从NCBI GenBank数据库中获得不同物种来源的MiaA同源蛋白,以邻近法构建系统进化树,结果表明MiaA蛋白在动物、植物、古菌和原核生物中均广泛存在,其中链霉菌来源的MiaA同源蛋白聚为同一分支(图 4)。在基因组中MiaB蛋白一般与MiaA偶联在一起,系统发育分布也具有类似性。这些生物信息学分析结果暗示这株菌中可能存在相似的核酸或者腺苷的修饰途径并产生此类修饰的转运核酸或者腺苷[18]。因此我们对星海链霉菌的发酵粗提物进行分离纯化。
|
| 图 2. 星海链霉菌中的MiaA同源蛋白及其序列比对 Figure 2. MiaA homolog from S. xinghaiensis and the sequence alignment. |

|
| 图 3. 星海链霉菌中的MiaB同源蛋白及其序列比对 Figure 3. MiaB homolog from S. xinghaiensis and the sequence alignment. |

|
| 图 4. 邻近法构建基于MiaA同源蛋白的系统发育树 Figure 4. Phylogenetic tree of MiaA homologues using Neighbour-Joining method. MiaA from S. xinghaiensis highlights in bold. |
化合物1为淡黄色粉末,易溶于甲醇和二甲基亚砜,微溶于氯仿。紫外吸收如图 5所示,其中λmax:244 nm,284 nm。对化合物进行液-质(LC-MS)分析,正离子模式下,其准分子离子峰为398.1464 [M+H]+,而其理论分子量为398.1493,因此确定它的分子式为C16H23N5O5S。将化合物1溶解在0.4 mL的氘代甲醇中,进行核磁共振(Nuclear Magnetic Resonance,NMR)分析,以四甲基硅烷(TMS)作为内标(表 1),1H NMR(CD3OD)显示该化合物含有一个芳香质子信号δH 8.12(s,H-9,1H),结合化合物的分子式中含有多个氮原子,推测化合物存在一个腺嘌呤单元,但相对于腺嘌呤缺少了一个H-2,由此可以确认该化合物存在一个2-位取代的腺嘌呤单元,这与该化合物的紫外吸收相一致。1H NMR还显示该化合物还有6个与杂原子相连的氢【δH 5.91(d,J=5.94 Hz,H-1′,1H),4.72(dd,J=5.52,5.47 Hz,H-2′,1H),4.31(dd,J=5.92,5.04 Hz,H-3′,1H),4.11(dd,J=5.94,2.94 Hz,H-4′,1H),3.85(dd,J=12.36,2.76 Hz,H-5′a,1H),3.72(dd,J=12.36,2.76 Hz,H-5′b,1H)】,根据1H-1H COSY相关(H-1′/H-2′/H-3′/H-4′/H-5′a/H-5′b)推测该化合物存在糖单元。该糖的端基氢偶合常数5.94 Hz,可知该结构中所含糖单元为β构型。结合HSQC谱,13C NMR显示该化合物有5个连氧碳【δC 89.8(C-1′),74.2(C-2′),71.2(C-3′),86.5(C-4′),62.3(C-5′)】,由此确认该化合物中β核糖单元。结合以上信息,该化合物存在一个取代腺嘌呤核糖核苷,即片段1(Fragment 1)。另外1H NMR还显示该化合物存在一个与N相连的亚甲基【δH 4.28(brs,H-1'',2H)】,以及一个双键质子信号【δH 5.48(brs,H-2'',1H)】,1H-1H COSY相关显示这组氢存在相关,表明该化合物存在片段2(Fragment 2)。1H NMR还显示该化合物存在一个与季碳相连的甲基信号【δH 1.82(s,H-5'',3H)】,一个与甲硫基信号【δH 2.55(s,H-10,3H),一个与氧相连的亚甲基信号【δH 4.20(s,H-4'',2H)】,这些数据都与13C NMR相一致【δC 20.6(C-15),13.6(C-10),60.6(C-14)】。根据HMBC,可知甲硫基位于C-4位。查阅相关文献,结合HMBC,将此化合物确定为2-甲硫基-N6-(4-羟基异戊烯基)-腺苷(ms2io6A)(图 6)。

|
| 图 5. 化合物1在HPLC上的保留时间和紫外吸收图谱 Figure 5. The retention time of 1 on the HPLC chromatogram and its UV-vis spectrum. |
| No. | δH(multi, J/Hz)a | δC(DEPT)b | 1H-1H COSY | HMBC |
| 2 | 166.3s | |||
| 4 | 148.9s | |||
| 5 | 117.2s | |||
| 6 | 168.3s | |||
| 8 | 8.10 (s) | 139.4d | 2, 7 | |
| 1′ | 5.91 (d, 5.94) | 89.8d | 2′ | 3′ |
| 2′ | 4.72 (dd, 5.52, 5.47) | 74.2d | 1′, 3′ | |
| 3′ | 4.31 (dd, 4.92, 5.04) | 71.2d | 2′, 4′ | 5a′ |
| 4′ | 4.11 (dd, 2.94, 5.94) | 86.5d | 3′, 5a′, 5b′ | 2′ |
| 5′ | 3.85 (dd, 2.76, 12.36) 3.72 (dd, 2.76, 12.36) | 62.3t | 4′, 5b′ 4′, 5a′ | |
| 1′′ | 4.28 (brs) | 65.1t | 2′′ | 3′′ |
| 2′′ | 5.48 (brs) | 123.3d | 1′′ | 4′′, 5′′ |
| 3′′ | 139.4s | |||
| 4′′ | 4.20 (s) | 60.6t | 2′′ | |
| 5′′ | 1.82 (s) | 20.6q | 4′′, 2′′ | |
| SCH3 | 2.55 (s) | 13.6q | 4 |

|
| 图 6. 化合物1的组成片段的化学结构 Figure 6. Chemical structures of compound 1 and its fragments. |
tRNA中腺苷修饰广泛存在于所有生物中,并在细胞生长、分化、转录、翻译等过程中发挥着重要作用,例如植物激素—细胞分裂素就是一类被修饰的腺苷衍生物。细胞分裂素能够促进植物的生长,调节植物的顶端优势、叶的衰老以及根的发育等生长过程[13]。它一般是在腺苷嘌呤环的N6位置上的类异戊二烯或者芳香族侧链修饰,或者以自由的碱基、核苷或核苷酸存在,或者在嘌呤环的N7和N9,或者在羟基化侧链上有糖基修饰[8]。植物中的ms2io6A就属于细胞分裂素的一种。
此外,ms2io6A修饰的核苷酸还有望成为癌症的标志物。核苷酸的修饰是复杂有序多酶催化的RNA成熟过程的一部分,但修饰的核苷酸在RNA代谢过程中由于缺乏相应的磷酸化酶而作为终产物随着尿液排出体外,因此尿液中的RNA水平可用于鉴别癌症患者和健康人的标准。德国科学家Bernd Kammerer分析了1-4期的乳腺癌患者的尿液,其中就发现了ms2io6A的存在[19]。
ms2io6A修饰也存在于克雷伯氏菌、铜绿假单胞菌、粘质沙雷氏菌、费氏柠檬酸杆菌和一些植物病原菌,如土壤农杆菌、根瘤菌、棒状杆菌中[7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20]。在沙门氏菌中的研究表明,ms2io6A修饰的tRNA提高了对酪氨酸、色氨酸、半胱氨酸和丝氨酸的选择,减少了对苯丙氨酸的选择[21]。tRNA37位碱基没有ms2io6A的修饰,将会导致菌体的翻译效率降低且移码突变的几率提高[22]。同时,沙门氏菌可以在琥珀酸、延胡索酸和苹果酸作为碳源的培养基上生长,也是依赖于其tRNA37位碱基的ms2io6A修饰[23]。
本文通过对星海链霉菌的基因组进行分析比对,找到了参与ms2io6A修饰的同源蛋白MiaA和MiaB,这两种蛋白与已经报道沙门氏菌中的相应蛋白具有较高的同源性,由此推测星海链霉菌中也可能存在类似的修饰方式。通过对星海链霉菌发酵产物的分离纯化,最终得到2-甲硫基-N6-(4-羟基异戊烯基)-腺苷。这是首次在链霉菌中分离发现2-甲硫基-N6-(4-羟基异戊烯基)-腺苷,通过核磁确定其结构,也是首次报道了该化合物的核磁共振波谱数据。传统的ms2io6A的分离是首先获得菌体RNA,然后将其降解所得[24],而在我们的研究中,ms2io6A却可以直接在发酵液中分离得到。因此推测,ms2io6A可能是在tRNA代谢中产生而被排出体外的,或者是该类酶具有一定的底物广谱性,从而催化腺苷发生了类似的修饰反应。
此外,在对NCBI GenBank数据库进行同源蛋白查找的过程中,我们发现MiaA和MiaB的同源蛋白广泛存在于动物、植物、古菌和原核生物中,也在链霉菌中普遍存在(图 4)。尽管在链霉菌中还没有对这类修饰的报道和功能研究,但其生物合成相关基因的普遍存在,以及本文在星海链霉菌中分离并确定了这类化合物的产生,说明这种修饰在链霉菌中具有普遍性,并且可能具有一定的生物学意义,值得开展深入的研究。
| [1] | Björk GR, Durand JMB, Hagervall TG, Leipuviene R, Lundgren HK, Nilsson K, Chen P, Qian Q, Urbonavicius J. Transfer RNA modification:influence on translational frameshifting and metabolism. FEBS Letters, 1999, 452(1/2):47-51. |
| [2] | Gustilo EM, Vendeix FAP, Agris PF. tRNA's modifications bring order to gene expression. Current Opinion in Microbiology, 2008, 11(2):134-140. |
| [3] | Lundgren HK, Björk GR. Structural alterations of the cysteine desulfurase IscS of Salmonella enterica serovar Typhimurium reveal substrate specificity of IscS in tRNA thiolation. Journal of Bacteriology, 2006, 188(8):3052-3062. |
| [4] | Persson BC. Modification of tRNA as a regulatory device. Molecular Microbiology, 1993, 8(6):1011-1016. |
| [5] | Gu C, Begley TJ, Dedon PC. tRNA modifications regulate translation during cellular stress. FEBS Letters, 2014, 588(23):4287-4296. |
| [6] | Endres L, Dedon PC, Begley TJ. Codon-biased translation can be regulated by wobble-base tRNA modification systems during cellular stress responses. RNA Biology, 2015, 12(6):603-614. |
| [7] | Persson BC, Bjork GR. Isolation of the gene (miaE) encoding the hydroxylase involved in the synthesis of 2-methylthio-cis-ribozeatin in tRNA of Salmonella typhimurium and characterization of mutants. Journal of Bacteriology, 1993, 175(24):7776-7785. |
| [8] | Tarkowski P, Václavíková K, Novák O, Pertry I, Hanuš J, Whenham R, Vereecke D, Šebela M, Strnad M. Analysis of 2-methylthio-derivatives of isoprenoid cytokinins by liquid chromatography-tandem mass spectrometry. Analytica Chimica Acta, 2010, 680(1/2):86-91. |
| [9] | Isono K. Nucleoside antibiotics:structure, biological activity, and biosynthesis. The Journal of Antibiotics, 1988, 41(12):1711-1739. |
| [10] | Niu GQ, Tan HR. Nucleoside antibiotics:biosynthesis, regulation, and biotechnology. Trends in Microbiology, 2015, 23(2):110-119. |
| [11] | Corder AL, Subedi BP, Zhang SA, Dark AM, Foss FW Jr, Pierce BS. Peroxide-shunt substrate-specificity for the Salmonella typhimurium O2-dependent tRNA modifying monooxygenase (MiaE). Biochemistry, 2013, 52(36):6182-6196. |
| [12] | Moore JA, Poulter CD. Escherichia coli dimethylallyl diphosphate:tRNA dimethylallyltransferase:a binding mechanism for recombinant enzyme. Biochemistry, 1997, 36(3):604-614. |
| [13] | Arragain S, Handelman SK, Forouhar F, Wei FY, Tomizawa K, Hunt JF, Douki T, Fontecave M, Mulliez E, Atta M. Identification of eukaryotic and prokaryotic methylthiotransferase for biosynthesis of 2-methylthio-N6-threonylcarbamoyladenosine in tRNA. Journal of Biological Chemistry, 2010, 285(37):28425-28433. |
| [14] | Pierrel F, Douki T, Fontecave M, Atta M. MiaB protein is a bifunctional radical-S-adenosylmethionine enzyme involved in thiolation and methylation of tRNA. Journal of Biological Chemistry, 2004, 279(46):47555-47563. |
| [15] | Mathevon C, Pierrel F, Oddou JL, Garcia-Serres R, Blonclin G, Latour JM, Ménage S, Gambarelli S, Fontecave M, Atta M. tRNA-modifying MiaE protein from Salmonella typhimurium is a nonheme diiron monooxygenase. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(33):13295-13300. |
| [16] | Zhao XQ, Li WJ, Jiao WC, Li Y, Yuan WJ, Zhang YQ, Klenk HP, Suh JW, Bai FW. Streptomyces xinghaiensis sp. nov., isolated from marine sediment. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(11):2870-2874. |
| [17] | Zhao XQ, Yang TH. Draft genome sequence of the marine sediment-derived actinomycete Streptomyces xinghaiensis NRRL B24674T. Journal of Bacteriology, 2011, 193(19):5543-5543. |
| [18] | Kaminska KH, Baraniak U, Boniecki M, Nowaczyk K, Czerwoniec A, Bujnicki JM. Structural bioinformatics analysis of enzymes involved in the biosynthesis pathway of the hypermodified nucleoside ms2io6A37 in tRNA. Proteins, 2008, 70(1):1-18. |
| [19] | Frickenschmidt A, Fröhlich H, Bullinger D, Zell A, Laufer S, Gleiter CH, Liebich H, Kammerer B. Metabonomics in cancer diagnosis:mass spectrometry-based profiling of urinary nucleosides from breast cancer patients. Biomarkers, 2008, 13(4):435-449. |
| [20] | Ajitkumar P, Cherayil JD. Presence of 2-methylthioribosyl-trans-zeatin in Azotobacter vinelandii tRNA. Journal of Bacteriology, 1985, 162(2):752-755. |
| [21] | Li JN, Esberg B, Curran JF, Björk GR. Three modified nucleosides present in the anticodon stem and loop influence the in vivo aa-tRNA selection in a tRNA-dependent manner. Journal of Molecular Biology, 1997, 271(2):209-221. |
| [22] | Qian Q, Björk GR. Structural alterations far from the anticodon of the tRNAGGGPro of Salmonella typhimurium induce +1 frameshifting at the peptidyl-site. Journal of Molecular Biology, 1997, 273(5):978-992. |
| [23] | Petersson L, Gräslund A, Ehrenberg A, Sjöberg BM, Reichard P. The iron center in ribonucleotide reductase from Escherichia coli. Journal of Biological Chemistry, 1980, 255(14):6706-6712. |
| [24] | Janzer JJ, Raney JP, McLennan BD. The transfer RNA of certain Enterobacteriacae contain 2-methylthiozeatin riboside (ms2io6A) an isopentenyl adenosine derivative. Nucleic Acids Research, 1982, 10(18):5663-5672. |
 2016, Vol. 56
2016, Vol. 56