中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 贾晓歌, 邓子新, 刘天罡
- Xiaoge Jia, Zixin Deng, Tiangang Liu
- 无细胞蛋白表达体系研究进展及在生物制药领域中的应用
- Progress of cell-free protein synthesis system and its applications in pharmaceutical engineering - A review
- 微生物学报, 2016, 56(3): 530-542
- Acta Microbiologica Sinica, 2016, 56(3): 530-542
-
文章历史
- 收稿日期:2015-10-27
- 修回日期:2015-12-21
- 网络出版日期:2016-01-05
无细胞蛋白表达体系(CFPS)利用细胞抽提物中的酶和蛋白质因子,以外源DNA或mRNA为模板,通过补充翻译所需的底物和能量物质,从而实现目标蛋白的体外合成。作为一种快速高效的蛋白质合成手段,CFPS一直被广泛应用于基础生物学领域的研究。早在1961年,Nirenberg和Matthaei就已经利用大肠杆菌的无细胞体系阐明了遗传密码子及其在蛋白质合成中的作用[1]。近年来随着分子生物学和蛋白质组学研究的不断深入以及传统的基于细胞的体内表达系统在生物制药领域中受到限制,如重组蛋白药物对细胞毒性大、易被体内蛋白酶降解、下游分离纯化成本高等,CFPS因可以克服上述缺点而再次受到关注[2]。与体内表达体系相比,CFPS不受胞内复杂环境的影响、可调控性强、对毒性蛋白的耐受力高、产物分离成本低,可以为解决生物制药领域发展的关键问题提供新的解决方案[3, 4]。早期的CFPS存在许多缺点,如反应持续时间短、生产效率低、能量再生系统成本高昂、蛋白易出现错误折叠、反应体积小等,严重阻碍了CFPS在工业上大规模生产的推广与应用[5, 6]。为了突破这些瓶颈限制,在过去的十几年中科学家们不断地对CFPS进行优化,通过简化制备工艺、开发价格低廉的能量再生系统、稳定底物供应、促进蛋白正确折叠、扩大反应体系体积等方式,成功地研发出反应持续时间长、生产效率高、成本低廉的表达体系(图 1),为其在高通量蛋白表达和工业化大规模生产方面奠定了坚实的基础。

|
| 图 1. 利用CFPS工业化生产重组蛋白药物示意图 Figure 1. Schematic illustration of industrial production of protein therapeutics using CFPS systems. |
自从1982年世界上第一个利用重组DNA技术生产的重组蛋白药物胰岛素问世以来,在短短的几十年时间里,重组蛋白药物因其安全性高,来源广泛以及便于对蛋白进行修饰等优点在生物制药领域迅猛发展,并在全球药物市场上占据非常重要地位[7, 8, 9]。大肠杆菌(Escherichia coli,E. coli),酿酒酵母(Saccharomyces cerevisiae) 和中国仓鼠卵巢细胞(Chinese hamster ovary,CHO)是表达重组蛋白药物最常用的系统[10],然而这些体内表达系统通常由于蛋白沉淀、易被蛋白酶降解、蛋白翻译后修饰不足等原因而导致一些复杂蛋白不能够顺利表达,因此迫切需要寻求更加快速高效的新技术生产重组蛋白药物。CFPS作为一种高通量和大规模的体外蛋白合成技术,在生物制药领域中具有重要的应用潜力,并且近年来已经逐渐应用于高通量药物筛选、大规模生产重组蛋白药物、多肽类药物、肿瘤疫苗等领域中。
本文总结了近年来CFPS在实现高通量和工业化生产进程中的研究进展,着重介绍其在此过程中遇到的困难及相应的解决方案;详细列出了CFPS在生物制药领域,特别是在重组蛋白药物生产方面的应用;并对该体系目前依然面临的挑战进行了深入的探讨,最后结合本实验室的研究成果,对无细胞表达技术在生物制药领域以及小分子天然产物生产方面的应用价值进行展望。
1 无细胞蛋白表达体系的研究进展理论上任何来源的生物都可以用于制备CFPS,但目前已经比较成熟的系统有E. coli系统(E. coli extract)、小麦胚芽系统(Wheat germ extract,WGE)和兔网织红细胞系统(Rabbit reticulocyte extract,RRE),且均有商业化的试剂盒出售[11, 12, 13]。总体而言,大肠杆菌来源的CFPS具有较高的生产效率、简便的制备工艺以及相对低廉的成本,因而成为最受欢迎的表达系统。在此基础上构建的PURE系统,只包含蛋白翻译所需的酶和辅助因子,大大降低了表达时的遗传背景、反应效率高、下游纯化工艺更加简便[14, 15],但是PURE系统复杂的制备工艺和高昂的成本阻碍了其在高通量蛋白表达和工业化大规模生产领域中的应用。与E. coli来源的CFPS相比,真核生物来源的表达体系虽然生产效率略低,但更适合表达一些复杂蛋白以及对蛋白进行翻译后修饰,常用于蛋白的功能研究。
早期的批次式无细胞反应模式由于体系内维持反应正常进行的底物和能量物质被迅速消耗以及抑制性副产物的大量积累,导致反应持续时间短、蛋白生产效率低。为了解决这一难题,Spirin等提出了连续流动式无细胞反应(Continuous-flow cell-free,CFCF)[2]。CFCF反应模式通过不断地向体系提供底物和能量物质,同时将抑制性副产物移出,保证了反应的顺利进行。随后进一步开发出更加高效的连续交换式无细胞反应[16](Continuous-exchange cell-free,CECF)。在CECF中,反应体系和补充体系之间被一层具有选择通透性的半透膜隔开,通过分子扩散作用实现了底物和能源物质的补充并消除了副产物的抑制作用,从而成功地将反应时间延长至14 h,蛋白产量提高到 1.2 mg/mL。虽然CECF系统可以有效地提高蛋白生产效率,但是高昂的生产成本严重阻碍了其在高通量和工业化生产上的应用。近年来研究人员不断改进无细胞蛋白表达技术,通过优化细胞抽提物的制备工艺、开发价格低廉的能量再生系统和稳定的底物供应、促进表达体系中蛋白的正确折叠等手段,进一步降低生产成本、提高生产效率、扩大反应体系体积。在这些工作的基础上,Zawada等利用E. coli来源的CFPS,通过改进细胞抽提物制备和贮存方式,优化合成的基因序列和体系的氧化还原参数,放大反应过程,简化下游纯化工艺等手段,成功地将反应体积扩大到100 L,并在10 h内合成了700 mg/L具有生物活性的细胞因子(granulocyte-macrophage colony-stimulating factor,GM-CSF)[17]。这一研究成果具有里程碑意义,标志着CFPS逐渐显示出工业大规模生产重组蛋白药物的应用价值。本文总结了近年来CFPS在实现高通量和工业化生产进程中的研究进展,着重介绍其在此过程中遇到的困难及相应的解决方案。
1.1 优化细胞抽提物的制备工艺在细胞抽提物的制备过程中,培养基类型、细胞裂解方式、后续处理工艺等因素对CFPS的生产效率起着决定性作用。以E. coli的细胞抽提物制备工艺为例,传统的操作流程为:菌体在普通培养基中生长到对数中期时开始收集,高压裂解法破碎细胞后,经过2次30000×g高速离心移除细胞碎片和基因组DNA,径流反应后通过透析改变体系中的缓冲液便于贮存[1, 18]。制备过程中涉及到多次高速离心和透析操作,以及成本高昂的径流反应,这些步骤不仅耗时且增加了CFPS的成本,不利于大规模生产,因此建立快速、稳定、成本低的细胞抽提物制备工艺迫在眉睫。
Zawada等通过向培养基中添加氨基酸和维生素,以及控制培养基中葡萄糖的补加速率,实现了菌体的高密度发酵,解决了传统发酵过程中菌体量较低这一难题[19]。随后Liu等在不影响CFPS反应活性的情况下,简化制备步骤成功地将制备时间缩短了一半,同时优化径流反应条件将成本削减了70%[20]。在此基础上,Kim等经过研究发现将传统制备工艺中高速离心、径流反应、透析等操作部分省去后并不会降低CFPS的活性[21]。于是他们进一步简化制备工艺,通过低速离心和缩短径流反应时间,极大地节省时间并降低成本。最近的研究表明通过改变体系中离子类型,采用珠磨法或超声破碎法取代传统的高压匀浆法等方式能够提高CFPS的生产效率,并进一步降低生产成本[22, 23]。细胞抽提物制备工艺的优化使得能够快速构建一个稳定、高效、成本低的反应体系,极大地推进了CFPS高通量表达和工业化生产的发展进程。
1.2 开发价格低廉的能量供应系统蛋白质合成过程中需要消耗大量的能量,研究表明供能不足将直接缩短CFPS的反应时间且降低体系的生产效率,因此选择合适的能量供应系统对于提高体系的生产效率至关重要[24]。通常CFPS内ATP的再生依赖于底物水平磷酸化,即通过向体系中添加高能物质(图 2-A),如磷酸烯醇式丙酮酸(Phosphoenol pyruvate,PEP)、乙酰磷酸、磷酸肌酸等(Creatine phosphate,CP),使其高能磷酸键转移到ADP从而实现ATP再生[18, 25]。然而使用价格高昂的磷酸物质不仅提高了CFPS的成本,而且磷酸物质很容易被细胞抽提物中残存的磷酸水解酶降解,增加体系中无机磷酸的浓度,进而抑制蛋白质的合成。

|
| 图 2. CFPS中的能量供应方式 Figure 2. Different strategies for providing energy for CFPS systems. A: ATP regeneration using PEP as an energy source. B: Energy supply by pyruvate and pyruvate oxidase. C: Energy supply through dual-energy system. PEP, phosphoenolpyruvic acid; ADP, adenosine diphosphate; ATP, adenosine triphosphate; Pi, inorganic phosphate; GAP, glyceraldehyde-3-phosphate; DPG, 1,3-diphosphoglycerate; NAD, nicotinamide adenine dinucleotide; NADH, reduced nicotinamide adenine dinucleotide; CP, creatine kinase. |
为了解决这个难题,Kim和Swartz通过向体系中外源添加丙酮酸氧化酶成功地利用丙酮酸作为能源物质使ATP再生(图 2-B),替代了价格高昂的磷酸烯醇式丙酮酸[26]。在此基础上他们进一步开发出PANOx系统,通过激活CFPS内源的丙酮酸脱氢酶和磷酸乙酰基转移酶,只需向反应体系中添加NAD和CoA就可以利用丙酮酸供能[27]。随后糖酵解的中间产物也被证明可以用于能量再生[28]。为了寻求成本更加低廉的能量再生系统,Calhoun等尝试使用Bis缓冲液代替传统的HEPES,实现了利用葡萄糖为反应体系供能,并用单磷酸代替原先的三磷酸核苷酸,进一步降低了CFPS的生产成本[29, 30]。然而利用葡萄糖为体系供能时,反应初始阶段ATP会被糖酵解途径大量消耗掉,导致体系内能量供应不足。Kim等研发出双供能系统,结合CP和葡萄糖这两种供能方式(图 2-C),很好地解决了这一难题,并将生产效率提高了2-3倍[31]。另外,利用麦芽糖糊精、可溶性淀粉、麦芽糖等代谢缓慢的糖类物质作为能量来源,可以提高反应体系中pH和无机磷酸浓度的稳定性,进而提高体系的生产效率[32, 33, 34]。例如,Caschera和Noireaux利用麦芽糖为能源物质,成功地在10 h内合成了2.3 mg/mL的蛋白质[34]。在过去价格高昂的能量再生系统一直是CFPS成本居高不下的主要原因,目前开发出来的高效、成本低廉的能量再生系统,毫无疑问为CFPS实现工业化大规模生产奠定了坚实的基础。
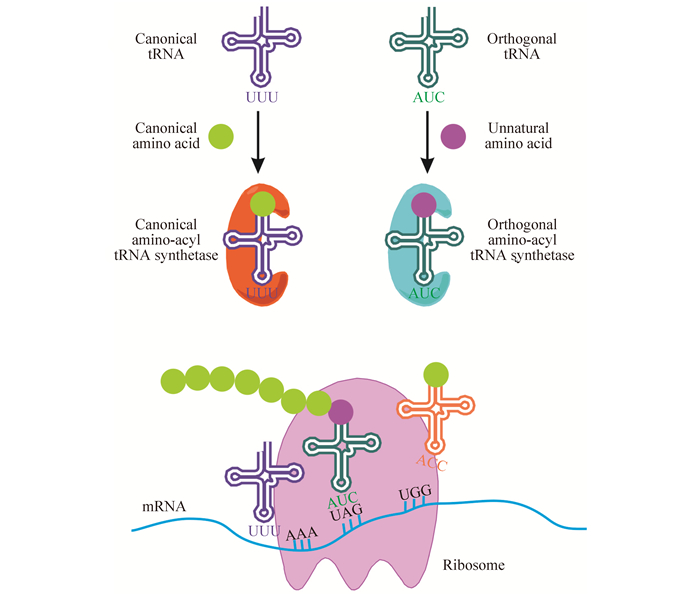
|
| 图 3. 利用CFPS引入非天然氨基酸 Figure 3. Scheme for site-specific incorporation of unnatural amino acids using CFPS systems. |
一系列的研究发现利用CFPS合成蛋白时,部分氨基酸在反应起始30 min内会被大量消耗而导致反应终止,通过补充相应的氨基酸可以有效延长体系反应持续时间,提高蛋白产量[27, 35]。这些结果表明,在CFPS中可能存在未知的酶会降解外源添加的氨基酸。为了稳定底物供应,Swartz课题组利用反向代谢工程手段,在不影响菌体生长的前提下,对用于制备CFPS的E. coli基因组进行改造,旨在防止氨基酸的快速降解[36]。在改造过程中,他们首先确定了CFPS中4种氨基酸,即精氨酸、色氨酸、半胱氨酸和丝氨酸,会在反应初始阶段被大量消耗;其次推断出可能导致氨基酸降解的酶,如精氨酸脱羧酶、色氨酸酶和丝氨酸脱氨酶;最后在E. coli基因组上对相应的基因进行敲除。改进后的CFPS促进了氨基酸的稳定性,延长了体系的反应持续时间,提高了蛋白的生产效率。
1.4 促进蛋白的正确折叠和翻译后修饰大多数的细胞质蛋白均可在CFPS中顺利表达,甚至结构相对复杂的硫氧还蛋白还原酶和谷胱甘肽还原酶,其催化活性需要在翻译后进行二聚化并与辅因子FAD共价结合,也成功地在CFPS中合成[37]。体外合成含有二硫键等复杂结构的蛋白质一直是CFPS的研究重点。在生物体内,蛋白质二硫键的形成通常发生在原核生物的周质空间和真核生物的内质网腔中,这些区域提供的氧化环境可以促进二硫键的形成。然而在CFPS中并没有这些特殊区域,且体系中含有的还原性物质不利于蛋白的折叠。
Swartz等利用碘乙酰胺处理细胞抽提物消除体系的还原性,同时使用谷胱甘肽缓冲液为体系提供一个稳定的氧化环境,并结合使用二硫键异构酶DsbC,在CFPS中合成了有活性的尿激酶和纤维酶原激活物片段[38, 39]。除了直接向反应体系中添加二硫键异构酶,人工合成的方法也能够用于体外促进二硫键的形成。Welsh等通过将大肠杆菌的触发因子与真核生物内质网上的Hsp70分子伴侣BiP融合表达,极大地促进了真核来源的分泌型蛋白在CFPS中的可溶性表达[40]。另外,两性多糖纳米凝胶可以作为有效的人造分子伴侣,在CFPS中促进蛋白折叠并阻止蛋白聚集,该策略已经成功地用于硫氰酸酶的体外表达[41]。针对结构更加复杂的膜蛋白,通常向CFPS中添加温和型去污剂或脂质体来促进其形成正确的构象[42, 43]。
2 无细胞蛋白表达体系在生物制药领域中的应用在过去CFPS由于生产效率低、成本高昂、反应体积小等缺点,通常仅限于基础生物学领域的研究。近年来随着研究人员对细胞抽提物制备工艺、能量再生系统和蛋白折叠等方面进行不断地优化,使得蛋白的体外高通量表达和工业化生产成为现实,并拓展了CFPS的应用范畴。作为一种高通量和大规模的体外蛋白合成技术,CFPS在生物制药领域中具有重要的应用潜力,为解决生物制药业长期面临的问题提供了新的解决方案。本文重点介绍了CFPS在重组蛋白药物的大规模生产、蛋白的高通量表达、非天然氨基酸的引入、类病毒颗粒的表达以及个体化定制肿瘤疫苗等方面的应用。
2.1 重组蛋白药物的大规模生产近年来随着科学家对无细胞蛋白表达技术不断地进行优化,无细胞表达技术渐趋成熟,已经成功地开发出生产效率更高、反应时间更长、反应体积更大的体外蛋白合成体系。此外CFPS在生产效率、蛋白活性和下游纯化工艺方面的明显优势,促进了其在重组蛋白药物生产领域中的广泛应用。重组蛋白药物粒细胞-巨噬细胞集落刺激因子(GM-CSF)是一种重要的细胞因子,其结构中有4个螺旋和2个二硫键,在E. coli中需要在分子伴侣的帮助下才能形成正确的构象[44],并且在其它常用的体内表达系统中产量很低,如在酿酒酵母(Saccharomyces cerevisiae)中产量为50-60 mg/L[45],在黑曲霉(Aspergillusniger)中为0.64 μg/L[46],在毕赤酵母(Pichiapastoris)中为200-250 mg/L[47],昆虫细胞(Insect cell)中为11-45 mg/L[48]。为了提高GM-CSF的表达量,Zawada等利用E. coli来源的CFPS,通过对细胞抽提物制备和贮存、合成的基因序列、氧化还原参数、反应体积放大、下游纯化工艺等过程进行系统的优化,能够成功地在100 L的反应体系中合成700 mg/L有活性的GM-CSF[17]。
除了常用的E. coli来源的CFPS之外,近年来真核系统来源的表达体系也被进一步开发用于生产结构更加复杂的膜蛋白以及进行蛋白的翻译后修饰等。Brodel等用CHO细胞建立了一套新型的哺乳动物CFPS,该体系含有进行蛋白翻译后修饰所必需的辅因子以及促进膜蛋白正确折叠的内源微粒体,因而在表达复杂蛋白方面更具优势[49]。利用这套系统,他们成功地在体外表达了具有生物活性的萤火虫荧光素酶和人促红细胞生成素,并顺利地对其进行糖基化修饰。这些研究成果具有里程碑意义,标志着无细胞蛋白表达技术在生产重组蛋白药物领域中显示出工业生产规模的应用前景。
2.2 蛋白的高通量表达在后基因组时代,科学家面临的一个重大挑战就是系统地阐明基因编码的全部蛋白质的结构、功能和相互作用关系,因此建立快速高效的高通量蛋白表达技术显得尤为重要。近年来,CFPS因其独特的优势而倍受青睐。与传统的体内表达体系相比,CFPS可以直接使用PCR片段作为模板,省去了耗时的分子克隆过程;其次,一些很难在体内系统表达的复杂蛋白,如富含半胱氨酸的蛋白质和结构复杂的膜蛋白,能够在CFPS中正确折叠;与此同时,通过在翻译过程中对蛋白质进行改造,可以获得特殊标记的蛋白[12, 50, 51]。Goshima等通过对麦胚来源的CFPS进行优化,构建了一个“人类蛋白工厂”[52]。在表达的13364种人源蛋白中,大多数均可以在该体系中成功表达,甚至是一些结构复杂的细胞因子也被证明具有细胞活性。CFPS高通量的蛋白合成优势,为研究人员进行体外蛋白质组学的研究,如分析酶与底物之间、蛋白与蛋白之间的相互作用关系、蛋白的翻译后修饰、肽链加工等,提供了一个更加灵活高效的研究手段。
此外,利用CFPS高通量的蛋白表达优势能够高效便捷地制备蛋白芯片[53, 54, 55]。传统的制备技术需要纯化大量的可溶性蛋白,随后与修饰的载玻片共价或非共价结合从而实现固定化。而利用CFPS能够在一步法中实现蛋白的表达与固定化,省去了费时且成本高昂的分子克隆、蛋白表达与纯化等过程。而且CFPS开放、灵活的特点能够实现在蛋白的翻译过程中直接对其进行特异性标记,如同位素标记、荧光标记和生物素标记等,极大地简化了后续的检测步骤。近年来蛋白芯片技术逐步应用于有毒物质的检测和临床诊断,如癌症、免疫和代谢类疾病的诊断[56, 57]。Davies等利用E. coli来源的CFPS成功地将185种病毒蛋白固定在蛋白芯片上,用于测定人和动物接种疫苗后的体液免疫反应[58]。CFPS在高通量蛋白表达和蛋白芯片制备技术上的快速发展,促进了其在疾病诊断、新药发现和疫苗研发领域的应用[59, 60, 61]。
2.3 非天然氨基酸的引入作为一种可以对蛋白进行工程改性的重要方法,编码非天然氨基酸技术一经问世,就得到了广泛关注。早期通过向CFPS中加入可识别特定终止密码子的氨酰化的抑制性tRNA,并以携带有特定位点无义突变的基因作为模板,可以将侧链带有反应基团的非天然氨基酸引入到目标蛋白的特定位点[62]。在此基础上科学家们发展了更加有效的编码非天然氨基酸技术,如利用改造后的tRNA和相应的氨酰tRNA合成酶扩展遗传密码子等[63, 64, 65]。近年来编码非天然氨基酸技术已经成功地用于对蛋白药物进行特异性修饰,赋予其独特的药理特性,如提高蛋白药物的药代动力学稳定性、生物利用度以及延长半衰期等[66, 67]。CFPS由于可以高效便捷地通过在反应体系中添加非天然氨基酸底物,实现对目标蛋白选择性地修饰,因而在开发新型药物蛋白以及蛋白药物升级领域中具有巨大的应用前景[68]。Zimmerman等利用CFPS成功地在曲妥珠(Trastuzumab)单抗中定点插入经过优化的非天然氨基酸pAMF (para-azidomethyl-L-phenylalanine,pAMF),并将携带pAMF的单抗通过无需铜离子催化的SPAAC (Strain-promoted azide−alkyne cycloaddition)点击化学方法与DBCO-PEG-MMAF (DBCO-PEG-monomethylauristatin)高效结合[69]。合成的抗体偶联药物(Antibody-drug conjugates,ADCs)在体外细胞毒性试验中具有更高的靶向特异性和生物利用度。
2.4 类病毒颗粒的表达类病毒颗粒(Virus-like particles,VLPs)是由一种或多种蛋白自我组装成的复合体,与病毒颗粒结构类似,但不包含其传染性的遗传物质DNA或RNA。VLPs具备病毒衣壳蛋白的真实构象,在体内能够引起B细胞介导的免疫应答,且克服了常规病毒疫苗存在复活可能性的缺点,是一种安全高效的亚单位疫苗[70]。到目前为止,科学家们成功研发出30多种VLPs用于治疗病毒引起的疾病,其中乙型肝炎病毒疫苗和人乳头瘤病毒疫苗已经被FDA批准,在世界范围内广泛使用。酵母、杆状病毒、昆虫细胞等表达系统目前在VLPs的生产中占据主导地位,然而产量低和蛋白翻译后修饰不足是这些体内系统面临的两大挑战,并且很难在二者之间找到一个平衡点[71, 72]。结果导致很多VLPs尽管在临床前效果显著,但因无法进行工业化生产而只能应用于小规模基础研究。
CFPS作为体内系统的重要补充,使得许多难以利用常规体内系统表达的蛋白在体外顺利表达。Bundy等利用E. coli来源的CFPS,成功地在体外合成了MS2衣壳蛋白[73]。利用CFPS生产VLPs不仅速度快,而且还可以对其进行表面功能化修饰,赋予其新的功效。例如,Patel等在体外合成VLPs的过程中引入非天然氨基酸进行标记,标记后的VLPs可以进一步通过铜离子催化的点击化学与蛋白质、核酸、聚乙二醇等结合,旨在开发用于药物运载和疾病诊断的新型VLPs生物结合物[74]。此外,他们通过改变CFPS内的氧化还原环境,实现控制VLPs结构中二硫键的形成,进一步提高了VLPs的稳定性[75]。
2.5 肿瘤疫苗的个体化定制B细胞淋巴瘤是由失去控制的B细胞恶性增殖而引发的实体肿瘤,通常情况下体内的免疫系统不能识别异常细胞。值得注意的是,恶性B细胞表面表达的免疫球蛋白的可变区,即独特型,却是特异的,这为治疗B细胞淋巴瘤提供了一个很好的靶标,可以通过注射由独特型蛋白制备的疫苗激活免疫系统从而达到抑制肿瘤的效果。为了增强独特型蛋白的免疫原,Tao等将针对肿瘤细胞的独特型蛋白与粒细胞-巨噬细胞集落刺激因子(granulocyte-macrophage colony-stimulating factor,GM-CSF)融合,不需要使用其它载体蛋白和佐剂[76]。临床数据表明该疫苗可以有效地激活体内的免疫系统控制肿瘤细胞[77]。然而每个B细胞淋巴瘤患者携带的免疫球蛋白的独特型是不一样的,因此需要为不同的病人个体化定制相应的疫苗,这是疫苗生产面临的一个重大挑战。
传统的利用人类细胞作为生物反应器生产疫苗的方法不仅周期长,而且成本高昂,不利于B细胞淋巴瘤疫苗的推广使用。如果利用CFPS生产个体化定制的肿瘤疫苗,那么整个过程将直接从患者的疾病诊断到疫苗生产,中间省去了周期很长的细胞培养阶段,并且降低了病人之间药物交叉污染的风险。Yang等在E. coli来源的CFPS中成功地实现了B细胞淋巴瘤疫苗的可溶性表达,并进一步利用薄膜反应模式扩大了生产,在早期的小鼠试验中,体系合成的疫苗在抑制肿瘤细胞生长方面效果显著[78]。这些研究成果表明CFPS在肿瘤个体化治疗领域中有巨大的应用潜力。
3 总结和展望CFPS作为一种快速高效的体外蛋白合成手段,弥补了传统的基于细胞的体内表达体系的不足,使得许多难以在体内合成的复杂蛋白在体外顺利表达。凭借其高通量和大规模的蛋白表达优势,CFPS为解决生物制药领域中面临的难题提供了新的解决思路。近年来随着研究人员对CFPS不断地进行优化,成功地开发出生产效率高、成本低廉、反应体积大的表达系统,为工业化大规模生产重组蛋白药物奠定了坚实的基础。
虽然CFPS已经广泛应用于生物制药领域,如大规模生产重组蛋白药物、高通量药物筛选、疫苗研发等,但是目前依然存在一些问题亟需解决。首先,E. coli来源的CFPS由于生产效率高、成本低廉而成为最受欢迎的表达体系,其它真核来源的表达体系,如WGE、RRE等,因技术限制仍然不能实现工业化大规模生产[79];其次,在CFPS中,反应持续时间直接决定了蛋白的生产效率,而体系的稳定性、底物供应的稳定性等对反应持续时间起着重要的影响作用;最后,虽然CFPS能够实现对一些结构相对复杂的蛋白进行简单的修饰,如促进二硫键形成等,但是大多数的重组蛋白药物必须经过糖基化、磷酸化、水解切割等更加复杂的翻译后修饰才具备生物活性[80]。因此,在后续改进CFPS的过程中,需要针对上述问题,进一步开发真核生物来源的表达体系、提高反应体系的稳定性和蛋白的翻译后修饰水平。毫无疑问,这些瓶颈问题的突破将会大大拓展CFPS在生物制药领域中的应用,促进其在高通量药物筛选和工业化生产重组蛋白药物等方面的发展。
无细胞表达技术不仅广泛应用于高通量蛋白合成和工业化大规模生产重组蛋白药物等领域,而且近年来其在小分子天然产物生产方面成方兴未艾之势。本实验室在利用无细胞表达技术生产新型生物能源等小分子天然产物方面有着丰富的研究经验,例如成功地在体外重建了E. coli的脂肪酸合成途径,并在此基础上进一步对合成途径中的限制性酶促反应、底物浓度、辅因子浓度、金属离子浓度等因素进行优化,寻求最优比例[81, 82]。此外,刘天罡课题组利用Saccharomyces cerevisiae来源的甲羟戊酸途径(Mevalonate pathway)构建了一个快速高效的体外重建体系,可以用于体外生产一种重要的新型喷气燃料法尼烯[83]。除新型生物能源外,无细胞表达技术在生产一些重要的萜类化合物,如紫杉醇前体紫杉二烯(Taxadiene)、青蒿素(Artemisinin)、番茄红素(Lycopene)等,同样具有巨大的潜在应用价值[83, 84]。虽然目前无细胞表达技术在合成小分子天然产物方面仍处于起始阶段,但是该技术具备的独特的优势,如反应速率快、生产效率高、下游分离纯化工艺简单等优点,使得其在生产新型生物能源以及生物制药方面有着广阔的应用前景。
总之,随着研究人员不断地对无细胞表达技术进行优化,相信在不久的将来,无细胞表达技术将充分发挥其在高通量和大规模生产方面的优势,逐渐显示出在生物制药以及小分子天然产物生产方面的工业化应用价值,为全人类的健康事业做出巨大的贡献!
| [1] | Nirenberg MW, Matthaei JH. The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides. Proceedings of the National Academy of Sciences of the United States of America, 1961, 47(10): 1588-1602. |
| [2] | Spirin AS, Baranov VI, Ryabova LA, Ovodov SY, Alakhov YB. A continuous cell-free translation system capable of producing polypeptides in high yield. Science, 1988, 242(4882): 1162-1164. |
| [3] | Hodgman CE, Jewett MC. Cell-free synthetic biology: thinking outside the cell. Metabolic Engineering, 2012, 14(3): 261-269. |
| [4] | Carlson ED, Gan R, Hodgman CE, Jewett MC. Cell-free protein synthesis: applications come of age. Biotechnology Advances, 2012, 30(5): 1185-1194. |
| [5] | Swartz J. Developing cell-free biology for industrial applications. Journal of Industrial Microbiology and Biotechnology, 2006, 33(7): 476-485. |
| [6] | Katzen F, Chang G, Kudlicki W. The past, present and future of cell-free protein synthesis. Trends in Biotechnology, 2005, 23(3): 150-156. |
| [7] | Leader B, Baca QJ, Golan DE. Protein therapeutics: a summary and pharmacological classification. Nature Reviews Drug Discovery, 2008, 7(1): 21-39. |
| [8] | Pavlou AK, Reichert JM. Recombinant protein therapeutics—success rates, market trends and values to 2010. Nature Biotechnology, 2004, 22(12): 1513-1519. |
| [9] | Walsh G. Biopharmaceutical benchmarks. Nature Biotechnology, 2000, 18(8): 831-833. |
| [10] | Schmidt FR. Recombinant expression systems in the pharmaceutical industry. Applied Microbiology and Biotechnology, 2004, 65(4): 363-372. |
| [11] | Endo Y, Sawasaki T. Cell-free expression systems for eukaryotic protein production. Current Opinion in Biotechnology, 2006, 17(4): 373-380. |
| [12] | Endo Y, Sawasaki T. High-throughput, genome-scale protein production method based on the wheat germ cell-free expression system. Biotechnology Advances, 2003, 21(8): 695-713. |
| [13] | Spirin AS. High-throughput cell-free systems for synthesis of functionally active proteins. Trends in Biotechnology, 2004, 22(10): 538-545. |
| [14] | Shimizu Y, Inoue A, Tomari Y, Suzuki T, Yokogawa T, Nishikawa K, Ueda T. Cell-free translation reconstituted with purified components. Nature Biotechnology, 2001, 19(8): 751-755. |
| [15] | Shimizu Y, Kanamori T, Ueda T. Protein synthesis by pure translation systems. Methods, 2005, 36(3): 299-304. |
| [16] | Kim DM, Choi CY. A semicontinuous prokaryotic coupled transcription/translation system using a dialysis membrane. Biotechnology Progress, 1996, 12(5): 645-649. |
| [17] | Zawada JF, Yin G, Steiner AR, Yang JH, Naresh A, Roy SM, Gold DS, Heinsohn HG, Murray CJ. Microscale to manufacturing scale-up of cell-free cytokine production—a new approach for shortening protein production development timelines. Biotechnology and Bioengineering, 2011, 108(7): 1570-1578. |
| [18] | Zubay G. In vitro synthesis of protein in microbial systems. Annual Review of Genetics, 1973, 7(1): 267-287. |
| [19] | Zawada J, Swartz J. Maintaining rapid growth in moderate-density Escherichia coli fermentations. Biotechnology and Bioengineering, 2005, 89(4): 407-415. |
| [20] | Liu DV, Zawada JF, Swartz JR. Streamlining Escherichia coli S30 extract preparation for economical cell-free protein synthesis. Biotechnology Progress, 2005, 21(2): 460-465. |
| [21] | Kim TW, Keum JW, Oh IS, Choi CY, Park CG, Kim DM. Simple procedures for the construction of a robust and cost-effective cell-free protein synthesis system. Journal of Biotechnology, 2006, 126(4): 554-561. |
| [22] | Sun ZZ, Hayes CA, Shin J, Caschera F, Murray RM, Noireaux V. Protocols for implementing an Escherichia coli based TX-TL cell-free expression system for synthetic biology. Journal of Visualized Experiments, 2013,(79): e50762. |
| [23] | Kwon YC, Jewett MC. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Scientific Reports, 2015, 5: 8663. |
| [24] | Kim HC, Kim DM. Methods for energizing cell-free protein synthesis. Journal of Bioscience and Bioengineering, 2009, 108(1): 1-4. |
| [25] | Ryabova LA, Vinokurov LM, Shekhovtsova EA, Alakhov YB, Spirin AS. Acetyl phosphate as an energy source for bacterial cell-free translation systems. Analytical Biochemistry, 1995, 226(1): 184-186. |
| [26] | Kim DM, Swartz JR. Prolonging cell-free protein synthesis with a novel ATP regeneration system. Biotechnology and Bioengineering, 1999, 66(3): 180-188. |
| [27] | Kim DM, Swartz JR. Prolonging cell-free protein synthesis by selective reagent additions. Biotechnology Progress, 2000, 16(3): 385-390. |
| [28] | Kim DM, Swartz JR. Regeneration of adenosine triphosphate from glycolytic intermediates for cell-free protein synthesis. Biotechnology and Bioengineering, 2001, 74(4): 309-316. |
| [29] | Calhoun KA, Swartz JR. An economical method for cell-free protein synthesis using glucose and nucleoside monophosphates. Biotechnology Progress, 2005, 21(4): 1146-1153. |
| [30] | Calhoun KA, Swartz JR. Energizing cell-free protein synthesis with glucose metabolism. Biotechnology and Bioengineering, 2005, 90(5): 606-613. |
| [31] | Kim TW, Oh IS, Keum JW, Kwon YC, Byun JY, Lee KH, Choi CY, Kim DM. Prolonged cell-free protein synthesis using dual energy sources: combined use of creatine phosphate and glucose for the efficient supply of ATP and retarded accumulation of phosphate. Biotechnology and Bioengineering, 2007, 97(6): 1510-1515. |
| [32] | Wang YR, Zhang YHP. Cell-free protein synthesis energized by slowly-metabolized maltodextrin. BMC Biotechnology, 2009, 9: 58. |
| [33] | Kim HC, Kim TW, Kim DM. Prolonged production of proteins in a cell-free protein synthesis system using polymeric carbohydrates as an energy source. Process Biochemistry, 2011, 46(6): 1366-1369. |
| [34] | Caschera F, Noireaux V. Synthesis of 2.3 mg/mL of protein with an all Escherichia coli cell-free transcription-translation system. Biochimie, 2014, 99: 162-168. |
| [35] | Kim RG, Choi CY. Expression-independent consumption of substrates in cell-free expression system from Escherichia coli. Journal of Biotechnology, 2000, 84(1): 27-32. |
| [36] | Michel-Reydellet N, Calhoun K, Swartz J. Amino acid stabilization for cell-free protein synthesis by modification of the Escherichia coli genome. Metabolic Engineering, 2004, 6(3): 197-203. |
| [37] | Knapp KG, Swartz JR. Cell-free production of active E. coli thioredoxin reductase and glutathione reductase. FEBS Letters, 2004, 559(1-3): 66-70. |
| [38] | Yin G, Swartz JR. Enhancing multiple disulfide bonded protein folding in a cell-free system. Biotechnology and Bioengineering, 2004, 86(2): 188-195. |
| [39] | Kim DM, Swartz JR. Efficient production of a bioactive, multiple disulfide-bonded protein using modified extracts of Escherichia coli. Biotechnology and Bioengineering, 2004, 85(2): 122-129. |
| [40] | Welsh JP, Bonomo J, Swartz JR. Localization of BiP to translating ribosomes increases soluble accumulation of secreted eukaryotic proteins in an Escherichia coli cell-free system. Biotechnology and Bioengineering, 2011, 108(8): 1739-1748. |
| [41] | Sasaki Y, Asayama W, Niwa T, Sawada SI, Ueda T, Taguchi H, Akiyoshi K. Amphiphilic polysaccharide nanogels as artificial chaperones in cell-free protein synthesis. Macromolecular Bioscience, 2011, 11(6): 814-820. |
| [42] | Klammt C, Löhr F, Schäfer B, Haase W, Dötsch V, Rüterjans H, Glaubitz C, Bernhard F. High level cell-free expression and specific labeling of integral membrane proteins. European Journal of Biochemistry, 2004, 271(3): 568-580. |
| [43] | Klammt C, Schwarz D, Fendler K, Haase W, Dötsch V, Bernhard F. Evaluation of detergents for the soluble expression of α-helical and β-barrel-type integral membrane proteins by a preparative scale individual cell-free expression system. FEBS Journal, 2005, 272(23): 6024-6038. |
| [44] | Bergès H, Joseph-Liauzun E, Fayet O. Combined effects of the signal sequence and the major chaperone proteins on the export of human cytokines in Escherichia coli. Applied and Environmental Microbiology, 1996, 62(1): 55-60. |
| [45] | Price V, Mochizuki D, March CJ, Cosman D, Deeley MC, Klinke R, Clevenger W, Gillis S, Baker P, Urdal D. Expression, purification and characterization of recombinant marine granulocyte-macrophage colony-stimulating factor and bovine interleukin-2 from yeast. Gene, 1987, 55(2/3): 287-293. |
| [46] | Kim MJ, Kwon TH, Jang YS, Yang MS, Kim DH. Expression of murine GM-CSF in recombinant Aspergillus niger. Journal of Microbiology and Biotechnology, 2000, 10(3): 287-292. |
| [47] | Sainathan SK, Tu LW, Bishnupuri KS, Han M, Li AG, Newberry RD, McDonald KG, Crimmins DL, Houchen C, Anant S, Dieckgraefe BK. PEGylated murine granulocyte-macrophage colony-stimulating factor: production, purification, and characterization. Protein Expression and Purification, 2005, 44(2): 94-103. |
| [48] | Chiou CJ, Wu MC. Expression of human granulocyte-macrophage colony-stimulating factor gene in insect cells by a baculovirus vector. FEBS Letters, 1990, 259(2): 249-253. |
| [49] | Brödel AK, Sonnabend A, Kubick S. Cell-free protein expression based on extracts from CHO cells. Biotechnology and Bioengineering, 2014, 111(1): 25-36. |
| [50] | Rungpragayphan S, Nakano H, Yamane T. PCR-linked in vitro expression: a novel system for high-throughput construction and screening of protein libraries. FEBS Letters, 2003, 540 (1/3): 147-150. |
| [51] | He MY. Cell-free protein synthesis: applications in proteomics and biotechnology. New Biotechnology, 2008, 25(2/3): 126-132. |
| [52] | Goshima N, Kawamura Y, Fukumoto A, Miura A, Honma R, Satoh R, Wakamatsu A, Yamamoto JI, Kimura K, Nishikawa T, Andoh T, Iida Y, Ishikawa K, Ito E, Kagawa N, Kaminaga C, Kanehori K, Kawakami B, Kenmochi K, Kimura R, Kobayashi M, Kuroita T, Kuwayama H, Maruyama Y, Matsuo K, Minami K, Mitsubori M, Mori M, Morishita R, Murase A, Nishikawa A, Nishikawa S, Okamoto T, Sakagami N, Sakamoto Y, Sasaki Y, Seki T, Sono S, Sugiyama A, Sumiya T, Takayama T, Takayama Y, Takeda H, Togashi T, Yahata K, Yamada H, Yanagisawa Y, Endo Y, Imamoto F, Kisu Y, Tanaka S, Isogai T, Imai JI, Watanabe S, Nomura N. Human protein factory for converting the transcriptome into an in vitro-expressed proteome. Nature Methods, 2008, 5(12): 1011-1017. |
| [53] | He MY, Stoevesandt O, Taussig MJ. In situ synthesis of protein arrays. Current Opinion in Biotechnology, 2008, 19(1): 4-9. |
| [54] | Chandra H, Srivastava S. Cell-free synthesis-based protein microarrays and their applications. Proteomics, 2010, 10(4): 717-730. |
| [55] | Ramachandran N, Hainsworth E, Bhullar B, Eisenstein S, Rosen B, Lau AY, Walter JC, LaBaer J. Self-assembling protein microarrays. Science, 2004, 305(5680): 86-90. |
| [56] | Pollard HB, Srivastava M, Eidelman O, Jozwik C, Rothwell SW, Mueller GP, Jacobowitz DM, Darling T, Guggino WB, Wright J, Zeitlin PL, Paweletz CP. Protein microarray platforms for clinical proteomics. Proteomics — Clinical Applications, 2007, 1(9): 934-952. |
| [57] | Mei Q, Fredrickson CK, Jin SG, Fan ZH. Toxin detection by a miniaturized in vitro protein expression array. Analytical Chemistry, 2005, 77(17): 5494-5500. |
| [58] | Davies DH, Liang X, Hernandez JE, Randall A, Hirst S, Mu Y, Romero KM, Nguyen TT, Kalantari-Dehaghi M, Crotty S, Baldi P, Villarreal LP, Felgner PL. Profiling the humoral immune response to infection by using proteome microarrays: high-throughput vaccine and diagnostic antigen discovery. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(3): 547-552. |
| [59] | Sanchez-Carbayo M, Socci ND, Lozano JJ, Haab BB, Cordon-Cardo C. Profiling bladder cancer using targeted antibody arrays. The American Journal of Pathology, 2006, 168(1): 93-103. |
| [60] | Fang Y, Lahiri J, Picard L. G protein-coupled receptor microarrays for drug discovery. Drug Discovery Today, 2003, 8(16): 755-761. |
| [61] | Tsuboi T, Takeo S, Iriko H, Jin L, Tsuchimochi M, Matsuda S, Han ET, Otsuki H, Kaneko O, Sattabongkot J, Udomsangpetch R, Sawasaki T, Torii M, Endo Y. Wheat germ cell-free system-based production of malaria proteins for discovery of novel vaccine candidates. Infection and Immunity, 2008, 76(4): 1702-1708. |
| [62] | Noren CJ, Anthony-Cahill SJ, Griffith MC, Schultz PG. A general method for site-specific incorporation of unnatural amino acids into proteins. Science, 1989, 244(4901): 182-188. |
| [63] | Kowal AK, Köhrer C, RajBhandary UL. Twenty-first aminoacyl-tRNA synthetase–suppressor tRNA pairs for possible use in site-specific incorporation of amino acid analogues into proteins in eukaryotes and in eubacteria. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(5): 2268-2273. |
| [64] | Hirao I, Ohtsuki T, Fujiwara T, Mitsui T, Yokogawa T, Okuni T, Nakayama H, Takio K, Yabuki T, Kigawa T, Kodama K, Yokogawa T, Nishikawa K, Yokoyama S. An unnatural base pair for incorporating amino acid analogs into proteins. Nature Biotechnology, 2002, 20(2): 177-182. |
| [65] | Wang L, Brock A, Herberich B, Schultz PG. Expanding the genetic code of Escherichia coli. Science, 2001, 292(5516): 498-500. |
| [66] | Cho H, Daniel T, Buechler YJ, Litzinger DC, Maio Z, Putnam A-MH, Kraynov VS, Sim B-C, Bussell S, Javahishvili T, Kaphle S, Viramontes G, Ong M, Chu S, Gc B, Lieu R, Knudsen N, Castiglioni P, Norman TC, Axelrod DW, Hoffman AR, Schultz PG, Dimarchi RD, Kimmel BE. Optimized clinical performance of growth hormone with an expanded genetic code. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(22): 9060-9065. |
| [67] | Tian F, Lu YC, Manibusan A, Sellers A, Tran H, Sun Y, Phuong T, Barnett R, Hehli B, Song F, DeGuzman MJ, Ensari S, Pinkstaff JK, Sullivan LM, Biroc SL, Cho H, Schultz PG, DiJoseph J, Dougher M, Ma DS, Dushin R, Leal M, Tchistiakova L, Feyfant E, Gerber HP, Sapra P. A general approach to site-specific antibody drug conjugates. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(5): 1766-1771. |
| [68] | Hong SH, Kwon YC, Jewett MC. Non-standard amino acid incorporation into proteins using Escherichia coli cell-free protein synthesis. Frontiers in Chemistry, 2014, 2: 34. |
| [69] | Zimmerman ES, Heibeck TH, Gill A, Li XF, Murray CJ, Madlansacay MR, Tran C, Uter NT, Yin G, Rivers PJ, Yam AY, Wang WD, Steiner AR, Bajad SU, Penta K, Yang WJ, Hallam TJ, Thanos CD, Sato AK. Production of site-specific antibody–drug conjugates using optimized non-natural amino acids in a cell-free expression system. Bioconjugate Chemistry, 2014, 25(2): 351-361. |
| [70] | Noad R, Roy P. Virus-like particles as immunogens. Trends in Microbiology, 2003, 11(9): 438-444. |
| [71] | Mellado MCM, Mena JA, Lopes A, Ramírez OT, Carrondo MJT, Palomares LA, Alves PM. Impact of physicochemical parameters on in vitro assembly and disassembly kinetics of recombinant triple-layered rotavirus-like particles. Biotechnology and Bioengineering, 2009, 104(4): 674-686. |
| [72] | Roldão A, Mellado MCM, Castilho LR, Carrondo MJ, Alves PM. Virus-like particles in vaccine development. Expert Review of Vaccines, 2010, 9(10): 1149-1176. |
| [73] | Bundy BC, Franciszkowicz MJ, Swartz JR. Escherichia coli-based cell-free synthesis of virus-like particles. Biotechnology and Bioengineering, 2008, 100(1): 28-37. |
| [74] | Patel KG, Swartz JR. Surface functionalization of virus-like particles by direct conjugation using azide−alkyne click chemistry. Bioconjugate Chemistry, 2011, 22(3): 376-387. |
| [75] | Bundy BC, Swartz JR. Efficient disulfide bond formation in virus-like particles. Journal of Biotechnology, 2011, 154(4): 230-239. |
| [76] | Tao MH, Levy R. Idiotype/granulocyte-macrophage colony-stimulating factor fusion protein as a vaccine for B-cell lymphoma. Nature, 1993, 362(6422): 755-758. |
| [77] | Hsu FJ, Caspar CB, Czerwinski D, Kwak LW, Liles TM, Syrengelas A, Taidi-Laskowski B, Levy R. Tumor-specific idiotype vaccines in the treatment of patients with B-cell lymphoma — long-term results of a clinical trial. Blood, 1997, 89(9): 3129-3135. |
| [78] | Yang JH, Kanter G, Voloshin A, Michel-Reydellet N, Velkeen H, Levy R, Swartz JR. Rapid expression of vaccine proteins for B-cell lymphoma in a cell-free system. Biotechnology and Bioengineering, 2005, 89(5): 503-511. |
| [79] | Shin J, Noireaux V. An E. coli cell-free expression toolbox: application to synthetic gene circuits and artificial cells. ACS Synthetic Biology, 2012, 1(1): 29-41. |
| [80] | Walsh G, Jefferis R. Post-translational modifications in the context of therapeutic proteins. Nature Biotechnology, 2006, 24(10): 1241-1252. |
| [81] | Liu TG, Vora H, Khosla C. Quantitative analysis and engineering of fatty acid biosynthesis in E. coli. Metabolic Engineering, 2010, 12(4): 378-386. |
| [82] | Yu XY, Liu TG, Zhu FY, Khosla C. In vitro reconstitution and steady-state analysis of the fatty acid synthase from Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(46): 18643-18648 |
| [83] | Zhu FY, Zhong XF, Hu MZ, Lu L, Deng ZX, Liu TG. In vitro reconstitution of mevalonate pathway and targeted engineering of farnesene overproduction in Escherichia coli. Biotechnology and Bioengineering, 2014, 111(7): 1396-1405. |
| [84] | Liu TG, Khosla C. A balancing act for taxol precursor pathways in E. coli. Science, 2010, 330(6000): 44-45. |
 2016, Vol. 56
2016, Vol. 56




