中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 孙东昌, 裘娟萍
- Dongchang Sun, Juanping Qiu
- Ⅰ-E型CRISPR/Cas系统介导适应性免疫分子机制研究进展
- Advances in molecular mechanisms of adaptive immunity mediated by type Ⅰ-E CRISPR/Cas system-A review
- 微生物学报, 2016, 56(1): 1-7
- Acta Microbiologica Sinica, 2016, 56(1): 1-7
-
文章历史
- 收稿日期:2015-03-25
- 修回日期:2015-05-18
生物可通过水平基因转移(horizontal gene transfer,HGT)的方式从其它物种基因组、噬菌体或质粒上获取外源基因,从而更好地应对外界环境压力[1, 2, 3]。例如,在许多细菌和古菌基因组上存在一套保守的和外源DNA摄取相关的基因,它们可编码一套用于组装DNA摄取装置的蛋白,从而主动地将外源DNA摄入细胞并整合至其基因组上[4]。然而,并非所有的外源DNA都有利于它们的生存竞争。例如,经噬菌体病毒DNA感染的细菌和古菌往往最终被杀死或裂解。
为抵御病原体入侵,真核生物进化出了一套依赖免疫球蛋白的适应性免疫(adaptive immunity)机制。长期以来,人们认为原核生物中不存在适应性免疫机制。近年来,人们在原核生物基因组中发现成簇出现的规律间隔的短回文重复序列(CRISPR,clustered regularly interspaced short palindromic repeats),在相关Cas (CRISPR associated)蛋白的介导下它的转录产物可特异地识别曾入侵宿主菌的外源DNA并将其降解[5, 6]。CRISPR/Cas系统不仅能够防御噬菌体侵染,还能阻止单链形式进入受体菌的转化DNA和接合DNA的入侵[7, 8, 9]。这种RNA介导的适应性免疫方式能阻断目前已知的几乎所有类型的水平基因转移方式。本文在简要介绍CRISPR/Cas系统的结构、分类和作用阶段的基础上,重点阐述Ⅰ型CRISPR/Cas系统的作用机制和调控机制。
1 CRISPR/Cas的结构、分类和作用阶段CRISPR由不连续的重复序列(repeat)、长度相似的间隔区序列(spacer)和前导序列(leader)所组成。外源DNA上与CRISPR序列的间隔区相匹配的序列被称作原型间隔区(protospacer)[6]。CRISPR序列的转录产物crRNA(CRISPR RNA)经剪切形成小RNA后与其上游保守基因编码的Cas蛋白形成核酸蛋白复合体(CRISPR-associated complex for antiviral defence,Cascade),识别并降解通过水平基因转移进入宿主菌的外源DNA[6]。
CRISPR/Cas系统主要存在3种基本类型(Ⅰ型、Ⅱ型和Ⅲ型),其中cas1是CRISPR/Cas系统的特征基因[10]。Ⅰ型CRISPR/Cas系统广泛存在于细菌和古菌中,主要标志是一个N端含HD磷酸水解酶结构域和C端含DExH解旋酶结构域的Cas3蛋白[10]。基于cas基因和CRISPR重复序列的系统进化特征,Ⅰ型CRISPR/Cas (CRISPR/Cas3)系统可分为6个亚型(A–F),其中大肠杆菌CRISPR/Cas系统为Ⅰ-E型[10, 11]。Ⅱ型CRISPR/Cas系统仅在细菌中发现,其最主要标志是一个具多重功能的大分子蛋白Cas9[10]。CRISPR/Cas9系统已广泛应用于包括哺乳动物在内的许多物种的基因组工程中[12]。Ⅲ型CRISPR/Cas系统仅在古菌中发现,由CRISPR特异性内切核酸酶Cas6和预测参与靶标DNA干涉的Cas10等蛋白组成[10]。
CRISPR/Cas系统免疫防御来自噬菌体或质粒的外源DNA入侵机制分为3个阶段:适应(adaptation)、表达(expression)和干涉(interference)[10]。在CRISPR/Cas系统的适应阶段,Cas1-Cas2组成的整合酶将来自外源DNA的原型间隔区域序列整合至基因组CRISPR序列相应位置中[13]。各类型的CRISPR/Cas系统在表达和干涉阶段存在较大差异。在crRNA成熟过程中,Cas6同源蛋白加工Ⅰ型和Ⅲ型CRISPR/Cas系统中crRNA,而Ⅱ型CRISPR系统中Cas9和RNase Ⅲ加工crRNA;在与靶DNA相结合过程中,Ⅰ型和Ⅲ型CRISPR系统中crRNA与多个蛋白组成复合体与靶DNA相结合,而Ⅱ型CRISPR系统中crRNA与单个蛋白(Cas9)组成复合体;在靶DNA切割识别方面,Ⅰ型和Ⅱ型CRISPR系统均通过原型间隔区邻近区域上一个3–5核苷酸组成的基序(protospacer adjacent motif,PAM)识别靶标DNA,而Ⅲ型CRISPR系统中未见类似保守基序的报道[10]。大肠杆菌中Ⅰ-E型CRISPR/Cas系统是目前研究最透彻的CRISPR/Cas系统。后文中,作者将重点阐述其作用机制和调控机制。
2 Ⅰ-E型CRISPR/Cas系统的作用机制Ⅰ-E型CRISPR/Cas系统由CRISPR序列和Cas蛋白(Cas1-3和CasA-E)组成,Cas1和Cas2蛋白通过识别位于靶标DNA原型间隔区3′端PAM基序(通常为CTT),进而将原型间隔区DNA片段整合到自身CRISPR的重复序列之间,形成新的间隔区[11, 13]。当再次遭受类似外源DNA入侵时,大肠杆菌表达cas基因和CRISPR序列:cas基因转录并翻译成相关的Cas蛋白,其中包括Cas1-3和CasA-E(又称作Cas1、Cas2、Cas7、Cas5e和Cas6e);CRISPR转录为前体crRNA(pre-crRNA)(图1)[10]。前体crRNA经核酸酶CasE(Cas6e)加工后形成成熟的crRNA[14]。与成熟crRNA继续结合的CasE招募CasA-D形成Cascade复合体[15]。在Cas3的介导下,Cascade与再次入侵的外源DNA上原型间隔区相结合并在PAM位点附近切割靶标DNA,从而导致靶标DNA降解(图1)[15]。
通过X射线分析,人们解析了Cascade复合体的结构[15]。它由1个CasE、5个CasC、2个CasB、1个CasD、1个CasA和1个crRNA组成(图1)。Cascade复合体呈海马形状,由头部、骨架部分和尾部三部分组成:头部由CasE与crRNA3’端茎环(stem-loop)组成,骨架部分由5个CasC单体与crRNA中部(belly)的引导序列组成,尾部由5’端源自CRISPR重复区的8碱基核苷酸手柄(handle)与CasA和CasD组成(图1)[15]。在头部和骨架部分组装时,CasE为最前端CasC提供结合位点;在骨架部分和尾部组装时,CasD终止CasC在crRNA尾部延伸[15]。除CasC外,CasE同时也与CasB相互作用。虽然CasB不与crRNA直接作用,但是CasB形成的二聚体两侧均带正电,暗示它可能在稳定被结合和置换的靶标DNA方面发挥作用[15]。目前,Cascade复合体与靶标DNA相结合的结构未见报道,因此CRISPR如何通过PAM基序识别靶标DNA以及Cascade复合体中crRNA如何介导Cas3切割靶标DNA等关键问题仍有待进一步探索。
3 Ⅰ-E型CRISPR/Cas系统的调控机制在大肠杆菌Ⅰ-E型CRISPR/Cas系统转录起始阶段,cas3、casA-E-cas1,2和CRISPR分别由Pcas3、Pcas和PCRISPR3个启动子所转录[16]。其中Pcas3为组成型启动子,Pcas和PCRISPR上均存在阻遏蛋白H-NS的结合位点[16]。此外,Pcas上还存在转录激活蛋白LeuO和全局调控因子cAMP-CRP(cAMP activated global transcriptional regulator)的结合位点[17, 18]。
DNA结合蛋白H-NS(histone-like nucleoid structure protein)可与启动子附近富含AT的DNA序列相结合,并在DNA上招募其它H-NS分子,使得成串相连的H-NS在DNA上形成核酸蛋白复合体,从而改变相应区域启动子的二级结构,阻止RNA聚合酶与该区域启动子结合,进而抑制相关基因转录[19]。LeuO是CRISPR/Cas系统表达的激活因子,它可与Pcas上游序列结合,其结合区域与H-NS结合区域部分重合(图2)[17]。通常,LeuO编码基因受H-NS抑制作用无法表达。过量表达LeuO可解除H-NS对Pcas的阻遏作用,从而激活CRISPR/Cas系统表达[17]。BglJ和RcsB组成的异源二聚体是一个多向转录调节因子,可协同或拮抗全局调控因子( 如CRP、LeuO和H-NS)的转录调控作用[20]。通过持续地表达BglJ从而增加BglJ-RcsB,可提高LeuO编码基因的转录表达,从而激活CRISPR/Cas系统的启动子Pcas(图2)[21]。 H-NS和LeuO的相对表达水平决定了同时受二者调控基因表达的开启与关闭。例如:在大肠杆菌指数生长时期,H-NS相对表达量较高,抑制LeuO表达,导致相关基因的表达下调;进入稳定生长期后,H-NS表达量逐渐降低,而LeuO表达量则持续升高,导致相关基因的表达上调[22]。然而,目前尚不清楚进入稳定生长期之后,CRISPR/Cas系统组分基因的转录表达是否因H-NS和LeuO相对表达量变化而被激活。
部分因子可在不依赖H-NS的条件下调控CRISPR/Cas系统。CRP是环磷腺苷(cAMP)的受体蛋白,可负调控CRISPR/Cas系统[18]。增加葡萄糖浓度可降低细胞内cAMP水平,导致cAMP-CRP浓度随之降低,从而解除cAMP-CRP对Pcas的抑制作用,提高CRISPR/Cas的表达水平,增强宿主菌对P1噬菌体感染的免疫能力[18]。在大肠杆菌Pcas上存在CRP结合位点,它与LeuO结合位点部分重合(图2)[18]。或许,CRP可与激活因子LeuO竞争Pcas上结合位点,从而阻遏CRISPR/Cas系统表达[18]。BaeSR双组分调控系统(two-component BaeSR regulatory system)由定位在细胞膜上的组氨酸激酶(histidine kinase)BaeS和胞浆中与DNA结合的反应调节因子(DNA-binding response regulator)BaeR组成[23]。在膜压力(membrane stress)作用下,活化的BaeS导致BaeR磷酸化,从而激活相关基因转录[24]。膜压力通过BaeSR双组分调节系统可不经H-NS激活CRISPR/Cas系统(图2)[25],但相关调控机制仍不清楚。
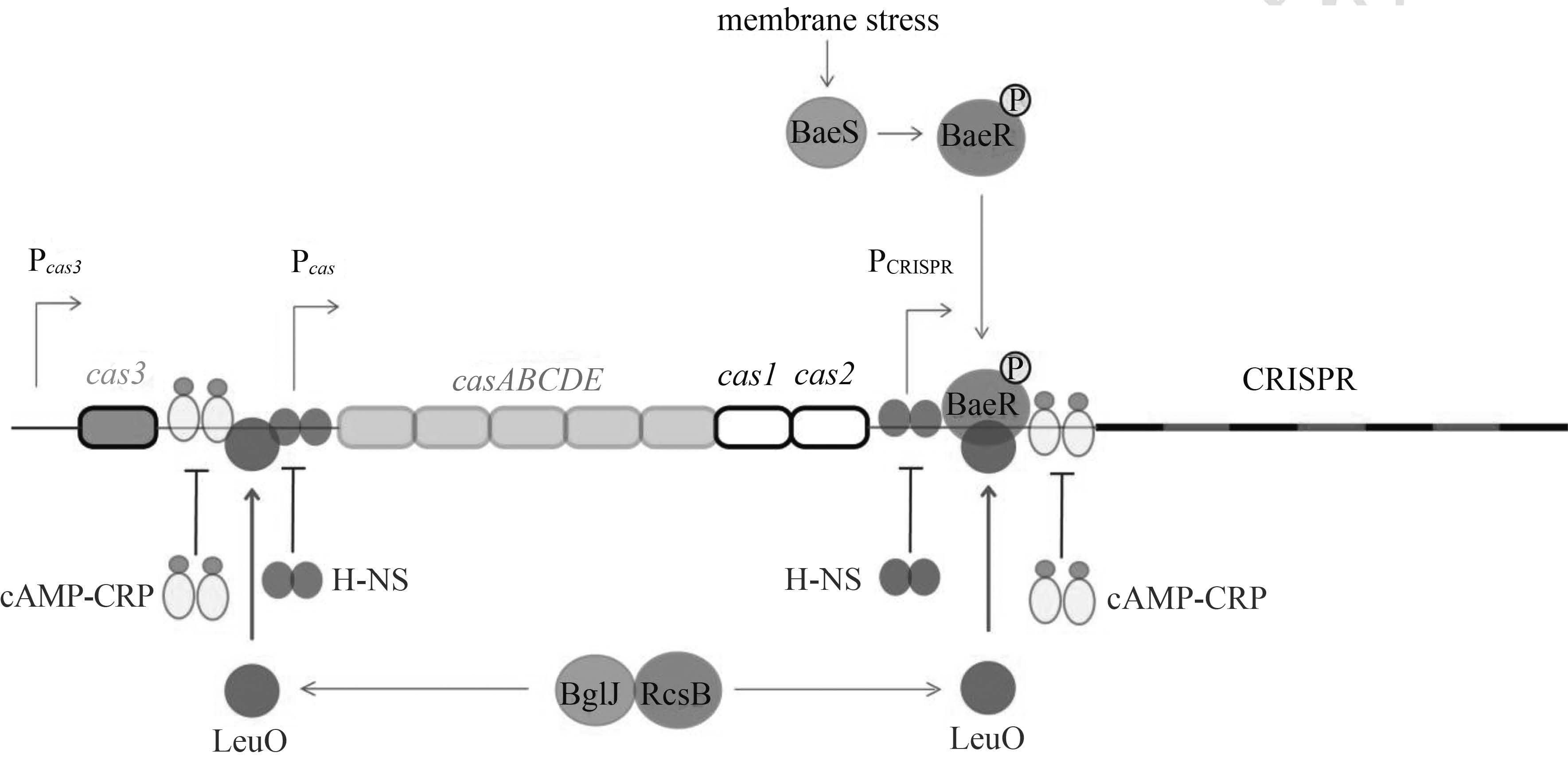
|
| 图2 Ⅰ-E型CRISPR/Cas系统的转录调控机制 Figure2 The mechanism of the transcriptional regulation of the type Ⅰ-E CRISPR/Cas system. |
真核生物免疫球蛋白介导的适应性免疫系统主要通过抗体抗原特异结合的方式识别进入的病原体,而原核生物中CRISPR介导的适应性免疫则主要通过RNA与DNA序列匹配的方式识别入侵的外源DNA。前者无需其它大分子介导,通过免疫球蛋白分子上的抗原结合簇与抗原分子上的抗原决定簇相互吸引实现抗原抗体的识别;而后者需通过Cas蛋白和PAM基序实现crRNA与靶标DNA的识别。通常情况下真核生物的适应性免疫系统处于未激活状态,当宿主受病原体侵染时该免疫系统被激活。类似的,大肠杆菌中Ⅰ-E型CRISPR/Cas系统因H-NS的阻遏作用处于关闭状态,然而目前仍然不清楚何种自然条件下该CRISPR/Cas系统可解除H-NS的阻遏作用,从而发挥其功能。大肠杆菌进入稳定生长期后,随着营养物质的匮乏,CRISPR/Cas系统的阻遏蛋白H-NS表达水平逐渐降低,而激活蛋白LeuO的表达水平却逐渐增高。笔者曾在自然条件下质粒入侵稳定生长期大肠杆菌的分子机制方面开展了大量工作[26, 27, 28, 29]。或许,随着H-NS表达的降低和LeuO表达的增高,在某种自然条件下稳定生长期的大肠杆菌中CRISPR/Cas系统可被激活,用于防御外源质粒DNA的入侵。另外,葡萄糖浓度增加导致cAMP-CRP浓度降低或膜压力通过BaeSR双组分系统也可激活CRISPR/Cas系统,但这两种CRISPR/Cas系统激活的方式却并不依赖H-NS。这些不解除H-NS阻遏的条件下CRISPR/Cas系统激活的机制仍有待进一步探索。
| [1] | Juhas M. Horizontal gene transfer in human pathogens. Critical Reviews in Microbiology, 2015, 41(1):101-108. |
| [2] | Polz MF, Alm EJ, Hanage WP. Horizontal gene transfer and the evolution of bacterial and archaeal population structure. Trends in Genetics, 2013, 29(3):170-175. |
| [3] | Soanes D, Richards TA. Horizontal gene transfer in eukaryotic plant pathogens. Annual Review of Phytopathology, 2014, 52:583-614. |
| [4] | Johnston C, Martin B, Fichant G, Polard P, Claverys JP. Bacterial transformation:distribution, shared mechanisms and divergent control. Nature Reviews Microbiology, 2014, 12(3):181-196. |
| [5] | Makarova KS, Haft DH, Barrangou R, Brouns SJJ, Charpentier E, Horvath P, Moineau S, Mojica FJM, Wolf YI, Yakunin AF, van der Oost J, Koonin EV. Evolution and classification of the CRISPR-Cas systems. Nature Reviews Microbiology, 2011, 9(6):467-477. |
| [6] | Westra ER, Swarts DC, Staals RHJ, Jore MM, Brouns SJJ, van der Oost J. The CRISPRs, they are a-changin':how prokaryotes generate adaptive immunity. Annual Review of Genetics, 2012, 46:311-339. |
| [7] | Bikard D, Hatoum-Aslan A, Mucida D, Marraffini LA. CRISPR interference can prevent natural transformation and virulence acquisition during in vivo bacterial infection. Cell Host & Microbe, 2012, 12(2):177-186. |
| [8] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science, 2008, 322(5909):1843-1845. |
| [9] | Zhang Y, Heidrich N, Ampattu BJ, Gunderson CW, Seifert HS, Schoen C, Vogel J, Sontheimer EJ. Processing-independent CRISPR RNAs limit natural transformation in Neisseria meningitidis. Molecular Cell, 2013, 50(4):488-503. |
| [10] | Sorek R, Lawrence CM, Wiedenheft B. CRISPR-mediated adaptive immune systems in bacteria and archaea. Annual Review of Biochemistry, 2013, 82:237-266. |
| [11] | Kiro R, Goren MG, Yosef I, Qimron U. CRISPR adaptation in Escherichia coli subtype I-E system. Biochemical Society Transactions, 2013, 41(6):1412-1415. |
| [12] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6):1262-1278. |
| [13] | Nuñez JK, Lee ASY, Engelman A, Doudna JA. Integrasemediated spacer acquisition during CRISPR-Cas adaptive immunity. Nature, 2015, 519(7542):193-198. |
| [14] | Carte J, Wang RY, Li H, Terns RM, Terns MP. Cas6 is an endoribonuclease that generates guide RNAs for invader defense in prokaryotes. Genes & Development, 2008, 22(24):3489-3496. |
| [15] | Jackson RN, Golden SM, van Erp PBG, Carter J, Westra ER, Brouns SJJ, van der Oost J, Terwilliger TC, Read RJ, Wiedenheft B. Crystal structure of the CRISPR RNA-guided surveillance complex from Escherichia coli. Science, 2014, 345(6203):1473-1479. |
| [16] | Pul Ü, Wurm R, Arslan Z, Geißen R, Hofmann N, Wagner R. Identification and characterization of E. coli CRISPR-cas promoters and their silencing by H-NS. Molecular Microbiology, 2010, 75(6):1495-1512. |
| [17] | Westra ER, Pul Ü, Heidrich N, Jore MM, Lundgren M, Stratmann T, Wurm R, Raine A, Mescher M, Van Heereveld L, Mastop M, Wagner EG, Schnetz K, Van Der Oost J, Wagner R, Brouns SJJ. H-NS-mediated repression of CRISPR-based immunity in Escherichia coli K12 can be relieved by the transcription activator LeuO. Molecular Microbiology, 2010, 77(6):1380-1393. |
| [18] | Yang CD, Chen YH, Huang HY, Huang HD, Tseng CP. CRP represses the CRISPR/Cas system in Escherichia coli:evidence that endogenous CRISPR spacers impede phage P1 replication. Molecular Microbiology, 2014, 92(5):1072-1091. |
| [19] | Will WR, Navarre WW, Fang FC. Integrated circuits:how transcriptional silencing and counter-silencing facilitate bacterial evolution. Current Opinion in Microbiology, 2015, 23:8-13. |
| [20] | Salscheider SL, Jahn A, Schnetz K. Transcriptional regulation by BglJ-RcsB, a pleiotropic heteromeric activator in Escherichia coli. Nucleic Acids Research, 2014, 42(5):2999-3008. |
| [21] | Arslan Z, Stratmann T, Wurm R, Wagner R, Schnetz K, Pul Ü. RcsB-BglJ-mediated activation of Cascade operon does not induce the maturation of CRISPR RNAs in E. coli K12. RNA Biology, 2013, 10(5):708-715. |
| [22] | Shimada T, Bridier A, Briandet R, Ishihama A. Novel roles of LeuO in transcription regulation of E. coli genome:antagonistic interplay with the universal silencer H-NS. Molecular Microbiology, 2011, 82(2):378-397. |
| [23] | Raffa RG, Raivio TL. A third envelope stress signal transduction pathway in Escherichia coli. Molecular Microbiology, 2002, 45(6):1599-1611. |
| [24] | Leblanc SKD, Oates CW, Raivio TL. Characterization of the induction and cellular role of the BaeSR two-component envelope stress response of Escherichia coli. Journal of Bacteriology, 2011, 193(13):3367-3375. |
| [25] | Perez-Rodriguez R, Haitjema C, Huang QQ, Nam KH, Bernardis S, Ke AL, DeLisa MP. Envelope stress is a trigger of CRISPR RNA-mediated DNA silencing in Escherichia coli. Molecular Microbiology, 2011, 79(3):584-599. |
| [26] | Sun DC, Wang B, Zhu LH, Chen MY, Zhan LL. Block and boost DNA transfer:opposite roles of OmpA in natural and artificial transformation of Escherichia coli. PLoS One, 2013, 8(3):e59019. |
| [27] | Sun DC, Zhang XW, Wang LY, Prudhomme M, Xie ZX, Martin B, Claverys JP. Transforming DNA uptake gene orthologs do not mediate spontaneous plasmid transformation in Escherichia coli. Journal of Bacteriology, 2009, 191(3):713-719. |
| [28] | Sun DC, Zhang YM, Mei YJ, Jiang H, Xie ZX, Liu HH, Chen XD, Shen P. Escherichia coli is naturally transformable in a novel transformation system. FEMS Microbiology Letters, 2006, 265(2):249-255. |
| [29] | Zhang YM, Shi CY, Yu JF, Ren JJ, Sun DC. RpoS regulates a novel type of plasmid DNA transfer in Escherichia coli. PLoS One, 2012, 7(3):e33514. |
 2016, Vol. 56
2016, Vol. 56





