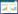中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 唐婧, 徐小蓉, 商传禹, 牛晓娟, 张习敏, 乙引. 2015
- Jing Tang, Xiaorong Xu, Chuanyu Shang, Xiaojuan Niu, Ximin Zhang, Yin Yi. 2015
- 南明河城区河段细菌多样性与环境因子的关系
- Association of bacterial diversity in city area of Nanming river with environmental factors
- 微生物学报, 2015, 55(8): 1050-1059
- Acta Microbiologica Sinica, 2015, 55(8): 1050-1059
-
文章历史
- 收稿日期:2014-12-01
- 修回日期:2015-02-01
目前,我国绝大部分地表水体的水质污染越来越严重,很多重要的城市供水水源河流受日益扩增城市化所带来的大量城市污水的影响,水质明显下降。现今对河流水体的治理包括物理法、化学法和生物修复[1]。生物修复具有环保、经济、无二次污染等优势,有利于改善水体生态系统,恢复水体质量。但是水体中本身含有丰富的微生物群,在实际进行生物修复过程中,微生物群会竞争、抑制微生物复合菌的生长及沉水植物根际微生态系统的形成,从而极大影响生物修复的效率。因此,分析了解河流的细菌群落结构对于开展河流的生物修复具有不可替代的重要作用。
水体生态系统中的细菌群落对降解有机物和营养物循环过程具有重要作用,同时其对外部环境十分敏感[2, 3]。目前,越来越多对于细菌多样性及其与环境因子的关系的研究[4],然而大部分集中在滨海地区、海洋和湖泊[5, 6],对于河流的研究较少,而城市黑河现象却日益严重,人们越来越关注城市河流问题。同时,近年来越来越多微生物多样性的研究由传统的培养研究转向以免培养的宏基因组学研究为主。例如:利用DGGE、稳定性同位素探测、基因芯片和高通量测序等方法研究人体、土壤与海洋内微生物多样性,且取得大量成果[7]。而较少有报道利用高通量测序研究水体特别是城市河流水体微生物多样性的研究。
南明河主要发源于贵州省平坝县林卡冷水冲,是贵阳市的母亲河,贯穿贵阳城区,位于城区的上流河段是花溪、小河和主城区的工业、生活与环境用水的主要用水源。由于城市污水排放等使南明河污染日趋严重[8],市政府对其进行多次治理,但是效果十分有限。基于水体生物修复中微生物群落结构的重要地位以及高通量测序分析微生物多样性的快速发展,本研究提取南明河河水中微生物总DNA,构建16S rDNA V4区片段PCR文库,双末端高通量测序分析南明河水细菌多样性,利用冗余分析(Redundancy analysis,RDA)对其与环境因子的关系进行分析,为了解城市河流的微生物多样性提供重要依据,为进一步城市河流生物修复奠定基础。
1 材料和方法 1.1 样品采样和处理2012年11月为讨论南明河贵阳城区段干流微生物群落特征,选取干流的5个采样点(如图1所示)进行采样,分别是花溪大桥采样点(上游)、五眼桥采样点(上中游)、冠洲桥采样点(中游)、水口寺采样点(中下游)和乌当桥采样点(下游)。上游采样点为南明河流入贵阳市市区的位置,下游采样点为南明河流经贵阳市主城区后流出的位置。每个采样点分上、中、下3个垂直层次,由于河水深度变化较大,上层采集深度(距离河水水面高度)为0.5 m、中层3-5 m、下层5-10 m左右,然后将3个层次水混合,每个采样点重复采样3次。1 L水样经过0.22 μm微孔滤膜过滤,-80℃保存。

|
| 图1. 南明河采样点 Figure 1. Location of sampling site in naming river. |
按文献[9]的方法适当修改后提取宏基因组DNA。将滤膜剪碎,然后加入3 mL抽提缓冲液(1% CTAB、2% PVPP、1.5 mol/L NaCl、100 mmol/L Tris-EDTA、0.1 mol/L磷酸钠缓冲液,pH8.0),加入600 μL 10% SDS,60℃振荡水浴2 h,液氮速冻5 min,80℃水浴5 min,反复冻融3次,8000×g,离心5 min,取上清液,加入等体积的酚-氯仿-异戊醇(体积比25∶24∶1),12000×g,离心10 min,重复1次,加入0.7倍体积的异丙醇-20℃沉淀3 h,12000×g,离心20 min,用70%乙醇1mL漂洗沉淀后室温挥发干燥,沉淀溶于200 μL ddH2O中,基因组DNA用0.8%的琼脂糖凝胶电泳检测。
1.3 细菌16S rDNA PCR扩增检查用引物27F:5′-AGAGTTTGATCCTGGCTCAG-3′和1492R:5′-TACCTTGTTACGACTT-3′,扩增提取宏基因组DNA的 16S rDNA。扩增条件:94℃ 5 min;94℃ 1 min,58℃ 2 min,72℃ 3 min,35 个循环;72℃ 10 min。
1.4 细菌16S rDNA V4区片段扩增及Illumina双末端测序将通过PCR验证的宏基因组DNA然后进行V4区扩增及其的双末端测序。
1.5 水体理化指标测定水质中总磷、总氮、DO、BOD5与COD测定的方法根据中华人民共和国国家环境保护标准中规定的方法进行测定[10],分光光度计测定浊度,铅隔汞砷测定根据GB/T 15337-2008 原子吸收光谱分析法通则。
1.6 细菌多样性数据分析使用QIIME[11]软件对测序数据进行过滤,然后通过flash[12]软件将有overlap的一对reads进行拼接。用uclust[13]软件对序列进行聚类,将97%相似性的序列聚类成为OTUs (Operational Taxonomic Units)。按众数原则对序列进行注释,即如果70%的比对结果都支持同一物种分类,则认为是属于该物种分类,否则则向上一个物种分类等级比较分析,用RDP classifier[14]对序列进行物种注释。统计每个样品在各分类水平上的构成,用柱状图进行可视化。统计每个样品中在属分类水平上的菌群做聚类(Cluster),用颜色表示该属类所含的OTU序列数目水平的相对高低。利用OTUs的数值及比对注释种类到属水平上计算各个采样点的香农-威纳指数(Shannon Wiener index)分析比较细菌群落多样性。
1.7 统计分析利用SPSS和Excel分析计算水体中指标参数。利用Canoco对水体中各个测定理化指标与环境样点进行主成份分析(PCA),同时对环境中菌群矩阵进行除趋势对应分析(DCA),结果显示最大梯度长度小于3,采用冗余分析(RDA)水体细菌群落结构和水体环境因子之间的关系。
2 结果和分析 2.1 水体理化性质统计南明河贵阳城区段5个采样点水体环境理化因子数据如表1。根据《地表水环境质量标准》[15]的规定,南明河河水从花溪大桥流经主城区至乌当桥后浊度明显增大,DO和COD指标从Ⅰ类指标迅速下降到Ⅳ类以下,水质明显下降。水中汞含量较高,达到国家Ⅲ类指标。同时总氮、总磷含量远远高于标准(湖泊)中提出的规定,利用t检验分析水体环境因子在水体流动过程各个点变化的显著性。如表1结果表明,水体流经水口寺到乌当桥后水质变化非常显著。此外,水体从花溪流进市区到五眼桥段水质变化也比较显著。针对结果实地考察后,发现水口寺到乌当桥流段有两个污水处理厂排污口,很有可能是污水处理厂排泄出来的水较大影响了河水水质。
| Sampling site | DO mg/L | BOD5 mg/L | COD mg/L | TN mg/L | TP mg/L | Li mg/L | Cd mg/L | Hg mg/L | As mg/L | Turbidity coefficient |
| **represented very significant difference(P≤0.01), * represented significant difference(0.01≤P≤0.05) | ||||||||||
| HuaXi bridge | 6.75± 0.53 | 8.33± 1.53 | 8.51± 0.79 | 7.63± 0.65 | 0.41± 0.01 | 0.0071± 0.0006 | 0.00012 | 0.00024 | 0.0022± 0.0003 | 1.70± 0.47 |
| WuYan bridge | 4.32± 0.83** | 13.33± 1.53** | 18.35± 1.60 | 8.11± 0.20** | 0.44± 0.01* | 0.0070± 0.0009 | 0.00016* | 0.00027 | 0.0027± 0.0007 | 3.36± 0.44** |
| GuanZhou bridge | 4.36± 0.89 | 10.67± 1.15* | 15.43± 1.80 | 11.48± 0.96 | 0.42± 0.05 | 0.0071± 0.0003 | 0.00016 | 0.00024 | 0.0031± 0.0003 | 4.51± 0.52* |
| ShuiKou Si | 3.81± 0.67 | 10.33± 1.15 | 16.00± 1.99 | 5.84± 0.06 | 0.3± 0.02* | 0.0058± 0.0002** | 0.00015 | 0.00025 | 0.0034± 0.0006 | 5.92± 1.33 |
| WuDang bridge | 0.58± 0.42** | 20.67± 1.53** | 28.63± 2.02 | 12.77± 0.25** | 0.76± 0.03** | 0.0069± 0.0020 | 0.00022** | 0.00028* | 0.0029± 0.0003 | 16.17± 1.91** |
环境因子的主成分分析(PCA)结果显示(图2),总氮和总磷之间显著正相关,PCA前两轴累积贡献率为95.99%,其中第一主轴为90.87%,综合大多数信息。根据测定的环境因子数据比较5个水体样品,花溪大桥采样点、五眼桥采样点和冠洲桥采样点的环境因子相近度明显高于后面两个采样点,同时距离上游采样点——花溪大桥越远,其水质中的环境因子整体差异越大。
|
| 图2. 采样点与环境理化因子的主成分分析图 Figure 2. PCA ordination graph for the study site of patterns and environmental factors. |
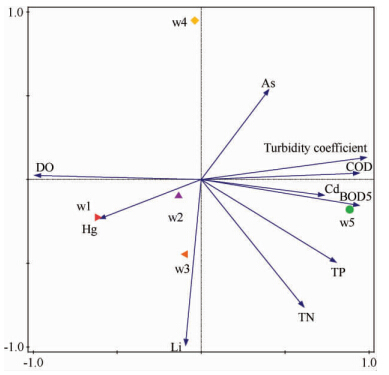
提取5个采样点水体微生物宏基因组DNA,对其进行16S rDNA扩增,然后进行琼脂糖凝胶电泳分析(图3)。在15 kb上和标准品等高处有清晰条带,获得完整质量较好的宏基因组DNA;以其为模版扩增得到1.5 kb左右大小的完整16S rDNA条带,说明提取获得的微生物宏基因组DNA能满足后续的扩增建库实验。
|
| 图3. 五个采样点水体微生物宏基因组DNA(A)及其PCR 16S rDNA(B)电泳图 Figure 3. Metagenomices of 5 sampling site and the PCR fragment of the 16S rDNA. C control sample 5 μL(10 ng/μL); w1 the sampling site in Huaxi bridge; w2 the sampling site in Wuyan bridge; w3 the sampling site in Guanzhou bridge; w4 the sampling site in Shuikou Si; w5 the sampling site in Wudang bridge.M,marker. |
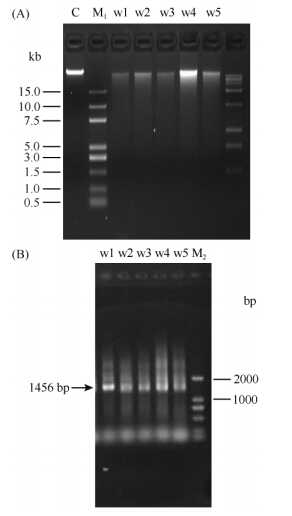
5个样品的细菌16S rDNA V4区的Illumina测序共得到89.7 Mb的原始数据,然后对原始数据拼接,按相似度97%进行聚类分析得到22446个OTU。利用香农-威纳指数比较分析5个样点中的细菌多样性,结果南明河贵阳区段细菌群落多样性多样性指数平均达7.5,乌当桥采样点的细菌多样性>水口寺采样点>五眼桥采样>花溪大桥采样点>冠洲桥采样点,其中五眼桥采样点细菌多样性和花溪大桥采样点细菌多样性十分接近。按照众数原则利用RDP对22446个OTU进行注释,然后统计其在门类别上的构成形成柱状图(图4),同时分析在各个水平上的菌群结构。结果显示这22446个OTU主要属于11个门类,分别是变形菌门(Proteobacterice,66.1%±3.30%)、浮霉菌门(Planctomycetes,12.9%±3.62%)、拟杆菌门(Bacteroidetes,11.2%±2.55%)、酸杆菌门(Acidobacteria,1.2%±0.34%)、放线菌门(Actinobacteria,2.6%±0.75%)、绿菌门(Chlorobi,1.5%±1.19%)、绿弯菌门(Chloroflexi,1.0%±0.24%)、泉古菌门(Crenarchaeota,0.2%±0.21%)、厚壁菌门(Firmicutes,1.9%±0.53%)、芽单胞菌门(Gemmatimonadetes,0.5%±0.24%)、疣微菌门(Verrucomicrobia,1.1%±0.34%),以及还有一部分分类地位不明确的细菌类群(1.6%±0.18%)和稀有细菌类别(1.28%±0.16%)。优势菌门变形菌门包含δ-变形菌纲(Deltaproteobacteria,0.92%±0.16%)、γ-变形菌纲(Gammaproteobacteria,54.76%±4.86%)、β-变形菌纲(Betaproteobacteria,4.54%±0.58)、α-变形菌纲(Alphaproteobacteria,5.62%±1.43%)以及0.3%的变形菌门内分类不明确菌群和极少量的ε-变形菌纲菌群(Epsilonproteobacteria),其中主要是γ-变形菌纲菌群。γ-变形菌纲菌群主要集中于假单胞菌目(Pseudomonadales,20.76%±2.79%)和黄色单胞菌目(Xanthomonadales,12.62%±2.39%)。五个水体样品中菌群变化主要集中在浮霉菌门和拟杆菌门。水体细菌菌群在属水平上包含327个属的细菌,在科水平上覆盖228个科的细菌,其中优势菌属为假单胞菌属(Pseudomonas,16.92%±0.02%)、寡养单胞菌属(Stenotrophomonas,9.23%±0.03%)、金黄杆菌属(Chryseobacterium,3.1%±0.01%)、希瓦氏菌属(Shewanella,2.64%±0.02%)、鞘氨醇杆菌属(Sphingobacterium,2.28%±0.015%)、叶杆菌属(Phyllobacterium,1.5%±0.01%)和地杆菌属(Pedobacter,1.34%±0.01%),同时含有在各个科下属分类地位不明确的菌属。
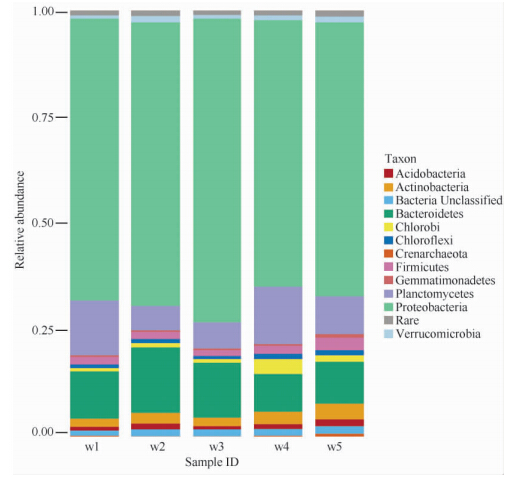
|
| 图4. 5个采样点细菌菌落结构分布图(分类水平按门) Figure 4. the bacterial community during the water of the 5 sampling site. w1 the sampling site in Huaxi bridge; w2 the sampling site in Wuyan bridge; w3 the sampling site in Guanzhou bridge; w4 the sampling site in Shuikou Si; w5 the sampling site in Wudang bridge. |
基于每个样点中物种的丰度水平,筛选主要菌群进行菌群分类地位聚类(横向聚类)及样本聚类(纵向聚类),利用不同颜色表示样点中主要菌群的相对丰度水平(图5)。结果表明,在五个采样点中w2五眼桥采样与和w3冠洲桥采样点菌群群落组成上最为相近,其次是与w4水口寺采样点;而与w5乌当桥采样点的菌落组成上差异最大。我们在对五个采样点的环境因子检测时发现乌当桥采样点的水质变化最大,这些改变有可能影响了水体细菌群落结构的变化。比较各个采样点菌群结构在花溪大桥采样点水体中农杆菌属(Agrobacterium)、微小杆菌属(Exiguobacterium)、不动杆菌属(Acinetobacter)和黄杆菌属(Flavobacterium)菌群明显较高,进入城区采样点后金黄杆菌属(Chryseobacterium)、Candidatus Solibacter、丰佑菌属(Opitutus)和寡养单胞菌属(Stenotrophomonas)菌群出现不同程度增加,最后流经城区到达乌当桥采样点时,细菌菌群最丰富,乳酸杆菌属(Lactobacillus)、Microvirgula、Kaistobacter、Gillisia、拟杆菌属(Bacteroides)、Candidatus Nitrososphaera和芽孢杆菌属(Bacillus)菌群明显增高。通过菌群的分类地位聚类,根据菌群的进化分支,我们将主要菌群划分为5簇(Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ),如图5。第Ⅲ簇内的菌属在花溪大桥采样点的丰度都相对较高,而Ⅳ簇内的菌属在乌当桥采样点明显增高。

|
| 图5. 5个采样点主要细菌菌落聚类 Figure 5. The cluster analysis of the bacterial community during 5 sampling site. w1 the sampling site in Huaxi bridge; w2 the sampling site in Wuyan bridge; w3 the sampling site in Guanzhou bridge; w4 the sampling site in Shuikou Si; w5 the sampling site in Wudang bridge. |
用RDA分析水体环境因子与细菌主要菌落之间的关系(图6),图中直接看出物种群落分布与环境因子之间的关系,结果表明芽孢杆菌属(Bacillus)、罗思河小杆菌属(Rhodanobacter)、Elizabethkingi、Wautersiella和希瓦氏菌属(Shewanella)5个属菌群丰度与砷、COD、镉、BOD、浊度显著正相关,而与汞、DO显著负相关。黄杆菌属(Flavobacterium)、地杆菌属(Pedobacter)、不动杆菌属(Acinetobacter)、鞘氨醇单胞菌(Sphingomonas)、出芽菌属(Gemmata)和苍白杆菌属(Ochrobactrum)6个属菌群丰度与汞、铅显著正相关。第Ⅳ簇菌群内的乳酸杆菌属(Lactobaillus)、Gillisia、Kaistobacter、DA101、Microvirgula、Candidatus_Nitrosophaera和拟杆菌属(Bacteroides)等7个菌属丰度和总氮、总磷显著正相关,而硝化螺菌属(Nitrospira)、A4、鞘氨醇杆菌属(Sphingobacterium)3个属菌群和总氮、总磷显著负相关。
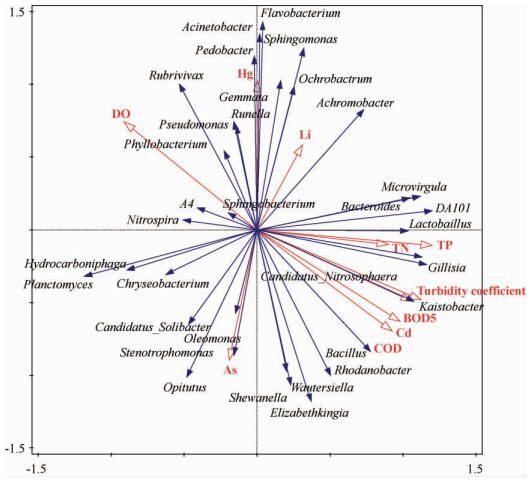
|
| 图6. 细菌群落与环境因子的RDA图 Figure 6. RDA ordination graph for the bacterial community and environmental factors. |
多样性指数是反映群落结构特征的重要指标,对于南明河贵阳区段细菌群落多样性分析结果表明,其多样性指数较高平均接近7.5,远远高于利用DGGE、16S rDNA文库分析,RFLP分析等方法对于细菌群落多样性的研究[16, 19],说明通过高通量测序16S rDNA V4区研究能较好的研究分析水体细菌群落多样性。本研究通过针对南明河河水中细菌16S rDNA V4区高通量测序,结果分析发现,河水中细菌除了部分分类地位不明确的菌群和稀有菌群外包含327个属的细菌,分布于228个科11个门内,河水中细菌菌群极其丰富。目前研究报道[20, 21, 22]的不同水体系统研究发现共出现过22个门类的细菌菌群,分别是变形菌门、蓝细菌门(Cyanobacteria)、拟杆菌门、放线菌门、疣微菌门、酸杆菌门、绿菌门、绿弯菌门、纤维杆菌门(Fibrobacteres)、厚壁菌门、梭杆菌门(Fusobacteria)、芽单胞菌门、黏胶球形菌门(Lentisphaerae)、硝化螺菌门、浮霉菌门、螺旋体门(Spirochaetes)、脱铁杆菌门(Deferribacteres)、BRC1、OD1、OP10、SR1和TM7,在本研究中发现在南明河水中还存在古细菌中的泉古菌门菌群。
目前对细菌多样性分布的机制有两大类,一个是Beijerinch[23]提出的微生物具有全球性的分布特征,但是只有环境条件合适,微生物才能够在新生境中定居成功和大量繁衍。另外一个是与Beijerinch所提出的细菌全球性分布的观点相对应的是meta-群落假说[24, 25]。该假说包含4个观点,其认为细菌多样性是因扩散作用而联系在一起的一系列本地群落的集合,它强调不同时空尺度(本地、区域)上群落的结构和动态过程。在meta-群落假说中,细菌群落组成有两个影响因子:内部环境因子和外部环境因子。内部环境因子指食物网中生物间的相互作用及物种的随机动态等物种本身之间的影响因子,外部环境因子指环境温度、主要离子浓度等外在因素。目前认为在静置水里停留时间长的湖泊中细菌群落主要受湖泊内部环境因子的影响,而相对的在流动湖泊中则认为细菌多样性是内部环境因子和不同生境间随机分布共同作用的影响[25]。本研究中随着贵阳南明河河水流动的过程中,河水的外部环境因子出现改变,通过主要菌群与外部环境因子的RDA分析发现,不同环境因子对于不同菌群具有不同影响,离子砷、镉含量提高促生芽孢杆菌属、罗思河小杆菌属等5个菌属细菌,而汞却会抑制其的丰度。汞、铅含量增加显著提高黄杆菌属、地杆菌属等6个菌属的菌群丰度。水体富营养化,总磷、总氮含量的提高,促进乳酸杆菌属、拟杆菌属等7个菌群丰度的提高,而硝化螺菌属、鞘氨醇杆菌属3个属菌群的丰度提高会降低总氮、总磷含量。在城市河流细菌多样性的分布中,外部环境因子随着河流进去城区后的流动变化显著,外部环境因子极大的影响了河流细菌多样性。
城市河流在流经城区的过程中,细菌多样性会随着环境因子的影响出现不同的变化。本研究发现南明河流入城区前花溪大桥处细菌多样性指数较低,而经过城区流出的乌当桥采样点细菌多样性指数明显高于花溪大桥处。不同程度的人类活动对于河流细菌群落结构具有不同的影响。在进入城区前,花溪大桥采样点中,第Ⅲ簇内细菌菌属丰度较高。在花溪南明河段存在大量沉水植物,河流主要流经区域两旁主要是农田,农民释放一定农用抗生素,存在部分的残留流入水体中,这些环境因素极大影响了这一个河段的水体细菌群落结构。目前研究发现,农杆菌属菌群生存在植物根的表面,依靠由根组织渗透出来的营养物质。黄杆菌属和不动杆菌属细菌广泛存在于水体、动物体和土壤等环境中,两个种属细菌大多容易对抗生素等具有抗药性。因此第Ⅲ簇内的农杆菌属、微小杆菌属、黄杆菌属和不动杆菌属等菌属对水体环境表现较好的适应性,其菌群丰度相对较高。在流出城区的乌当桥采样点,水体的总氮、总磷含量显著增高,第Ⅳ簇内的菌属表现出明显的丰度增高现象。用RDA分析菌属与环境因子相关性发现,第Ⅳ簇内的乳酸杆菌属(Lactobaillus)、Gillisia、Kaistobacter、DA101、Microvirgula、Candidatus_Nitrosophaera和拟杆菌属(Bacteroides)等7个菌属和总氮、总磷含量显著正相关。目前研究报道乳酸杆菌属、Microvirgula、Kaistobacter、Gillisia、拟杆菌属、Candidatus Nitrososphaera和芽孢杆菌属的细菌和糖代谢、氮代谢、脂化酶、污水处理等密切有关,对于氮和磷表现良好的适应性,因此经过城区河段,由于城区污水的排放,增高河水中氮磷含量,导致乳酸杆菌属、Microvirgula、Kaistobacter、Gillisia、拟杆菌属、Candidatus Nitrososphaera和芽孢杆菌属细菌丰度明显增高。城市河流流入城区前,河水中含氮磷量较低,水质清澈,导致在这样的生境处微生物生长受到营养的限制,细菌多样性较低,而经过城区河段,由于城区污水的排放,增高河水中氮磷含量,导致细菌多样性增高,且细菌群落结构发生变化。而临近医院的冠洲桥采样点,由于医院的排放污水影响了细菌的生长,导致了该处细菌多样性的下降。
通过对城区河流细菌多样性的不同采样点研究发现,城区河流受人类生活影响巨大,其河流流段在不同采样点会出现较湖泊等复杂的环境因数,因此为揭示城市河流的细菌种群类别与环境因子的相关性,我们后续还需进一步对于城市河流细菌多样性进行更多的调查,最终为城市河流生物修复治理提供更为重要的依据。
| [1] | Wang YC, Liu Y, Hao Z, Zhang L, Shen Y, Li G, Li D, Chen D, Ma Q, Yang H. Comparing and the control effect of several black and stinking rivers in Shanghai. Acta Hydrobiologica Sinica, 2009, 33(2): 355-359. (in Chinese) 王英才,刘永定,郝宗杰,张兰,沈银武,李根保,李敦海,陈德辉,马庆,杨洪芳.上海市几条黑臭河道治理效果的比较与分析.水生生物学报,2009,33(2):355-359. |
| [2] | Stuart Findlay. Stream microbial ecology. Journal of the North American Benthological Society, 2010, 29 (1): 170-181. |
| [3] | Jiang J, Shen Y. Development of the microbial communities in Lake Donghu in relation to water quality. Environ Monit Assess, 2007, 127(1): 227-236. |
| [4] | Laque T, Farjalla VF, Rodado AS, Esteves FA. Spatiotemporal variation of bacterial community composition and possible controlling factors in tropical shallow lagoons. Microbial Ecology, 2010, 59 (4): 819-829 |
| [5] | del Giorgio PA, Cole JJ. Bacterial growth efficiency in natural aquatic systems. Annual Review Ecology and Systematics, 1998, 29: 503-541. |
| [6] | Xu P, Leff LG. Longitudinal changes in the benthic bacterial community of the Mahoning River (Ohio, U.S.A.). Hydrobiologia, 2004, 522 (1): 329-335. |
| [7] | Guo L. Progress of microbial species diversity research in China. Biodiversity Science, 2012, 20(5): 572-580. (in Chinese) 郭良栋. 中国微生物物种多样性研究进展. 生物多样性,2012,20(5):572-580. |
| [8] | Xie C, Zhou J, Zhang H. A preliminary investigation on water quality in different sections of urban segment of Nanming River in Guiyang city. Journal of Guiyang Medical College, 2011, 36(2): 165-167 (in Chinese) 谢春,周婕,张华. 南明河贵阳城区段不同面水质分析. 贵阳医学院学报,2011,36(2):165-167. |
| [9] | Verma d, Satyanarayana T. An improved protocol for DNA extraction from alkaline soil and sediment samples for constructing metagenomic libraries. Applied Biochemistry and Biotechnology, 2011, 165(2): 454-464. |
| [10] | 国家环境保护部. 1989,GB 11893-89,GB11914-89,GB11894-89.中华人民共和国国家环境保护标准. 北京:中国国家环境出版社. |
| [11] | J Gregory C, Justin K, Jesse S, Kyle B, Frederic DB, Elizabeth K C, Noah F, Antonio GP, Julia KG, Jeffrey IG, Gavin AH, Scott TK, Dan K, Jeremy EK, Ruth EL, Catherine AL, Daniel M, Brian DM, Meg P, Jens R, Joel RS, Peter JT, William AW, Jeremy W, Tanya Y, Jesse Z, Rob K. QIIME allows analysis of high-throughput community sequencing data. Nature Method, 2010, 7(5): 335-336. |
| [12] | Mago Dč T, Salzberg SL. FLASH: fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21): 2957-2963. |
| [13] | Edgar RC. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461. |
| [14] | Wang Q, Garrity MG, Tiedje MJ, Cole RJ, Naïve. Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. |
| [15] | 国家环保部.2002, GB3838-2002. 地表水环境质量标准.北京:中国国家环境出版社. |
| [16] | Zhang W, Hu YG, Huang GH, Gao HW. Soil microbial diversity of artificial peash rub plantation on North Loess Plateau of China. Acta Microbiologic Sinica, 2007, 47(5): 751-756 (in Chinese) 张薇,胡跃高,高洪文. 西北黄土高原柠条种植区土壤微生物多样性分析. 微生物学报,2007,47(5):751-756. |
| [17] | Pang X, Lu L, Niu Y, Shen H, Yuan X, Chen W, Chen J, Xie P. The spatial pattern of bacterioplankton community composition in lake Taihu in summer. Acta Hydrobiologica Sinica, 2014, 38(2): 335-341. (in Chinese) 庞兴红,吕丽媛,牛远,沈宏,袁希功,陈文捷,陈隽,谢平. 太湖夏季浮游细菌群落多样性的空间格局. 水生生物学报,2014,38(2):335-341. |
| [18] | Liu W, Mao Z, Yang Y, Xie B. Analysis of soil bacterial diversity by using the 16S rRNA gene library. Acta Microbiologic Sinica, 2008, 48(10): 1344-1350.(in Chinese) 刘玮琦,茆振川,杨宇红,谢丙炎.应用16SrRNA基因文库技术分析土壤细菌群落的多样性. 微生物学报,2008,48(10):1344-1350. |
| [19] | Yu Y, Wang X, Zhang P. Spatial distribution of planktonic bacterial community and its relationship to water quality in Beiyun River. Asian Journal of Ecotoxicology, 2012, 7(3): 337-344. (in Chinese) 于洋,王晓燕,张鹏飞.北运河水体浮游细菌群落的空间分布特征及其与水质的关系. 生态毒理学报,2012,7(3):337-344. |
| [20] | Zwart G, Crump BC, Kamst-van Agterveld MP, Hagen F, Han SK. Typical fresh water bacteria: an analysis of available 16S rRNA gene sequences from plankton of lakes and rivers. Aquatic Microbial Ecology, 2002, 28(2): 141-155. |
| [21] | Newton RJ, Jones SE, Eiler A, Mc Mahon KD, Bertilsson S. A guide to the natural history of freshwater lake bacteria. Microbiology and Molecular Biology Reviews, 2011,75(1):14-49. |
| [22] | Dong Y, Zhao Y, Zhang W, Li Y, Zhou F, Liu C, Wu Y, Liu S, Zhang W, Xiao T. Bacterial diversity and community structure in the East China Sea by 454 sequencing of the 16S rRNA gene. Chinese Journal of Oceanology and Limnology, 2014,32(3): 527-541. |
| [23] | Ren L, He D, Xing P, Wang Y, Wu Q. Bacterial diversity and ecological function in lake water bodies. Biodiversity Science, 2013, 21(4): 421-432. 任丽娟,何聃,邢鹏,王毓菁,吴庆龙. 湖泊水体细菌多样性及其生态功能研究进展. 生物多样性,2013,21(4):421-432. |
| [24] | Leibold MA, Holyoak M, Mouquet N, Amarasekare P, Chase JM, Hoopes MF, Holt RD, Shurin JB, Law R, Tilman D, Loreau M, Gonzalez A. The metacommunity concept: a framework for multi-scale community ecology. Ecology Letters, 2004, 7(7): 601-613. |
| [25] | Brendan Logue J, Lindström E S. Biogeography of bacterioplankton in inland waters. Fresh water Reviews, 2008, 1(1): 99-114. |
 2015, Vol. 55
2015, Vol. 55