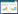中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 李璐, 郝春博, 王丽华, 裴理鑫. 2015
- Lu Li, Chunbo Hao, Lihua Wang, Lixin Pei. 2015
- 巴丹吉林沙漠盐湖微生物多样性
- Microbial diversity of salt lakes in Badain Jaran desert
- 微生物学报, 2015, 55(4): 412-424
- Acta Microbiologica Sinica, 2015, 55(4): 412-424
-
文章历史
- 收稿日期:2014-08-18
- 修回日期:2014-11-14
巴丹吉林沙漠(Badain Jaran Desert),位于我国内蒙古自治区阿拉善高原的西北部(39° 20′ N-41° 30′N,100° E-104° E),面积49000 km2[1],是我国第三大沙漠。巴丹吉林沙漠属极端大陆性气候,其西南部位于东亚季风的北部边缘,东亚季风提供了降水的主要来源,70%的降雨发生在7至9月,每年有10-35 d降雨期。该地区的平均年降雨量为150 mm,而蒸发量却高达1500 mm。
自然界中的盐湖有相当一部分伴有高碱特性,称为碱性盐湖,又称苏打盐湖,其一般形成于气候较干的内陆地区。水分的蒸发使Na元素和K元素迅速聚集,而在Ca和Mg元素含量较低的情况下,富含Na元素的岩石被含CO2的地下水浸蚀,碳酸的存在形成了独特且稳定的碱性栖息地[2]。与世界其他大沙漠明显不同的是,在巴丹吉林沙漠的东南部,200-300 m的高大沙丘广布,个别甚至超过400 m;而在这些高大沙丘之间还分布着众多常年性湖泊。据统计,面积在1.5 km2以下的沙漠湖泊有140多个,且多为咸水湖。巴丹吉林沙漠人烟稀少,因此沙漠区湖泊和地下水的化学特征基本不受人类活动的影响[3]。
盐湖环境中栖息着大量的嗜盐微生物,它们种类丰富,在细菌、古细菌和真核生物三域中均有分布。对于嗜盐菌的研究由来已久,嗜盐机理的研究也相对成熟。但是对于嗜盐碱微生物,尤其是嗜盐碱古菌的研究甚少,对于嗜盐碱菌在高盐碱环境下的双重适应机制的研究微乎其微,是有待于进一步开发研究的领域。据我们所知,对于巴丹吉林沙漠苏打盐湖中嗜盐碱微生物多样性及群落结构的研究还未见报道。
本研究采集了巴丹吉林沙漠中苏打盐湖的湖水样品,采用分子生物学技术系统研究了不同样品中微生物的种群结构及分布规律。研究结果不但拓展了我们对高盐碱环境中嗜盐微生物多样性的认识,还有利于了解极端环境中微生物的适应机制和生存状态,为进一步开发嗜盐碱微生物资源提供理论基础和技术支持。
1 材料和方法 1.1 样品采集本研究中的盐湖是位于内蒙古巴丹吉林沙漠中的小沙枣海子(1号湖)、散根吉林南湖(2号湖)以及中淖尔图(3号湖),采样时间为2013年9月。1号湖较小,因此只采1份水样。2、3号湖较大,因此在平缓的顺风坡和较为陡峭的逆风坡各采1份水样,分别记为2-1、2-2和3-1、3-2。采样点距离湖边约0.5 m,所采水样均为表层水。水样采集后储存于4℃保温箱中,尽快返回实验室进行操作。
1.2 物化参数的测定及分析水样温度在现场用哈纳水质测定仪测定。样品带回实验室后,立即进行pH、矿化度、碳酸根、重碳酸根、氨氮、硝氮、总磷等参数的测定[4]。离子浓度用美国Thermo公司ICS-600离子色谱仪测定,TIC、TOC用美国OI公司1030W型TOC分析仪测定。微生物丰度(microbial abundance,MA)采用吖啶橙染色直接计数法(acridine orange direct count,AODC)测定。
用AqQA软件对水样阴阳离子进行水化学分析,确定盐湖水质类型。
1.3 基因组DNA的提取及16S rRNA基因基因片段的扩增水样于0.22 μm细菌滤膜上进行真空抽滤,将滤膜剪碎放入DNA试剂盒,按生产商提供的步骤提取基因组DNA,将提取的总DNA保存于-80℃冰箱。细菌使用16S rRNA基因通用引物27F(5′-AGAGTTTGATCMTGGCTCAG-3′)和1492R(5′-TACGGYTACCTTGTTACGACT T-3′)进行目的基因扩增。PCR反应条件为: 95℃ 5 min,94℃ 45 s,52 45 s,72℃ 90 s,共35个循环,最后在72℃下延伸 5 min。古菌使用16S rRNA基因通用引物ARC21F(5′-TTCYGGTTGATCCYGCCRGA-3′)和ARC958R(5′-YCCGGCGTTGAMTCCAWTT-3′)进行目的基因扩增,PCR反应条件为: 95℃预变性5 min,94℃变性40 s,48℃退火40 s,72℃延伸90 s,共35个循环,最后在72℃下延伸 2 min。反应产物用1%琼脂糖凝胶电泳检测。
1.4 克隆文库的构建及测序PCR产物连接到pGEM®-T载体上,将连接好的环状质粒转化到大肠杆菌感受态细胞内,建立细菌和古菌样品的克隆文库,并进行蓝白斑筛选,利用载体特异性引物对M13M4(5′-GTTTTCCCAG TCACGAC-3′)和M13RV(5′-CAGGAAACAG CTATGAC-3′)进行PCR扩增筛选插入片段。最后将鉴定出的阳性克隆送至测序公司测序。
1.5 类群比例分析及聚类分析获得序列后,首先用Bellerophon程序检测是否有嵌合体存在,剔除存在的嵌合体,并除去低质量序列和较短的序列。将筛选得到的高质量序列经过RDP中的Classifier程序进行分类,得出每个样品中的所有序列在各个门类中所占的比例,据此绘制类群比例条形图。
通过Mothur软件中的make.group命令生成groups格式文件;dist.seqs命令生成距离矩阵;cluster命令进行聚类分析;bin.seqs命令输出OTU;get.oturep命令得到代表性OTU,并计算OTU个数及OTU所属样品;make.shared命令生成OTU table;classify.seqs命令将序列与RDP上的高质量序列比对,得出比对结果。序列比对同源性大于97%的认为属于同一OTU。
基于Mothur做出的OTU table,经过一系列矩阵格式的转化之后,利用R语言gplot程序包中的heatmap.2()函数绘制热图,所选参数均为默认参数,OTU中的克隆数经过ln(x+1)变换;利用R语言VennDiagram程序包中的draw.quintuple.venn()函数绘制韦恩图。采用Canoco for Windows 4.5软件对微生物群落和环境因子进行典范对应分析(CCA)。
1.6 系统发育分析基于Mothur软件生成的代表性序列,将所得代表性序列输入NCBI网站,用Blast程序与GenBank中已有的序列进行比对,下载相似性最高的序列和相似性较高的已知种的序列作为参考。将所有序列用BioEdit中的ClustalW程序比对后,用MEGA 6.0软件绘制系统发育树,bootstrap重复次数为1000,距离系数细菌为0.02,古菌为0.01。
1.7 序列登录号本研究所得序列均已提交GenBank中,序列登录号为KJ733746-KJ733768(古菌)和KJ733769-KJ733838(细菌)。
2 结果和分析 2.1 样品的物理化学参数所采样品的物理化学参数见表1。1号湖水样透明,可见少量卤虫;2号湖水样呈红色,卤虫数量较多,采样瓶中有盐结晶析出;3号湖水样几乎透明,可见少量绿色藻类。经分析测试发现,所有样品均呈强碱性,pH值都在10以上。其中,1号湖的碱性最强,pH值高达10.83。同时,几个湖的矿化度都很高,均在165 g/L以上,2号湖更是高达343.82-397.33 g/L。Na+是最主要的金属离子,在2号湖中高达124.8-166.08 g/L;在1号和3号湖中略低,为59.93-78.96 g/L。K+是另一种主要的阳离子,在2号湖中的含量为30.22-49.81 g/L;在1号和3号湖中则较低,均在5.2 g/L以下。Ca2+在3个湖中的含量普遍较低,都在1 g/L以下。在中性和酸性盐湖中含量较高的Mg2+在本研究样品中未检出。在几种金属离子中,Na+的含量与样品的矿化度成正比。
| Sample | 1 | 2-1 | 2-2 | 3-1 | 3-2 |
| DO:Dissolved Oxygen; TIC:Total Inorganic Carbon; TOC:Total Organic Carbon; TP:Total Phosphorus; MA:Microbial Abundance. | |||||
| T/℃ | 23.92 | 28.73 | 31.79 | 22.37 | 28.19 |
| pH | 10.83 | 10.27 | 10.18 | 10.40 | 10.40 |
| DO(mg/L) | 3.50 | 0.25 | 0.22 | 3.60 | 1.90 |
| Salinity /(g/L) | 172.76 | 397.33 | 343.82 | 205.21 | 165.48 |
| c(Na)/(g/L) | 78.96 | 166.08 | 124.8 | 83.85 | 59.93 |
| c(K)/(g/L) | 2.20 | 49.81 | 30.22 | 5.13 | 3.44 |
| c(Ca)/(g/L) | 0.15 | 0.62 | 0.62 | 0.62 | 0.29 |
| c(Cl-)/(g/L) | 17.19 | 249.56 | 198.80 | 106.55 | 76.95 |
| c(SO42-)/(g/L) | 23.58 | 49.81 | 27.55 | 21.19 | 13.55 |
| c(CO32-)/(g/L) | 31.67 | 18.32 | 13.97 | 9.27 | 9.55 |
| c(HCO3-)/(g/L) | 16.18 | 1.48 | 3.68 | 6.87 | 4.93 |
| c(TIC)/(g/L) | 15.84 | 8.60 | 5.54 | 4.73 | 4.97 |
| c(TOC)/(mg/L) | 301.33 | 250.94 | 151.02 | 114.27 | 101.10 |
| c(NO3—N)/(mg/L) | 18.21 | 18.51 | 13.77 | 11.24 | 14.68 |
| c(NH4+-N)/(mg/L) | 6.03 | 3.54 | 3.82 | 2.71 | 2.30 |
| c(TP)/(mg/L) | 22.93 | 12.78 | 9.20 | 1.24 | 1.35 |
| MA/(cells/mL) | 6.76×108 | 5.44×108 | 6.46×108 | 4.71×108 | 5.13×108 |
| Type of Lake | Na-CO3 Lake | Na-Cl Lake | Na-Cl Lake | Na-Cl Lake | Na-Cl Lake |
Cl-是最主要的阴离子,它在2号湖中含量最高,达198.8-249.56 g/L;1号湖最低,为17.19 g/L。SO42-同样在2号湖中含量最高,达27.55-49.81 g/L;3号湖最低,为13.55-21.19 g/L。CO32-的分布规律与以上两者有很大不同,它在1号湖中的含量最高,达31.67 g/L;在3号湖中最低,为9.27-9.55 g/L。HCO3-同样在1号湖中浓度最高,达16.18 g/L;在2号湖中最低,仅为1.48-3.68 g/L。相应的,TIC含量在1号湖中也最高,达15.84 g/L。经水化学分析表明,1号湖为Na-CO3型盐湖,2、3号湖为Na-Cl型盐湖。
TOC含量在1号湖中最高,达0.3 g/L,证明它含有较高浓度的有机质。此外,1号湖的微生物丰度也最高,达6.76×108 cells/mL。这可能是因为1号湖的碳氮磷浓度都比较高,营养物质较为丰富。
2.2 生物类群比例分析2.2.1 细菌类群比例分析:将筛选得到的242个高质量细菌序列在RDP数据库内进行相似性比对,发现5个样品的16S rRNA基因序列分别属于γ-变形菌亚门(Gammaproteobacteria)、拟杆菌门(Bacteroidetes)、α-变形菌亚门(Alphaproteobacteria)、厚壁菌门(Firmicutes)、疣微菌门(Verrucomicrobia)、β-变形菌亚门(Betaproteobacteria)、放线菌门(Actinabacteria)、δ-变形菌亚门(Deltaproteobacteria)(图1)。由图可知,样品1、3-1和3-2中的类群分布比较相似,其主导类群都为γ-变形菌亚门,分别占其文库的61.2%、68.0%和71.8%。样品2-1以拟杆菌门和γ-变形菌亚门同时为主导类群,所占比例分别为48.1%和30.9%。样品2-2中以α-变形菌亚门和拟杆菌门为主导类群,占有率分别为35.5%和26.3%。
|
| 图1. 样品中各细菌生物类群在门的水平所占比例 Figure 1. Proportion of bacteria phyla in the samples. |
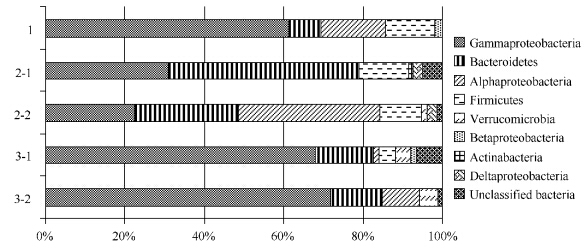
2.2.2 古菌类群比例分析:以同样的方式对古菌的189个有效序列进行相似性比对,发现5个样品的16S rRNA基因基因序列全部属于广古菌门(Euryarchaeota)的盐杆菌科(Halobacteriaceae),主要分布于Halorubrum、Natronomonas、Halonotius、Halohasta以及Halovenus这5个属,同时存在一些未分类的盐杆菌(图2)。由图2可以看出,3个湖中的古菌分布有显著差异:1号湖主导类群并不明显,生物多样性较高,涵盖了OTU分类单元中所有的属;2号湖以Halonotius和Halohasta为主导类群,在样品2-1和2-2中都有43.2%和38.6%的占有率;3号湖以一个未分类的盐杆菌科(Halobacteriaceae)类群为主导,在3-1和3-2样品文库中的比例高达84.6%和84.2%。Halorubrum和Natronomonas属在5个样品中均有分布,但相对较少,所占比例均小于12%。由2号湖和3号湖来看,古菌的分布情况在同一湖中较为相似,但在不同的湖之间差异明显。
|
| 图2. 样品中各古菌生物类群在属的水平所占比例 Figure 2. Proportion of archaea genus in the samples. |
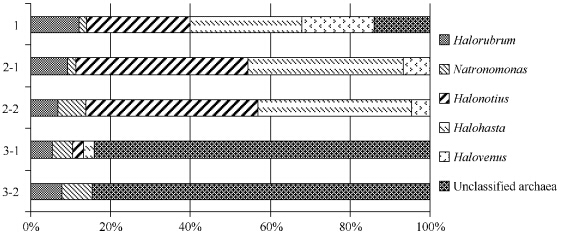
2.3.1 细菌聚类分析:将5个样品的366个有效序列经Mothur软件聚类后,得到75个OTU,根据这些OTU在各样品中的分布情况绘制了如图3-A所示的韦恩图。由图可看出,5个样品没有共同的OTU存在,样品间的差异性较大,多数OTU只分布于其中的一个样品。香浓威尔指数表明,3号湖的生物多样性较高,其次为2号湖和1号湖(见表2),但其中的差异并不是太明显。样品1和3-1、3-2有4个共同的OTU,样品2-1和2-2有4个共同的OTU。2号湖与其它湖的交集较小。
|
| 图3. 5个样品细菌群落的聚类分析 Figure 3. Clustering analysis of bacterial community in 5 samples. A: Venn Diagram. B: Heat Map. |
| Clone library | Total clones | Number of OTUs | Coverage (C)* | Shannon (H) |
| *Coverage(C)=1-(n/N) (n=the number of a clone being only contained in OTUs,N=total sequence number). | ||||
| Bac 1 | 49 | 15 | 0.90 | 2.28 |
| Bac 2-1 | 81 | 18 | 0.86 | 2.10 |
| Bac 2-2 | 76 | 23 | 0.85 | 2.53 |
| Bac 3-1 | 75 | 21 | 0.89 | 2.69 |
| Bac 3-2 | 85 | 25 | 0.86 | 2.43 |
| Arc 1 | 50 | 16 | 0.85 | 2.42 |
| Arc 2-1 | 44 | 9 | 0.91 | 1.64 |
| Arc 2-2 | 44 | 9 | 0.86 | 1.71 |
| Arc 3-1 | 38 | 7 | 0.85 | 0.72 |
| Arc 3-2 | 13 | 3 | 0.85 | 0.54 |
在细菌的75个OTU中,有36个OTU包含两个及以上基因序列,占总克隆数的89.3%。以此36个OTU为基础,取ln(x+1)值绘制了如图3-B所示的细菌群落结构热图。图中,纵向聚类代表样品之间的群落结构差异。横向聚类代表每个OTU在5个样品中的分布情况差异,聚为一簇的分支说明这些OTU在五个样品中的分布情况是接近的。此图只是反映样品间多样性的远近关系,聚为一簇的两种OTU并不一定是同种细菌。由图可以看出,样品1和3-2聚为一簇,它们与3-1聚在一个大分枝上,而样品2-1和2-2聚为单独的一簇,并没有一种基因型在5个样品中同时出现,这与韦恩图所表征的数据是一致的。5个样品间的群落组成差异非常明显,分析其水化学特征发现,2号湖的矿化度很高,几乎达到饱和,而1、3号湖的矿化度相对较低。尤其是样品1和3-2的矿化度,都只在170 g/L左右,说明矿化度是影响细菌群落结构的主要因素。
对细菌群落和环境因子进行典范对应分析(CCA)也表明,矿化度与第一排序轴呈最大正相关,相关系数高达0.9822。DO与第一排序轴呈最大负相关,相关系数为-0.9032,而盐湖中DO又受矿化度的直接影响。一般来说,盐湖的矿化度越大,DO就越低[5]。由此可见,矿化度是这些碱性盐湖中细菌群落结构的控制因素。
2.3.2 古菌聚类分析:将5个样品的189个古菌有效序列经Mothur软件聚类后,得到23个OTU,并绘制了韦恩图和热图。由图4-A可知:1号湖共有16个OTU,而2、3号湖的总OTU数分别只有11个和8个。同时,1号湖有8个独有的OTU,2、3号湖只有1至2个,这意味着1号湖古菌具有更高的多样性。香浓威尔指数(见表2)也表明,1号湖的生物多样性远大于2、3号湖。热图4-B显示,2-1和2-2聚为一簇,然后和1聚为一个大分枝,而3-1和3-2聚为一簇,说明在古菌群落分布上,1号湖与2号湖具有更高的相似性。

|
| 图4. 5个样品古菌群落的聚类分析 Figure 4. Clustering analysis of archaea community in 5 samples. A: Venn Diagram. B: Heat Map. |
对古菌群落和环境因子进行CCA分析发现,氨氮、硝氮、总磷、矿化度、TOC等几个主要的环境因子与第一排序轴的相关系数均在0.62-0.78之间。这说明古菌的分布没有决定性的环境因子,而是由多个物化参数综合影响。
2.4 系统发育学分析2.4.1 细菌文库的系统发育学分析:细菌75个OTU中,有18个为含有4个及以上基因序列的OTU,占总克隆数的77%,将每种基因型的代表序列输入NCBI网站,用BLAST程序与数据库中的已有序列进行比对,主要比对结果见表3。
| Clone ID | Gene frequency/% | Closest cultured bacteria (accession number, similarity) | Phylogenetic classification | ||||
| 1 | 2-1 | 2-2 | 3-1 | 3-2 | |||
| OTU01 | 32.7 | 0 | 2.6 | 22.7 | 21.2 | Halomonas sp. A1 (FJ157163, 99%) | Gammaproteobacteria |
| OTU18 | 0 | 0 | 0 | 8.0 | 0 | Halomonas sp. LYG4C (GU447290, 97%) | Gammaproteobacteria |
| OTU02 | 14.3 | 0 | 0 | 13.3 | 31.8 | Spiribacter salinus strain M19-40 (NR103952, 98%) | Gammaproteobacteria |
| OTU03 | 0 | 28.4 | 14.5 | 0 | 0 | Halorhodospira halophila strain 9622 (FN293051, 97%) | Gammaproteobacteria |
| OTU08 | 0 | 0 | 0 | 10.7 | 2.4 | Halomonadaceae bacterium 20P2 (JX434732, 99%) | Gammaproteobacteria |
| OTU11 | 4.1 | 0 | 0 | 1.3 | 3.5 | Saccharospirillum sp. GCWy1 (KJ195687, 94%) | Gammaproteobacteria |
| OTU13 | 6.1 | 0 | 0 | 0 | 1.2 | Aquisalimonas asiatica strain CG12 (NR042570, 98%) | Gammaproteobacteria |
| OTU05 | 0 | 0 | 32.9 | 0 | 2.4 | Roseinatronobacter monicus (DQ659237, 99%) | Alphaproteobacteria |
| OTU14 | 8.2 | 0 | 1.3 | 0 | 0 | Roseibaca ekhonensis EL-50 (NR042212, 98%) | Alphaproteobacteria |
| OTU07 | 6.1 | 0 | 0 | 1.3 | 5.9 | Paracoccus sp. MBIC4036 (AB025192, 97%) | Alphaproteobacteria |
| OTU04 | 2.0 | 44.4 | 2.6 | 0 | 0 | Salinibacter ruber strain M31 (NR074895, 91%) | Bacteroidetes |
| OTU10 | 0 | 1.2 | 0 | 4.0 | 2.4 | Salinibacter sp. NA9-38 (KF569485, 87%) | Bacteroidetes |
| OTU06 | 4.1 | 0 | 0 | 2.7 | 3.5 | Gracilimonas mengyeensis YIM J14 (NR109748, 91%) | Bacteroidetes |
| OTU09 | 0 | 0 | 14.5 | 0 | 1.2 | Psychroflexus sp. antwl407 (JF811035, 93%) | Bacteroidetes |
| OTU12 | 0 | 4.9 | 2.6 | 0 | 0 | Halarsenatibacter silvermanii (AY965613, 95%) | Firmicutes |
| OTU16 | 0 | 0 | 3.9 | 0 | 0 | Halarsenatibacter silvermanii (AY965613, 93%) | Firmicutes |
| OTU15 | 6.1 | 0 | 0 | 0 | 0 | Piscibacillus sp. 401C1-1 (HM222702, 96%) | Firmicutes |
| OTU17 | 0 | 0 | 0 | 4.0 | 3.5 | Puniceicoccus vermicola (NR043906, 90%) | Verrucomicrobia |
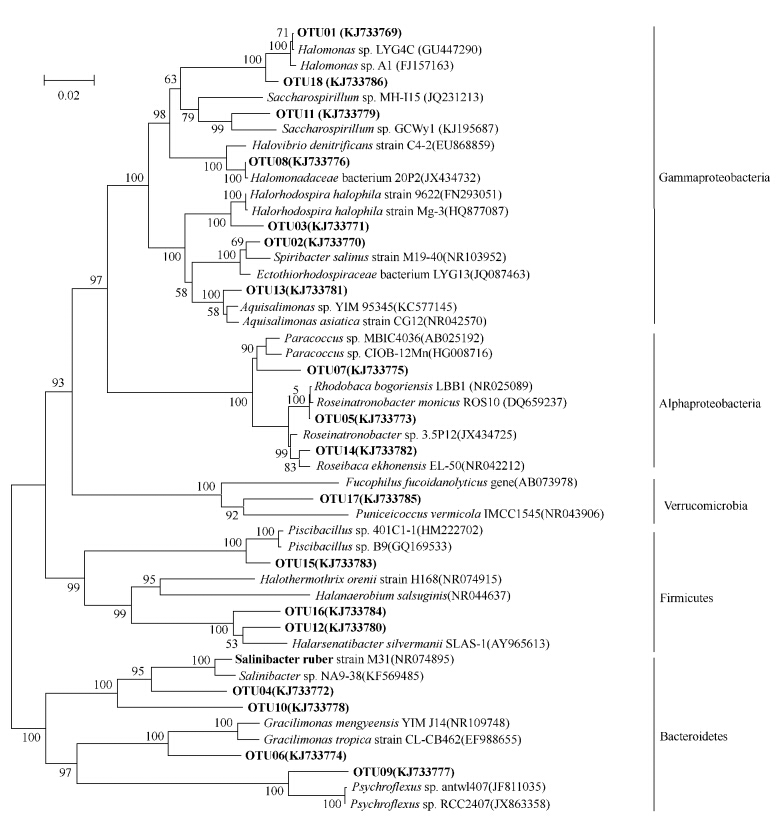
为进一步了解每个OTU的系统发育地位,以表3中的18个OTU为基础,绘制了细菌的系统发育树(图5)。
|
| 图5. 细菌系统发育树 Figure 5. Phylogenetic tree for bacteria. The number in parentheses is accession number in GenBank, node number represents the confidence level of relatives, the length of branches represents the evolutionary distance and the coefficient is 0.02. |
OTU01和OTU18属于盐单胞菌属(Halomonas),在样品1和3-1中占主导地位,分别占克隆文库的33%和23%,同时为3-2的第二大属,占21%。但该OTU在2号湖的样品中没有检出。该菌为中度嗜盐菌,在NaCl浓度小于200 g/L的环境中可大量生长。可耐受高至pH=12的碱性环境,并且所适应的温度范围广泛,在10-45℃之间都可生长[6, 7, 8]。因此,其最适生长条件与1、3号湖的环境条件极为相似。而2号湖的盐度均在30%以上,过高的盐度导致了2号湖中没有该属细菌的分布。
OTU02为Spiribacter属成员,在样品3-2中占主导地位,占其细菌克隆文库的32%;同时为样品1号和3-1的第二大分类单元,分别占14%和13%,但该基因型在其他样品中没有出现。该属最早分离自西班牙的一个盐田,同样为中度嗜盐菌,最适生长条件为150 g/L NaCl[9]。其最适盐度与样品3-2和样品1的盐度均比较接近,因此成为3-2和1的优势种属。而在盐度高于200 g/L的其他样品中则完全不能生长。
OTU03为盐红螺菌属(Halorhodospira)成员,在样品2-1和2-2中都为第二大分类单元,分别占28%和14%;在其他样品中没有出现。该属1996年从外硫红螺菌属(Ectothiorhodospira)中独立出来,并定义为极端嗜盐菌[10]。与其同源性为97%的H.halophila为光能自养的紫色硫细菌(purple sulfur bacteria,PSB),可在厌氧条件下获得光能并将H2S氧化为硫酸盐,在硫的生物循环中起重要作用[11]。
通常,厌氧环境是紫色硫细菌生存的必要条件,过高的溶解氧抑制硫细菌的生长。此外,较高的pH会对微生物的硫循环产生正面影响。首先,高碱环境下,硫化物完全以HS-离子形式存在,与H2S相比,HS-不能自由通过细胞膜,因此毒性较小。所以,嗜碱性硫细菌比嗜中性硫细菌能耐受更高的基质和产物浓度。其次,硫化物与单质硫发生化学反应,形成在高pH下较稳定的聚硫化物,因而为硫细菌提供相对较高的基质浓度。高盐度对硫细菌的氧化作用也是有利的,因为在细胞周质中,硫酸盐在硫化物氧化过程中起到缓冲作用[12]。因此,2号湖中较低的溶解氧以及极高的盐度、pH为该菌属的生长提供有利条件。
OTU04为样品2-1中最为丰富的OTU,占其细菌文库总克隆数的44%。该克隆属与拟杆菌门的Rhodothermaceae科,与其相似性最高的已培养属为Salinibacter,但相似性只有91%。
OTU05为Roseinatronobacter属成员,在样品2-2中占主导地位,占其细菌文库总克隆数的33%。该属目前有两个种,与其同源性为99%的R. monicus菌株分离自苏打盐湖,该菌嗜碱性(pH 8.5- 10.0)、中度嗜盐(0-80 g/L NaCl)[13]。与其同源性为97%的R. thiooxidans分离自干旱草原中的苏打盐湖,可在pH为8.5-10.4,NaCl浓度为6-120 g/L的条件下生长,可以产生橙红色的类胡萝卜素[14]。值得注意的是,已知的该属菌种多为中度嗜盐菌,生长范围为0-120 g/L NaCl。但本研究的结果表明,该菌可在盐度高达344 g/L的2-1样品中出现,并占主导地位。可推断此菌种对高盐环境有一定的耐受性,但其如何成为高盐环境中的优势菌种,其原因仍待进一步研究。
由此可见,因为2号湖矿化度远大于其他两湖,其NaCl浓度已达到饱和并有结晶析出,而同时还具有较高的pH,因此湖中有较多与已鉴定菌种同源性较低的细菌种属。
2.4.2 古菌文库的系统发育学分析:基于Mothur软件生成的代表性序列,将每种基因型的代表序列输入NCBI网站,用BLAST程序与数据库中的已有序列进行比对,主要比对结果见表4。样品中的古菌全部属于广古菌门(Euryarchaeota)的盐杆菌科(Halobacteriaceae),例如Halonotius、Halohasta、Natronomonas、Halorubrum、Halovenus等。
| Clone ID | Gene frequency/% | Closest cultured bacteria (accession number, similarity) | Phylogenetic classification | ||||
| 1 | 2-1 | 2-2 | 3-1 | 3-2 | |||
| OTU01 | 24.0 | 43.2 | 43.2 | 2.6 | 0 | Halohasta litorea strain R30 (NR_117806, 94%) | Euryarchaeota |
| OTU02 | 26.0 | 38.6 | 38.6 | 2.6 | 0 | Halohasta litchfieldiae strain tADL (NR_118135, 96%) | Euryarchaeota |
| OTU13 | 2.0 | 0 | 0 | 0 | 0 | Halobacteriaceae archaeon T58PU (KJ161494, 94%) | Euryarchaeota |
| OTU22 | 2.0 | 0 | 0 | 0 | 0 | Halohasta litorea strain R30 (NR_117806, 97%) | Euryarchaeota |
| OTU03 | 8.0 | 0 | 0 | 84.2 | 84.6 | Archaeon JDS-1 (AF196290, 99%) | Euryarchaeota |
| OTU11 | 2.0 | 0 | 0 | 0 | 0 | Archaeon JDS-1 (AF196290, 97%) | Euryarchaeota |
| OTU04 | 2.0 | 2.3 | 4.5 | 2.6 | 7.7 | Natronomonas moolapensis (EF127644, 98%) | Euryarchaeota |
| OTU16 | 0 | 0 | 2.3 | 0 | 0 | Natronomonas sp. SA2 (JF950946, 96%) | Euryarchaeota |
| OTU18 | 0 | 0 | 0 | 2.6 | 0 | Natronomonas gomsonensis strain SA3 (JF950943, 94%) | Euryarchaeota |
| OTU05 | 6.0 | 4.5 | 2.3 | 0 | 0 | Halovenus aranensis strain: JCM 18639 (AB840772, 94%) | Euryarchaeota |
| OTU06 | 8.0 | 2.3 | 2.3 | 0 | 0 | Haloarchaeon EB27K(JN202399, 94%) | Euryarchaeota |
| OTU12 | 2.0 | 0 | 0 | 0 | 0 | Halovenus aranensis strain: JCM 18639 (AB840772, 92%) | Euryarchaeota |
| OTU17 | 2.0 | 0 | 0 | 0 | 0 | Halovenus aranensis strain: JCM 18639 (AB840772, 92%) | Euryarchaeota |
| OTU07 | 6.0 | 2.3 | 2.3 | 0 | 0 | Halorubrum tibetense strain: JCM 11889 (AB663423, 97%) | Euryarchaeota |
| OTU08 | 4.0 | 2.3 | 0 | 0 | 0 | Halorubrum luteum strain CGSA15 (DQ987877, 99%) | Euryarchaeota |
| OTU09 | 0 | 2.3 | 2.3 | 2.6 | 0 | Halorubrum sp. TRM20006 (KF620438, 99%) | Euryarchaeota |
| OTU10 | 0 | 0 | 0 | 0 | 7.7 | Halorubrum alkaliphilum (NR113468, 98%) | Euryarchaeota |
| OTU14 | 0 | 0 | 2.3 | 0 | 0 | Halorubrum sp. Cb34 strain Cb34 (HG421007, 94%) | Euryarchaeota |
| OTU15 | 2.0 | 0 | 0 | 0 | 0 | Halorubrum lipolyticum (AB663415, 96%) | Euryarchaeota |
| OTU21 | 0 | 0 | 0 | 2.6 | 0 | Halorubrum luteum gene (AB576125, 96%) | Euryarchaeota |
| OTU23 | 0 | 2.3 | 0 | 0 | 0 | Halorubrum sp. GC-8 (JX188264, 98%) | Euryarchaeota |
| OTU19 | 2.0 | 0 | 0 | 0 | 0 | Salinarchaeum sp. MC-74 (AB850662, 92%) | Euryarchaeota |
| OTU20 | 2.0 | 0 | 0 | 0 | 0 | Haloterrigena sp. L52 (GQ181208, 94%) | Euryarchaeota |
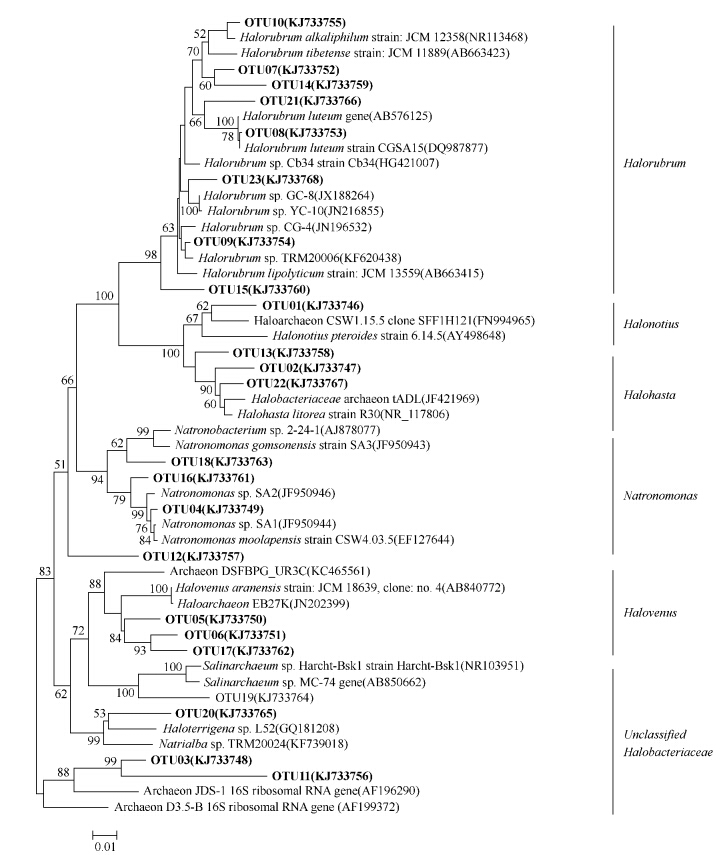
为进一步了解样品中OTU的系统发育学地位,绘制了如图6所示的古菌系统发育树。
|
| 图6. 古菌系统发育树 Figure 6. Phylogenetic tree for archaea. The number in parentheses is accession number in GenBank, node number represents the confidence level of relatives, the length of branches represents the evolutionary distance and the coefficient is 0.01. |
OTU01在2-1和2-2样品中占主导地位,都占克隆文库的43%,同时为1号样的第二大属,占24%。Halonotius pteroide与其同源性最高,但只有93%[15]。该菌种最早由Burns在2009年分离自盐场结晶器,嗜中性,最适盐度为200-240 g/L。
OTU02和OTU13、OTU22都属于Halohasta属。为1号样品的主导菌属,占克隆文库的26%,同时为2-1和2-2的第二大属,都占到其古菌文库总克隆数的39%。该属有两个种,分别为H. litorea和H. litchfieldiae。该属古菌中度嗜盐,可在NaCl为120-280 g/L(最适150-180 g/L),pH 5.9-9.0(最适7.0-7.5)的环境中生长[16]。而采样点的pH均在10以上,大于该属已知种的生长范围。同时,2号湖的矿化度大于340 g/L,为该属最适盐度的两倍以上。由此可推断,OTU02有可能为一个新种或新属,其性质仍待进一步研究。
OTU03为3号湖的优势种,在3-1和3-2中分别达到84%和85%。与GenBank比对发现,OTU03与JDS-1 (AF196290)有99%的同源性,该序列与Haloferax属、Halococus属和Halobacterium属均具有较近的亲缘关系[17]。
OTU04和OTU16、OTU18属于嗜盐碱单胞菌属(Natronomonas),在所有样品中均有分布但比例较低。该属为极端嗜盐菌,并可在pH为5.5-10.0的范围内生长,最适为7.0-8.5。最适温度37-40℃[18]。该属最适pH远低于采样点pH(>10),同时最适温度远高于采样点温度(≈25℃),因此不能在这种环境中大量繁殖。
同样在5个样品中同时出现的盐红菌属(Halorubrum)在文库中有8个基因型,分别为OTU7、8、9、10、14、15、21、23。尽管物种的丰度很高,但在数目上所占的比例很小。盐红菌属是盐杆菌科研究最早的属,属下已培养的种多达24个[19],其中有4个种为嗜碱性,其余为嗜中性,多数首先分离自中国的新疆、西藏及内蒙古地区[20, 21, 22]。本研究检测到的盐红菌属既有嗜碱性菌,也有嗜中性菌[23, 24]。
3 讨论对沙漠盐湖水样的物化分析表明,此区域为高盐高碱双重极端环境,水样的矿化度均大于165 g/L,同时pH均在10以上。研究发现,特殊的高盐碱环境使得嗜盐碱及耐盐碱微生物大量存在,并具有较高的生物多样性。这可使我们深入了解极端环境下微生物群落与环境因素之间的关系,同时可以分离培养出新的微生物类群。
从多样性上来讲,三个盐湖均呈现出较高的细菌生物多样性,而古菌生物多样性在2、3号湖中较低。水样中的细菌主要属于变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、疣微菌门(Verrucomicrobia)、放线菌门(Actinabacteria);而古菌只在广古菌门(Euryarchaeota)中出现。
细菌的分布与环境因子之间的关系比较明显,盐度是产生湖泊间细菌群落结构差异的主要因素,在中、高盐度的样品之间,生物群落存在很大差异:1、3号湖以Halomonas属和Spiribacter属等中度嗜盐菌为主,而2号湖则聚集了大量像盐红螺菌属(Halorhodospira)这样的可耐受高盐碱强度的极端嗜盐碱菌。同时,研究发现,Roseinatronobacter属为样品2-2的优势菌属,该属中度嗜盐,可生长盐度为0-120 g/L,但样品2-2的盐度却高达344 g/L。这是在嗜盐菌研究中的新发现,对Roseinatronobacter属的生长条件范围做了一个新的补充。
此外,2号湖中有较多的紫色硫细菌,而在1、3号湖中没有发现。已有研究表明,盐度越高,溶解氧就越低,微生物便趋于利用光能。这种不产氧光合细菌(anoxygenic phototrophic bacteria),对于整个湖泊的初级生产力有很大贡献[5]。低溶解氧可以保证硫细菌接受光能。高pH有利于减小H2S对微生物的毒性,并可保证较高的反应基质浓度。高盐度可在细胞周质中为硫化物的氧化过程起到缓冲作用。因此,高盐碱的厌氧环境对硫细菌的氧化作用是有利的。
同一湖泊两个样品间细菌群落结构也有细微差异,这主要是因为不同的采样点物化参数有一定差异。如3-1和3-2的盐度分别为205.21和165.48 g/L。相应的,Spiribacter属细菌在3-2中所占比例为32%,而在3-1中仅为13%。经鉴定,该属为中度嗜盐菌,最适盐度为150 g/L,在盐度高于200 g/L的环境中生长缓慢,因此盐度是产生这种差异的主要原因。
古菌的分布没有决定性的环境因子,而是由多个物化参数综合影响。综合分析这5个样品发现,古菌文库中都只有少数几种菌在文库中占主导地位,而其他的古菌数量较少,尤其是3号湖最为明显。Oren等人发现,高盐湖泊中的嗜盐古菌种类很少,一般只有一个或几个属[25]。产生嗜盐菌素是嗜盐古菌的普遍性质[26],嗜盐菌素可以抑制其他古菌的生长,优先利用环境中的营养,或溶解古菌,释出内容物以丰富环境中的营养。嗜盐菌素在嗜盐古菌的生态分布中起重要作用,使古菌群落有明显的占据主导地位的菌种。在3号湖中占主导地位的OTU03在系统发育树上与Haloferax属、Halococus属和Halobacterium属具有较近的亲缘关系,而这几个属的菌都可产生抑菌普较广的嗜盐菌素[27],从而抑制其他古菌的生长。
由于环境条件特殊,在细菌和古菌文库中都发现了很多与已知序列的同源性在93%以下的类群,并且这些类群在各盐湖中有较高的丰度,说明有培养和鉴定出新种或新属的可能,如细菌的OTU04和古菌的OTU01。另外,出于对嗜盐嗜碱菌生理生化性质及生态功能的研究,本课题组已从样品中分离纯化出多株γ-变形菌亚门(Gammaproteobacteria)Halononas属和Idiomarina属以及厚壁菌门(Firmicutes)芽孢杆菌属(Bacillus)的细菌。
| [1] | Yan MC, Wang GQ, Li BS, Dong GR. Formation and growth of high megadunes in Badain Jaran Desert. Journal of Geographical Sciences, 2001, 56(1): 83-91. (in Chinese)闫满存,王光谦,李保生,董光荣. 巴丹吉林沙漠高大沙山的形成发育研究. 地理学报, 2001, 56(1): 83-91. |
| [2] | Jones BF, Eugster HP, Rettig SL. Hydrochemistry of the lake Magadi Basin, Kenya. Geochimica et Cosmochimica Acta, 1977, 41(1): 53-72. |
| [3] | Shao TJ, Zhao JB, Dong YB. Water chemistry of the lakes and groundwater in the Badain Jaran Desert. Journal of Geographical Sciences, 2011, 66(5): 662-672. (in Chinese)邵天杰,赵景波,董治宝.巴丹吉林沙漠湖泊及地下水化学特征.地理学报, 2011, 66(5): 662-672. |
| [4] | 国家环境保护总局.水和废水监测分析方法.第四版.北京:中国环境科学出版社, 2002. |
| [5] | Grant WD, Sorokin DY. Distribution and diversity of soda lake alkaliphiles//Horikoshi K. Extremophiles handbook.1. Japan:Springer Verlag, 2011. |
| [6] | Jiang J, Pan Y, Hu S, Zhang X, Hu B, Huang H, Hong S, Meng J, Li C, Wang K. Halomonas songnenensis sp. nov., a moderately halophilic bacterium isolated from saline and alkaline soils. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt 5): 1662-1669. |
| [7] | Miao C, Jia F, Wan Y, Zhang W, Lin M, Jin W. Halomonas huangheensis sp. nov., a moderately halophilic bacterium isolated from a saline-alkali soil. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt 3): 915-920. |
| [8] | Sánchez-Porro C, Kaur B, Mann H, Ventosa A. Halomonas titanicae sp. nov., a halophilic bacterium isolated from the RMS Titanic. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(12): 2768-2774. |
| [9] | León MJ, Fernández AB, Ghai R, Sánchez-Porro C, Rodriguez-Valera F, Ventosa A. From metagenomics to pure culture: isolation and characterization of the moderately halophilic bacterium Spiribacter salinus gen. nov., sp. nov. Applied and Environmental Microbiology, 2014, 80(13): 3850-3857. |
| [10] | Imhoff JF, Süling J. The phylogenetic relationship among Ectothiorhodospiraceae: a reevaluation of their taxonomy on the basis of 16S rDNA analyses. Archives of Microbiology, 1996, 165(2): 106-113. |
| [11] | Tank M, Thiel V, Imhoff JF. Phylogenetic relationship of phototrophic purple sulfur bacteria according to pufL and pufM genes. International Microbiology, 2010, 12(3): 175-185. |
| [12] | Sorokin DY, Kuenen JG, Muyzer G.The microbial sulfur cycle at extremely haloalkaline conditions of soda lakes. Frontiers in Microbiology, 2011, 44(2): 1-16. |
| [13] | Boldareva EN, Bryantseva IA, Tsapin A, Nelson K, Sorokin DY, Tourova TP, Boichenko VA, Stadnichuk IN, Gorlenko VM. The new alkaliphilic bacteriochlorophyll a-containing bacterium Roseinatronobacter monicus sp. nov. from the hypersaline soda Mono Lake (California, United States). Microbiology, 2007, 76(1): 82-92. |
| [14] | Sorokin DY, Kuznetsov BB, Bryantseva IA, Gorlenko VM, Tourova TP. Roseinatronobacter thiooxidans gen. nov., sp, nov., a new alkaliphilic aerobic bacteriochlorophyll a-containing bacterium isolated from a soda lake.Microbiology,2000, 69 (1):75-82. |
| [15] | Burns DG, Janssen PH, Itoh T; Kamekura M; Echigo A; Dyall-Smith ML. Halonotius pteroides gen. nov., sp. nov., an extremely halophilic archaeon recovered from a saltern crystallizer. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(5): 1196-1199. |
| [16] | Mou YZ, Qiu XX, Zhao ML, Cui HL;Dickson O, Dyall-Smith ML. Halohasta litchfieldiae sp. nov., isolated from the Daliang aquaculture farm, China and from deep lake, antarctica, respectively. Extremophiles, 2012, 16(6): 895-901. |
| [17] | Cytryn E, Minz D, Oremland RS, Cohen Y. Distribution and diversity of archaea corresponding to the limnological cycle of a hypersaline stratified lake(solar lake, Sinai, Egypt),Applied and Environmental Microbiology,2000,66(8):3269-3276. |
| [18] | Burns DG, Janssen PH, Itoh T, Minegishi H, Usami R, Kamekura M, Dyall-Smith ML. Natronomonas moolapensis sp. nov., non-alkaliphilic isolates recovered from a solar saltern crystallizer pond, and emended description of the genus Natronomonas. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(5): 1173-1176. |
| [19] | Gutiérrez MC, Castillo AM, Corral P, Kamekura M, Ventosa A. Halorubrum aquaticum sp. nov., an archaeon isolated from hypersaline lakes. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(5): 1144-1148. |
| [20] | Mwatha WE, Grant WD. Natronobacterium vacuolatum sp. nov., a haloalkaliphilic archaeon isolated from lake Magadi, Kenya. International Journal of Systematic Bacteriology, 1993, 43: 401-404. |
| [21] | Fan H, Xue Y, Ma Y, Ventosa A, Grant WD. Halorubrum tibetense sp. nov., a novel haloalkaliphilic archaeon from lake Zabuye in Tibet, China. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(4): 1213-1216. |
| [22] | Feng J, Zhou P, Zhou YG, Liu SJ, Warren-Rhodes K. Halorubrum alkaliphilum sp. nov., a novel haloalkaliphile isolated from a soda lake in Xinjiang, China. International Journal of Systematic and Evolutionary Microbiology, 2005,55(1): 149-152. |
| [23] | Cui HL, Tohty D, Zhou PJ, Liu SJ. Halorubrum lipolyticum sp. nov. and Halorubrum aidingense sp. nov., isolated from two salt lakes in Xin-Jiang, China. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(7): 1631-1634. |
| [24] | Hu L, Pan H, Xue Y, Ventosa A, Cowan DA, Jones BE, Grant WD, Ma Y. Halorubrum luteum sp. nov., isolated from lake Chagannor, Inner Mongolia, China. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(7): 1705-1708. |
| [25] | Oren A. The Prokaryotes: A Handbook on the Biology of Bacteria: Ecophysiology, Isolation, Identification, Applications. 3rd. New York: Springer-Verlag, 1999. |
| [26] | Torreblanca M, Meseguer I, Ventosa A. Production of halocin is a practically universal feature of archaeal halophilic rods. Letters in Applied Microbiology,1994,19(4):201-205. |
| [27] | Li Y, Xiang H, Tan HR. Halocin: Protein antibiotics produced by extremely halophilic archaea. Acta Microbiologica Sinica, 2002,42(4):502-505. (in Chinese)厉云,向华,谭华荣.极端嗜盐古菌蛋白类抗生素-嗜盐菌素.微生物学报, 2002,42(4):502-505. |
 2015, Vol. 55
2015, Vol. 55