中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 王琳琳, 王颖芳, 段广才, 薛泽润, 郭向娇, 王鹏飞, 郗园林, 杨海燕. 2015
- Linlin Wang, Yingfang Wang, Guangcai Duan, Zerun Xue, Xiangjiao Guo, Pengfei Wang, Yuanlin Xi, Haiyan Yang. 2015
- 志贺菌CRISPR的检测及其与耐药的关系
- Detection of CRISPR and its relationship to drug resistance in Shigella
- 微生物学报, 201555(4): 476-483
- Acta Microbiologica Sinica, 201555(4): 476-483
-
文章历史
- 收稿日期:2014-06-22
- 修回日期:2014-10-13
2. 河南科技大学预防医学教研室,河南 洛阳 471003
3. 新乡医学院分子诊断与医学检验技术河南省协同创新中心,河南 新乡 453003
2 Department of Preventive Medicine, Henan University of Science and Technology, Luoyang 471003, Henan Province, China
3 Henan Innovition Center of Molecular Diagnosis and Laboratory Medicine, Xinxiang Medical University, Xinxiang 453003, Henan Province, China
志贺菌属是细菌性痢疾(简称菌痢)的病原体,菌痢是全球流行的传染病,至今仍是影响人类健康的主要肠道传染病之一,是全球所面临的重要公共卫生问题[1]。随着抗生素的广泛使用甚至滥用,志贺菌频繁变异产生耐药株,多重耐药志贺菌株不断出现,给细菌性痢疾的防治带来新的挑战[2]。
近来研究发现成簇规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR)广泛分布于细菌和古细菌中,CRISPR主要由重复序列(repeat)和间隔序列(spacer)间隔排列构成的R-S结构组成,其与CRISPR相关蛋白基因(CRIPSPR associated,cas)组成CRISPR/cas系统,为原核生物提供对噬菌体、质粒等外源基因的获得性免疫能力,从而抵抗噬菌体感染,限制基因的水平转移(HGT)[3, 4]。耐药基因的水平转移是细菌耐药性产生的主要原因[5],目前有关CRISPR在原核生物中的功能及应用受到广泛关注,如细菌分型[6]、细菌进化分析[7]、抵御噬菌体[8]及Cas9介导的基因定点编辑技术[9]等,但其与细菌耐药关系的研究较少,且不同细菌中CRISPR与耐药的关系不一致,研究表明CRISPR/cas系统与肠球菌获得性耐药关系相反[10, 11],即使CRISPR存在,耐药质粒仍可在大肠杆菌间传播[12]。本研究通过检测志贺菌中的CRISPR及各菌株的耐药情况,探讨志贺菌CRISPR的结构及分布与耐药的关系,为进一步阐明CRISPR的功能以及其对细菌耐药的影响提供参考依据。
1 材料和方法 1.1 菌株临床分离的志贺菌60株,1996年分离自江西6株,1999-2001、2003-2013年分离自河南48株,2013年分离自北京市5株,2004年购于中国药检所1株,其中,45株为福氏志贺菌,13株为宋内志贺菌,2株为痢疾志贺菌;药敏试验标准控制菌:大肠埃希菌ATCC 25922,购自中国普通微生物菌种管理保藏中心。
1.2 药品和试剂药敏纸片氨苄西林(Ampicillin,AM)、头孢噻吩(Cephalothin,CF)、四环素(Tetracycline,TE)、复方新诺明(Trimethoprim/sulfamethoxazole,SXT)、氯霉素(Chloramphenicol,Cl)及诺氟沙星(Norfloxacin,NOR)购自杭州天和微生物试剂有限公司,聚合酶链反应(PCR)相关试剂、100 bp DNA Ladder购自上海生工生物工程股份有限公司,细菌基因组DNA 提取试剂盒购于上海莱枫生物科技有限公司,MH(A)培养基购于北京奥博星生物技术有限责任公司,酵母浸粉、胰蛋白胨购自英国Oxoid公司,琼脂粉购自Sigma公司。其余常规试剂均为国产分析纯级。
1.3 CRISPR检测根据CRISPR DB数据库公布的志贺菌确定的CRISPR结构序列CRISPR-S2、CRISPR-S4 和可能的CRISPR结构序列CRISPR-S1、CRISPR-S3设计引物(引物序列见表1),其中,CRISPR-S4与cas基因组成CRISPR/cas系统,CRISPR-S1、CRISPR-S2和CRISPR-S3均为孤立位点。
| Gene | Primer | Sequence (5′→3′) | PCR products /bp | Annealing temperature /℃ |
| CRISPR-S1 | S1F | ATTAGTCGGCGTAAGAAAGA | 609 | 56 |
| S1R | GAACAGCGTGATTATGGATGC | |||
| CRISPR-S2 | S2F | TTGTYAGGTAGGTTGGTGAAG | 715 | 56 |
| S2R | GCGAAGAGAAAGAACGAGTA | |||
| CRISPR-S3 | S3F | ATCTCTGCTAACACCAACTAC | 711 | 56 |
| S3R | CTACGACCCTGAATGGAATC | |||
| CRISPR-S4 | S4F | AGCGACTAACTGGAAT CTTG | 651 | 56 |
| S4R | CAATCTG GCTACTGGAAGTG |
以试剂盒提取的志贺菌基因组DNA为模板,对志贺菌的CRISPR进行扩增和序列测定。扩增体系为50 μL,其中2×Taq PCR Master Mix 25 μL,ddH2O 19 μL,5 pmol/μL正、反向引物各1 μL,200-300 μg/mL模板DNA 4 μL。CRISPR反应条件均为:94℃预变性5 min;94℃变性60 s,51℃退火60 s,72℃延伸60 s,32个循环;72℃延伸10 min。取5 μL PCR扩增产物进行2%琼脂糖凝胶电泳,用凝胶成像仪观察并记录结果。CRISPR的扩增产物送上海生物工程股份有限公司测序,利用CRISPR Finder,分析CRISPR的重复和间隔序列,确认CRISPR的存在和结构,并利用序列比对软件ClustalX 2.1比较各CRISPR位点的重复序列和间隔序列。
1.4 药敏试验采用改良Kirby-Bauer(K-B)法进行药敏试验,参照临床和实验室标准化研究所(CLSI)指南进行[13],质控菌株为大肠埃希菌ATCC 25922。分析志贺菌对不同药物的耐药分布情况,对3种及以上不同种类抗生素耐药的细菌为多重耐药菌。
1.5 统计学分析采用SPSS17.0 统计软件处理数据,CRISPR与耐药的分布采用x2检验或Fisher’s确切概率检验,P <0.05 为差异有统计学意义。
2 结果 2.1 60株志贺菌CRISPR的分布和结构 2.1.1 CRISPR的检测结果:以60株志贺菌基因组DNA为模板,PCR扩增CRISPR-S1、 CRISPR-S2、CRISPR-S3和 CRISPR-S4,采用CRISPR Finder查找分析CRISPR,结果见表2,含3个及以上spacer的CRISPR位点检出率为31.67%。| Gene | Spacer number | Strain number | Percentage /% |
| CRISPR-S1 | 1 | 20 | 33.33 |
| CRISPR-S2 | 1 | 1 | 51.67 |
| 2 | 30 | ||
| CRISPR-S3 | 1 | 48 | 80 |
| CRISPR-S4 | 2 | 25 | 73.33 |
| 3 | 18 | ||
| 9 | 1 |
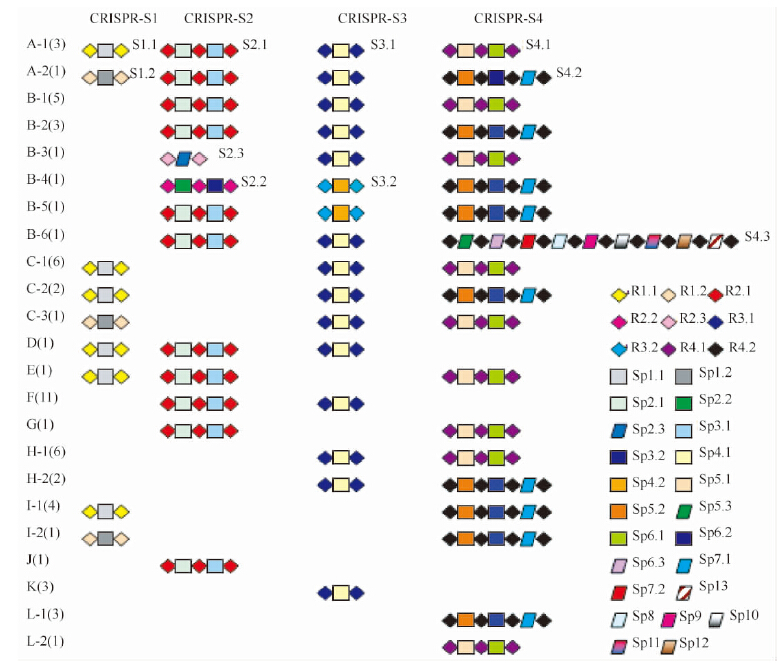
| Type | CRISPR spectral pattern | Strain number | Percentage /% |
| A | CRISPR-S1+CRISPR-S2+CRISPR-S3+CRISPR-S4 | 4 | 6.67 |
| B | CRISPR-S2+ CRISPR-S3+ CRISPR-S4 | 12 | 20 |
| C | CRISPR-S1+ CRISPR-S3+ CRISPR-S4 | 9 | 15 |
| D | CRISPR-S1+ CRISPR-S2+ CRISPR-S3 | 1 | 1.67 |
| E | CRISPR-S1+ CRISPR-S2+ CRISPR-S4 | 1 | 1.67 |
| F | CRISPR-S2+ CRISPR-S3 | 11 | 18.34 |
| G | CRISPR-S2 + CRISPR-S4 | 1 | 1.67 |
| H | CRISPR-S3+ CRISPR-S4 | 8 | 13.32 |
| I | CRISPR-S1+ CRISPR-S4 | 5 | 8.32 |
| J | CRISPR-S2 | 1 | 1.67 |
| K | CRISPR-S3 | 3 | 5 |
| L | CRISPR-S4 | 4 | 6.67 |

|
| 图 1. CRISPR的重复序列比对 Figure 1. Sequence alignment of CRISPR repeats. Figures in bracket represented the number of strains with corresponding repeat. “*” represented perfect match, “:” represented strong similarity, “.” represented weak similarity. |

|
| 图 2. CRISPR的间隔序列比对 Figure 2. Sequence alignment of CRISPR spacers. Figures in bracket represented the number of strains with corresponding spacer. “*” represented perfect match, “:” represented strong similarity, “.” represented weak similarity. |
|
| 图 3. 60株志贺菌CRISPR的结构 Figure 3. The CRISPR structures in the 60 Shigella strains A→L represented the 12 CRISPR spectral patterns. Figures in bracket represented the number of strains with corresponding CRISPR spectral pattern. Rhombus represented new spacers found in this study. Two different layouts of repeat and spacer sequences existed in CRISPR-S1 (S1.1, S1.2) and CRISPR-S3 (S3.1, 3.2) respectively, three different layouts in CRISPR-S2 (S2.1, S2.2, S2.3) and CRISPR-S4 (S4.1, S4.2, S4.3) respectively. |
60株志贺菌对所检测的6种抗生素的耐药率如下:AM 为56.67%,CF为26.67%,TE为66.67%,SXT为65%,C为48.33%,NOR为16.67%。其中,对6种抗生素均不耐药的志贺菌8株,仅耐1种抗生素的志贺菌11株,耐2种抗生素的志贺菌9株,3种及以上耐药的多重耐药菌32株,多重耐药率为53.33%。
对6种抗生素均不耐药的志贺菌株的CRISPR谱型多为C型,多重耐药志贺菌株的CRISPR谱型多为B型、F型和H型(表4)。
|
Drug resistance | CRISPR spectral pattern | Total | |||||||||||
| A | B | C | D | E | F | G | H | I | J | K | L | ||
| none | 0 | 2 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 8 |
| single | 1 | 2 | 0 | 0 | 0 | 2 | 0 | 2 | 3 | 0 | 1 | 0 | 11 |
| two | 2 | 1 | 2 | 0 | 0 | 0 | 0 | 0 | 2 | 1 | 0 | 1 | 9 |
| multi | 1 | 7 | 2 | 1 | 1 | 9 | 1 | 6 | 0 | 0 | 1 | 3 | 32 |
| total | 4 | 12 | 9 | 1 | 1 | 11 | 1 | 8 | 5 | 1 | 3 | 4 | 60 |
CRISPR-S4 阳性志贺菌株与阴性志贺菌株之间,多重耐药的分布差异无统计学意义,TE耐药性的分布差异亦无统计学意义,见表5。
| * Fisher’s exact text; S: sensitivity, I: intermediated resistance, R: resistance. | ||||||||||||||||||||||
| Drug resistance | CRISPR-S4 | Repeat | Sp5 | Sp6 | Sp7 | |||||||||||||||||
| - | + | P | - | R4.1 | R4.2 | P | - | Sp5.1 | Sp5.2 | Sp5.3 | P | - | Sp6.1 | Sp6.2 | Sp6.3 | P | - | Sp7.1 | Sp7.2 | P | ||
|
Antibiotics Type | none | 1 | 7 | 0.361 | 1 | 4 | 3 | 0.034 | 1 | 4 | 2 | 1 | 0.022* | 1 | 4 | 2 | 1 | 0.022* | 5 | 2 | 1 | 0.008* |
| single | 3 | 8 | 3 | 1 | 7 | 3 | 1 | 7 | 0 | 3 | 1 | 7 | 0 | 4 | 7 | 0 | ||||||
| Two | 1 | 8 | 1 | 4 | 4 | 1 | 4 | 4 | 0 | 1 | 4 | 4 | 0 | 5 | 4 | 0 | ||||||
| MDR | 11 | 21 | 11 | 16 | 5 | 11 | 16 | 5 | 0 | 11 | 16 | 5 | 0 | 27 | 5 | 0 | ||||||
| TE | S | 4 | 15 | 0.667* | 4 | 5 | 10 | 0.039* | 4 | 5 | 9 | 1 | 0.052* | 4 | 5 | 9 | 1 | 0.052* | 9 | 9 | 1 | 0.015* |
| I | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | ||||||
| R | 12 | 28 | 12 | 20 | 8 | 12 | 20 | 8 | 0 | 12 | 20 | 8 | 0 | 32 | 8 | 0 | ||||||
CRISPR-S4 的重复序列与多重耐药的分布差异有统计学意义,与TE耐药性的分布差异亦有统计学意义。多重耐药志贺菌株和耐TE志贺菌株CRISPR-S4的重复序列多为R4.1。
CRISPR-S4 的间隔序列Sp5、Sp6、Sp7与多重耐药的分布差异有统计学意义,Sp7与TE耐药性的分布差异亦有统计学意义。多重耐药志贺菌株CRISPR-S4的间隔序列多为Sp5.1和Sp6.1,多重耐药志贺菌株和耐TE志贺菌株大多无Sp7。CRISPR-S4的间隔序列Sp8到Sp13,与Sp5.3、Sp6.3及Sp7.2同在1株志贺菌中存在,该志贺菌株对6种抗生素均不耐药。
CRISPR-S4 阳性志贺菌株与阴性志贺菌株之间,AM、CF、SXT、Cl和NOR耐药性的分布差异均无统计学意义;CRISPR-S4 的重复序列与AM、CF、SXT、Cl和NOR耐药性的分布差异均无统计学意义;CRISPR-S4 的间隔序列Sp5、Sp6、Sp7,与AM,CF,SXT,Cl和NOR耐药性的分布差异均无统计学意义。
3 讨论1996年志贺菌被WHO认定为给人类带来巨大威胁、耐药性日渐增长的菌株[14]。随着抗生素的滥用,志贺菌不断变异产生耐药株,甚至多重耐药株。我们对60株志贺菌的耐药情况进行检测,发现志贺菌对AM、TE、SXT、C的耐药率较高,而对CF和NOR的耐药率较低,多重耐药率为53.33%,与任静朝等[15]报道结果一致。CRISPR广泛分布于细菌和古细菌的基因组中,本研究检测了60株志贺菌的可能的CRISPR结构CRISPR-S1、CRISPR-S3,及确定的CRISPR结构 CRISPR-S2、CRISPR-S4。4个CRISPR位点均被检出,它们形成了12种谱型(A-L),除K型仅含可能的CRISPR结构CRISPR-S3,其他各型均含确定的CRISPR结构,确定的CRISPR结构的总阳性率为95%,与CRISPR DB数据库中公布的已测序的基因组中,81.82%志贺菌(9/11)和87.72%(5/57)的大肠杆菌中存在确定的CRISPR结构一致,与国内邓凯波在2株德氏乳杆菌和8株嗜热链球菌中均检测出CRISPR 位点结果一致[16, 17],并且薛泽润等[18]在志贺菌中检测到CRISPR相关蛋白基因cas1和cas2。
CRISPR与cas基因组成CRISPR/cas系统,间隔序列靶向定位入侵的外源性基因,通过Cas蛋白将外源性基因裂解,从而限制基因的水平转移,抵抗噬菌体感染及质粒接合转移等[19]。而耐药基因的水平转移是志贺菌耐药的主要机制之一,目前有关CRISPR与耐药关系的报道在不同的细菌中尚不一致,Katie M.等[10]检测了粪肠球菌的CRISPR相关蛋白基因cas1和cas3,发现:cas1或cas3与多重耐药的获得关系相反,Kelli[11]分析粪肠球菌的CRISPR/cas,亦发现:CRISPR/cas与获得性耐药关系相反。但是,大肠杆菌的cas I-E在敏感株和耐药株间无差异,并且耐药质粒仍可以在CRISPR阳性大肠杆菌之间传播[12]。本研究中CRISPR-S1、CRISPR-S2、CRISPR-S3均为孤立位点,不与cas基因相连接,而CRISPR-S4与cas基因组成CRISPR/cas系统。因此,我们分析了CRISPR-S4与志贺菌多重耐药的分布,发现:虽然CRISPR-S4阳性菌株与阴性菌株之间,多重耐药的分布差异无统计学意义,但多重耐药志贺菌株的重复序列主要为R4.1,间隔序列主要为Sp5.1及Sp6.1。
同一CRISPR位点的重复序列相对保守,在同种细菌的不同菌株间高度相似,仅在少数菌株中发生变异[20]。本研究检测到的4个位点,仅3株志贺菌CRISPR-S1的重复序列、2株志贺菌CRISPR-S2的重复序列、2株志贺菌CRISPR-S3的重复序列和部分CRISPR-S4的重复序列发生变异。重复序列的变异并不是简单的碱基突变,重复序列的变化会引起紧邻的间隔序列的变化。间隔序列与CRISPR/cas系统发生免疫抑制作用密切相关,间隔序列可靶向定位入侵的外源性基因,而CRISPR/cas系统可获取入侵的外源基因的一段标志基因为新的间隔序列,从而抵御这一外源基因的再次侵入。本研究中发现多重耐药菌株缺乏Sp5.2,Sp5.3、Sp6.2、Sp6.3及Sp7,TE耐药菌株缺乏Sp7,Marraffini LA[21]发现CRISPR/cas系统的间隔序列与质粒切口酶同源,从而限制耐药基因的水平转移,提示Sp5.2、Sp5.3、Sp6.2、Sp6.3及Sp7可能与携带相应耐药基因的质粒同源。间隔序列的变异可导致免疫逃逸[22],有趣的是,Bikard D等研究发现[23],通过间隔序列变异甚至CRISPR的丢失,连接有耐药基因的前间隔序列(prospacer,间隔序列的同源基因序列)可逃逸CRISPR干扰,使肺炎链球菌获得相应的耐药基因。在志贺菌中,我们发现:CRISPR-S4阳性的多重耐药志贺菌株,其重复序列的3′末端缺失碱基AC,其后排列的间隔序列为Sp5.1和Sp6.1,而非Sp5.2、Sp5.3、Sp6.2、Sp6.3及Sp7,提示CRISPR尽管能限制外源性基因的侵入,在抗生素的选择性生存压力下,细菌的CRISPR可能发生变异,使细菌获得外源性耐药基因而存活下来,CRISPR重复序列的变异和间隔序列的多样性可能与志贺菌耐药有关。
| [1] | WHO. Shigellosis: disease burden, epidemiology and case management. Weekly Epidemiological Record, 2005, 80(11): 94-99. |
| [2] | Zhang W, Luo Y, Li J, Lin L, Ma Y, Hu C, Jin S, Ran L, Cui S. Wide dissemination of multidrug-resistant Shigella isolates in China. The Journal of Antimicrobial Chemotherapy, 2011, 66(11): 2527-2535. |
| [3] | Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea. Science, 2010, 327(5962): 167-170. |
| [4] | Wiedenheft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea. Nature, 2012, 482(7385): 331-338. |
| [5] | Rowe-Magnus DA, Guerout AM, Mazel D. Bacterial resistance evolution by recruitment of super-integron gene cassettes. Molecular Microbiology, 2002, 43(6): 1657-1669. |
| [6] | Cain AK, Boinett CJ. A CRISPR view of genome sequences. Nature Reviews Microbiology, 2013, 11(4): 226. |
| [7] | Comas I, Homolka S, Niemann S, Gagneux S. Genotyping of genetically monomorphic bacteria: DNA sequencing in Mycobacterium tuberculosis highlights the limitations of current methodologies. PLoS One, 2009, 4(11): e7815. |
| [8] | Horvath P, Romero DA, Coute-Monvoisin AC, Richards M, Deveau H, Moineau S, Boyaval P, Fremaux C, Barrangou R. Diversity, activity, and evolution of CRISPR loci in Streptococcus thermophilus. Journal of Bacteriology, 2008, 190(4): 1401-1412. |
| [9] | Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F. Genome engineering using the CRISPR-Cas9 system. Nature Protocols, 2013, 8(11): 2281-2308. |
| [10] | Burley KM, Sedgley CM. CRISPR-Cas, a prokaryotic adaptive immune system, in endodontic, oral, and multidrug-resistant hospital-acquired Enterococcus faecalis. Journal of Endodontics, 2012, 38(11): 1511-1515. |
| [11] | Palmer KL, Gilmore MS. Multidrug-resistant enterococci lack CRISPR-cas. MBio, 2010, 1(4): e00227-10. |
| [12] | Touchon M, Charpentier S, Pognard D, Picard B, Arlet G, Rocha EPC, Denamur E, Branger C. Antibiotic resistance plasmids spread among natural isolates of Escherichia coli in spite of CRISPR elements. Microbiology, 2012, 158: 2997-3004. |
| [13] | Clinical and Laboratory Standards Institute. Performance Standards for Antibiotic Disk Susceptibility Tests. Eleventh edition: Clinical and Laboratory Standards Institute, 2012. |
| [14] | Davies J. Bacteria on the rampage. Nature, 1996, 383(6597): 219-220. |
| [15] | Ren JC, Duan GC, Song CH, Lv RL, Zhang WD, Xi YL. Relation of acrAB-tolC efflux pump and marOR regulated gene mutation with antimicrobial resistance in Shigella. Journal of Jilin University, 2010, 36(1): 45-48. (in Chinese) 任静朝, 段广才, 宋春花, 吕锐利, 张卫东, 郗园林. 志贺菌主动外排泵acrAB-tolC及调控基因marOR突变与耐药的关系. 吉林大学学报,2010,36(1):45-48. |
| [16] | Deng KB, Huo GC. Detection and homology analysis of CRISPR in Streptococcus thermophilus. Food Science, 2013, 34(3): 153-157. (in Chinese) 邓凯波, 霍贵成. 嗜热链球菌中CRISPR序列的检测与同源性分析. 食品科学,2013,34(3):153-157. |
| [17] | Deng KB, Huo GC, Liu GX. Detection and homology analysis of CRISPR in Lactobacillus delbruekii. Journal of Northeast Agricultural University, 2011,42(11): 19-23. (in Chinese) 邓凯波, 霍贵成, 柳国霞. CRISPR在德氏乳杆菌中的检测与同源性分析. 东北农业大学学报,2011,42(11): 19-23. |
| [18] | Xue ZR, Wang YF, Duan GC, Wang PF, Wang LL, Guo XJ, Xi YL. Clustered regularly interspaced short palindromic repeat associated protein genes cas1and cas2 in Shigella. Chinese Journal of Epidemiology, 2014, 5(35): 105-108. (in Chinese) 薛泽润, 王颖芳, 段广才, 王鹏飞, 王琳琳, 郭向娇, 郗园林. 志贺菌成簇规律间隔短回文重复序列相关蛋白基因cas1和cas2研究. 中华流行病学杂志,2014,5(35):105-108. |
| [19] | Jansen R, Embden JD, Gaastra W, Schouls LM. Identification of genes that are associated with DNA repeats in prokaryotes. Molecular Microbiology, 2002, 43(6): 1565-1575. |
| [20] | Deveau H, Garneau JE, Moineau S. CRISPR/Cas system and its role in phage-bacteria interactions. Annual Review of Microbiology, 2010, 64: 475-493. |
| [21] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science, 2008, 322(5909): 1843-1845. |
| [22] | Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712. |
| [23] | Bikard D, Hatoum-Aslan A, Mucida D, Marraffini LA. CRISPR interference can prevent natural transformation and virulence acquisition during in vivo bacterial infection. Cell Host & Microbe, 2012, 12(2): 177-186. |
 2015, Vol. 55
2015, Vol. 55