中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 李尔汉, 杨慧林, 王筱兰, 万令飞, 潘红梅, 朱笃
- Erhan Li, Huilin Yang, Xiaolan Wang, Lingfei Wan, Hongmei Pan, Du Zhu
- 肌苷生产菌枯草芽孢杆菌ATCC 13952的全基因组测序及序列分析
- Whole-genome sequencing and analysis of inosine-producing strain Bacillus subtilis ATCC 13952
- 微生物学报, 2015,55(12): 1560-1567
- Acta Microbiologica Sinica, 2015,55(12): 1560-1567
-
文章历史
- 收稿日期:2015-04-03
- 修回日期:2015-05-18
2江西科技师范大学,江西省生物加工过程重点实验室,江西 南昌 330013
2Jiangxi Key Laboratory of Bioprocess, Jiangxi Science & Technology Normal University, Nanchang 330013, Jiangxi Province, China
肌苷(Inosine)学名为9-β-D-呋喃核糖基次黄嘌呤,分子结构由次黄嘌呤和核糖两部分组成。肌苷的用途十分广泛:在食品工业中,肌苷主要用于生产食品增鲜剂肌苷酸二钠[1];在医药工业中,肌苷适用于各种原因引起的白细胞减少症、血小板减少症、各种心脏疾患、急性及慢性肝炎、肝硬化等各种疾病[2]。
目前,肌苷主要是通过微生物发酵法生产,该法具有产率高、产量大、周期短、易控制等优点[3]。肌苷生产菌主要是革兰氏阳性细菌,包括解淀粉芽孢杆菌(Bacillus amyloliquefaciens)、枯草芽孢杆菌(Bacillus subtilis)和短小芽孢杆菌(Bacillus brevis)等[4],这些菌株的共同特点是磷酸戊糖途径(Pentose phosphate pathway,PPP)比较活跃,有利于葡萄糖合成嘌呤合成途径的直接前体物磷酸核糖焦磷酸(PRPP)。
枯草芽孢杆菌ATCC 13952是一株用于生产肌苷的专利菌株[5],最初肌苷产量并不是很高,随后经多轮常规育种和分子育种,肌苷的生产水平逐步提高[6, 7, 8, 9, 10, 11]。然而,要想进一步提高菌株积累肌苷的产量,必须对菌株的遗传背景有更加深入的了解。杨慧林[12]和陈宁[13]等分别发表鸟苷生产菌解淀粉芽孢杆菌XH7和TA208株全基因组测序简报,但是肌苷生产菌全基因组测序仍未见报道。本研究首次报道了肌苷生产菌枯草芽孢杆菌ATCC 13952的全基因组序列,分析了基因组基本特征,初步探讨了该菌株积累肌苷的分子机制,为后续的进一步分子育种提供了理论基础。
1 材料和方法 1.1 菌株培养与基因组DNA的提取枯草芽孢杆菌ATCC 13952(Bacillus subtilis ATCC 13952)购自中国工业微生物菌种保藏管理中心(China Center of Industrial Culture Collection,CICC)。菌种冻干粉复苏后,于LB培养基中28 ℃培养24 h,采用OMEGA基因组DNA提取试剂盒提取基因组DNA,操作步骤按照试剂盒说明书进行。
1.2 基因组测序、组装与注释基因组DNA提取后采用Illumina Hiseq 2000测序平台完成全基因组测序,采用拼接软件velvet 1.2.10完成序列拼接。采用序列对比软件Mummer将contig序列(>1 kb)比对到解淀粉芽孢杆菌LL3株[14]的基因组序列上,确定contig序列间的相互位置关系,设计引物进行PCR,对PCR产物进行测序完成补洞。采用Glimmer 3.02[15]软件进行开放阅读框(ORF)预测,将所有预测蛋白序列与NR(非冗余蛋白数据库)、Swiss-Prot数据库[16]、COG(Clusters of orthologous groups,直系同源基因簇数据库)、KEGG(Kyoto encyclopedia of genes and genomes,京都基因组和基因组百科全书数据库)[17]、Interpro数据库[18]和Gene ontology(GO)数据库[19]进行BLASTp(E-value<1e-10)比对,完成蛋白序列功能注释。采用RNAmmer[20]和tRNAscan-SE[21]软件分别进行rRNA和tRNA的预测。
1.3 比较基因组学分析将枯草芽孢杆菌ATCC 13952株的基因组序列与其他已经测序完成的芽孢杆菌基因组序列进行比较分析,包括鸟苷工业生产菌株解淀粉芽孢杆菌XH7株(GenBank: CP002927)和TA208株(GenBank: CP002627)、不积累核苷的标准菌株DSM 7(GenBank: FN597644)、枯草芽孢杆菌168株(GenBank: AL009126),4株菌的基因组序列及注释信息下载自GenBank数据库。统计5株菌基因组的基本特征,采用BLASTp对5株菌进行共有基因和特异基因的分析,采用Mauve[22]软件进行基因组共线性分析,程序采用默认参数运行。
1.4 嘌呤代谢相关基因分析根据预测蛋白序列与KEEG数据库比对后的结果及KEGG PATHWAY数据库,重建菌株的嘌呤代谢通路(Purine metabolism,PATH:ko00230)图,并对定位属于嘌呤代谢通路的65个基因及COG功能分类中归入核苷酸转运与代谢(F,Nucleotide transport and metabolism)类别的其他嘌呤代谢相关基因进行分析,研究菌株积累肌苷的分子机制。
2 结果和分析 2.1 基因组组装与注释使用Velvet 1.2.10软件完成序列拼接(原始序列7655714个paired-end reads,读长90 bp,插入片段大小500 bp,测序深度170×),拼接获得48个contigs(其中25个大于1000 bp),N50大小为412061 bp。补洞完成后,获得完整的枯草芽孢杆菌ATCC 13952基因组序列,序列全长3876276 bp,其GC含量为45.8%,编码4005个基因,正链上1932个,负链上2073个,编码序列占整个基因组序列的89.3%,平均长度为864 bp,其中包含7个16S-23S-5S核糖体rRNA操纵子和72个tRNA。基因组圈图见图1。

|
| 图1 枯草芽孢杆菌ATCC 13952基因组圈图 Figure 1 Circle map of the genome of B. subtilis ATCC 13952. Key for circular diagrams (outside to inside): 1: scale (in Mb); 2: gene in the positive strand; 3: gene in the negative strand,CDSs are colored according to predicted functions; 4: G+C content; 5: GC skew [(G-C)/(G+C)] (green indicates values>0,and purple indicates values<0). |
枯草芽孢杆菌ATCC 13952基因组包含3852个CDS,将其编码对应蛋白序列与COG数据库比对完成同源基因注释分类,得到包括信息储存及处理、细胞生物学过程及信号传导、基本代谢、未知功能的编码基因(图2)。如图2所示,共有2609个蛋白获得COG功能注释,其中参与能量产生及传递(C,Energy production and conversion)、氨基酸代谢及转运(E,Amino acid transport and metabolism)、糖类转运及代谢(G,Carbohydrate transport and metabolism)、翻译,核糖体结构及合成(J,Translation,ribosomal structure and biogenesis)、转录(K,Transcription)、细胞壁/细胞膜/包膜合成(M,Cell wall/membrane/envelope biogenesis)的蛋白占据较大比例,分别为7%、13%、6%、6%、10%和6%。

|
| 图2 B. subtilis ATCC 13952的COG功能分类 Figure 2 Gene distribution based on COG classification of B. subtilis ATCC 13952. |
将CDS编码蛋白序列与Interpro数据库和Gene ontology(GO)数据库比对,获得GO注释信息,使用WEGO(http://wego.genomics.org.cn/cgi-bin/wego/index.pl)在线工具进行GO功能聚类分析(图3)。如图3所示,GO功能分类主要对细胞组分(Cellular component)、分子功能(Molecular function)、生物学过程(Biological process)进行分类,其中归类为细胞(Cell)、细胞部位(Cell part)、结合(Binding)、催化(Catalytic)、细胞内过程(Cellular process)、代谢过程(Metabolic process)的基因数量占据优势,表明菌株活性多集中于分化、酶催化及代谢等。

|
| 图3 B. subtilis ATCC 13952的GO功能分类 Figure 3 Gene distribution based on Gene Ontology of B. subtilis ATCC 13952. |
5株芽孢杆菌基因组的基本特征如表1所示,菌株在基因组大小、GC含量、基因编码密度等方面非常相似,表明B. subtilis ATCC 13952具有芽孢杆菌的典型特征。菌株在CDS数目、CDS平均长度和RNA基因数目方面存在一些差异,表明菌株处在不同进化阶段,进化过程中丢失或新获得了一些不同的基因。采用BLASTp将B. subtilis ATCC 13952编码的所有基因与其他4株菌比对发现,2249个基因为5株菌所共有,223个基因为B. subtilis ATCC 13952所特有。对这些特有基因编码的蛋白进行分析发现,其中包括一些tRNA和rRNA相关蛋白(主要是甲硫氨酰tRNA合成酶、rRNA小亚基甲基转移酶、tRNA-二氢尿嘧啶核苷合成酶等一些蛋白)、转录调控蛋白、酶、未知蛋白等,这些特有基因导致了B. subtilis ATCC 13952特有的遗传性状。
| B. subtilis ATCC 13952 | B. amyloliquefaciens XH7 | B. amyloliquefaciens TA208 | B. amyloliquefaciens DSM 7 | B. subtilis 168 | |
| Genome size/bp | 3876276 | 3939203 | 3937511 | 3980199 | 4215606 |
| G+C content/% | 45.8 | 45.8 | 45.8 | 46.1 | 43.5 |
| Protein-coding sequences | 3852 | 4190 | 4089 | 3922 | 4255 |
| Average CDS size/bp | 877 | 824 | 844 | 885 | 872 |
| Percent of coding region | 89.3 | 88.6 | 88.5 | 88.6 | 89.7 |
| Ribosomal RNA operons | 7 | 7 | 6 | 10 | 10 |
| Number of tRNA | 72 | 72 | 70 | 94 | 86 |
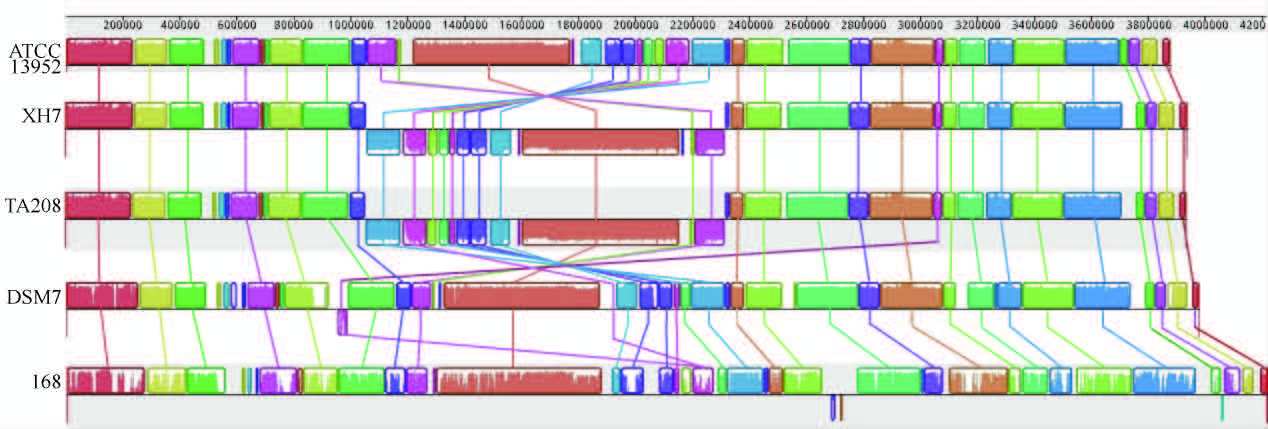
采用Mauve软件对5株芽孢杆菌的全基因组进行比对分析,比对结果见图4。如图4所示,5株芽孢杆菌基因组间的共线性关系较好,有38个LCBs(Locally collinear blocks)。5株菌基因组之间存在着一些插入和缺失,以及少量的倒位、易位等基因组重排事件。其中,解淀粉芽孢杆菌XH7和TA208株相对于其他基因组存在1个较大的DNA片段(1.26 Mb)倒位,Bose等发现的两个新的潜在的原噬菌体正好位于该DNA片段的侧翼[23],推断此倒位很可能是由于感染噬菌体,从而介导水平基因转移造成的[13]。
|
| 图4 芽孢杆菌基因组共线性分析 Figure 4 Synteny block of Bacillus spp. Genomes. The same color modules joined by linear indicate collinear region,and the white color modules indicate the low-similarity region. The above is forward strand,and the below is reverse strand. |
将通过KEGG定位属于嘌呤代谢通路的基因所编码的蛋白序列分别取出来与不积累核苷的标准株解淀粉芽孢杆菌DSM 7的相应蛋白进行BLASTp比对分析,比对结果见表2。表2分析结果表明,枯草芽孢杆菌ATCC 13952株中发生了一些非常明显地有利于肌苷生成的突变,如purA基因发生终止密码子突变,合成的腺苷酰琥珀酸合成酶失去活性,导致IMP生成AMP的支路途径中断,从而使IMP生成肌苷或GMP的代谢流量增加;缺失drm基因,无法合成磷酸核糖变位酶,5-磷酸核糖不再向1-磷酸核糖转化,转而参与到嘌呤合成途径中;缺失pbuX基因的大部分片段,合成的黄嘌呤通透酶失去活性,IMP生成GMP支路途径的中间产物XMP不能被及时转运到胞外,引起产物反馈抑制,有利于IMP生成肌苷。另外,发现了一些潜在的可能促进菌株生成肌苷的突变,如purF、guaA、pgi、purM、purL、purC等,其编码的蛋白均发生了1个或多个氨基酸突变,可能会改变相应酶的活性,对代谢造成影响。然而,多数蛋白并未发生突变,表明菌株有进一步菌种改良的潜力。
| Mutant forms | Genes | Proteins | Functions |
| No amino acid mutation | prs | Ribose-phosphate pyrophosphokinase | Converts ribose 5-phosphate into phosphoribosyl pyrophosphate |
| guaB | Inosine-monophosphate dehydrogenase | Converts inosine monophosphate to xanthosine monophosphate | |
| guaC | GMP reductase | Catalyses the irreversible and NADPH-dependent reductive deamination of GMP into IMP | |
| yunD | Nucleotidase | Nucleotidase | |
| pupG | Purine nucleoside phosphorylase | Metabolizes adenosine into adenine,inosine into hypoxanthine,and guanosine into guanine | |
| purR | Purine operon repressor | Repress purine operon transcription | |
| deoD | Purine nucleoside phosphorylase | Metabolizes adenosine into adenine | |
| pbuE | Hypoxanthine efflux transporter | Inosine specific transporter | |
| One or more amino acid mutations | purF | Glutamine-PRPP amidotransferase | Converts PRPP into 5-phospho-β-D-ribosylamine |
| guaA | GMP synthetase | Converts XMP into GMP | |
| pgi | Glucose-6-phosphate isomerase | Catalyzes the conversion of glucose-6-phosphate into fructose 6-phosphate | |
| purM | Phosphoribosylaminoimidazole synthetase | Converts FGAM into AIR | |
| purL | Phosphoribosylformylglycinamidine synthetase II | Converts FGAR into FGAM | |
| purC | Phosphoribosylaminoimidazole succinocarboxamide synthetase | Converts CAIR into SAICAR | |
| Insertion or deletion mutation (Loss of function) | purA | Adenylosuccinate synthetase | Converts IMP into SAMP |
| drm | Phosphopentomutase | Catalyzes the conversion of alpha-D-ribose 1-phosphate into D-ribose 5-phosphate | |
| pbuX | Xanthine permease | Transport xanthine through membrane |
本研究通过全基因组测序方法,获得枯草芽孢杆菌ATCC 13952的基因组序列,基因组的基本特征与其他芽孢杆菌属菌株非常相似,比较基因组分析显示几株芽孢杆菌间的共线性关系较好,枯草芽孢杆菌ATCC 13952具有保守的基因组结构。枯草芽孢杆菌ATCC 13952作为肌苷工业生产菌,通过嘌呤代谢相关基因的分析,发现了一些非常关键的突变,这些突变导致了嘌呤代谢途径的改变,最明显的改变是由于缺失了purA基因导致了IMP→AMP支路途径的中断,这也与菌株的腺苷酸缺陷型特征相符合。另外,还发现了一些潜在的有利于菌株积累肌苷的突变,这些突变究竟起到了多大的作用,还需要后续的实验进行验证。然而,多数的相关基因并未发生突变,其中一些基因对于肌苷合成也起到了非常重要的作用,菌株仍然具有较大进一步改良的潜力。对菌株进行改良大致可以从以下几个方面进行:增加前体物质;阻断支路途径;解除反馈阻遏及反馈抑制等不利调控机制。对菌株进行改造,目前主要地操作就是根据需要对相应的基因进行突变、敲除或其他修饰等,以达到改变菌株代谢特性的目的。然而,在一株菌中引入如此多的突变势必会极大地增加菌株的负担,产生不良的影响,结果就会适得其反[24]。采用新的遗传操作技术,可以避免这些不利因素,达到预期的目的。Harris H. Wang等[25]采用MAGE(multiplex automated genome engineering)技术成功地同时对24个目的基因进行了修饰,提高了番茄红素在大肠杆菌中产量。随着对工业核苷生产菌的遗传背景有了足够深入的研究,MAGE也能够用于枯草芽孢杆菌生产肌苷的改造工程中。
| [1] | Zhou RJ, Ning ZX, Song XL. Review on processes couplings in food engineering and bioengineering. Food Science, 2002, 23(12): 125-128. 周如金, 宁正祥, 宋贤良. 食品及生物工程中的过程耦合技术. 食品科学, 2002, 23(12): 125-128. |
| [2] | Dong M, Shao QF. Physical and chemical properties of inosine and its application. Journal of Nanchang Vocational and Technical Normal College, 1997, (2): 92-95. (in Chinese) 董明, 邵琼芳. 肌苷的理化性质及其应用. 南昌职业技术师范学院学报, 1997, (2): 92-95. |
| [3] | Demain AL, Jackson M, Vitali RA, Hendlin D, Jacob TA. Production of guanosine-5′-monophosphate and inosine-5′-monophosphate by fermentation. Applied and Environmental Microbiology, 1966, 14(5): 821-825. |
| [4] | Zakataeva NP, Livshits VA, Gronsky SV, Kutukova EA, Novikova AE, Kozlov YI. Method for producing purine nucleosides and nucleotides by fermentation using bacterium belonging to the genus Bacillus or Escherichia. US Patent: 8034767.2011. |
| [5] | Tominaga M, Shimazaki K, Matsuno K, Yamashita M. Method of converting heterocyclic bases into the corresponding nucleosides by bacterial action. US Patent: 3535207.1970. |
| [6] | Mori H, Iida A, Fujio T, Teshiba S. A novel process of inosine 5′-monophosphate production using overexpressed guanosine/inosine kinase. Applied Microbiology and Biotechnology, 1997, 48(6): 693-698. |
| [7] | Shimaoka M, Takenaka Y, Mihara Y, Kurahashi O, Kawasaki H, Matsui H. Effects of xapA and guaA disruption on inosine accumulation in Escherichia coli. Bioscience, Biotechnology and Biochemistry, 2006, 70(12): 3069-3072. |
| [8] | Chen SX, Chu J, Zhuang YP, Zhang SL. Enhancement of inosine production by Bacillus subtilis through suppression of carbon overflow by sodium citrate. Biotechnology Letters, 2005, 27(10): 689-692. |
| [9] | Li HJ, Zhang GQ, Deng AH, Chen N, Wen TY. De novo engineering and metabolic flux analysis of inosine biosynthesis in Bacillus subtilis. Biotechnology Letters, 2011, 33(8): 1575-1580. |
| [10] | Shimaoka M, Takenaka Y, Kurahashi O, Kawasaki H, Matsui H. Effect of amplification of desensitized purF and prs on inosine accumulation in Escherichia coli. Journal of Bioscience and Bioengineering, 2007, 103(3): 255-261. |
| [11] | He KF, Ma YC, Du SS, Xie XX, Xu QY, Chen N. Effects of overexpression of key enzyme genes on guanosine accumulation in Bacillus amyloliquefaciens. Acta Microbiologica Sinica, 2012, 52(6): 718-725. (in Chinese) 何逵夫, 马跃超, 杜姗姗, 谢希贤, 徐庆阳, 陈宁. 解淀粉芽胞杆菌关键酶基因过表达对鸟苷积累的影响. 微生物学报, 2012, 52(6): 718-725. |
| [12] | Yang HL, Liao YL, Wang B, Lin Y, Pan L. Complete genome sequence of Bacillus amyloliquefaciens XH7, which exhibits production of purine nucleosides. Journal of Bacteriology, 2011, 193(19): 5593-5594. |
| [13] | Zhang GQ, Deng AH, Xu QY, Liang Y, Chen N, Wen TY. Complete genome sequence of Bacillus amyloliquefaciens TA208, a strain for industrial production of guanosine and ribavirin. Journal of Bacteriology, 2011, 193(12): 3142-3143. |
| [14] | Geng WT, Cao MF, Song CJ, Xie H, Liu L, Yang C, Feng J, Zhang W, Jin YH, Du Y, Wang SF. Complete genome sequence of Bacillus amyloliquefaciens LL3, which exhibits glutamic acid-independent production of poly-γ-glutamic acid. Journal of Bacteriology, 2011, 193(13): 3393-3394. |
| [15] | Delcher AL, Bratke KA, Powers EC, Salzberg SL. Identifying bacterial genes and endosymbiont DNA with Glimmer. Bioinformatics, 2007, 23(6): 673-679. |
| [16] | Boeckmann B, Bairoch A, Apweiler R, Blatter MC, Estreicher A, Gasteiger E, Martin MJ, Michoud K, O'Donovan C, Phan I, Pilbout S, Schneider M. The SWISS-PROT protein knowledgebase and its supplement TrEMBL in 2003. Nucleic Acids Research, 2003, 31(1): 365-370. |
| [17] | Kanehisa M, Goto S. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Research, 2000, 28(1): 27-30. |
| [18] | Mulder NJ, Apweiler R, Attwood TK, Bairoch A, Bateman A, Binns D, Biswas M, Bradley P, Bork P, Bucher P, Copley R, Courcelle E, Durbin R, Falquet L, Fleischmann W, Gouzy J, Griffith-Jones S, Haft D, Hermjakob H, Hulo N, Kahn D, Kanapin A, Krestyaninova M, Lopez R, Letunic I, Orchard S, Pagni M, Peyruc D, Ponting CP, Servant F, Sigrist CJA. InterPro: an integrated documentation resource for protein families, domains and functional sites. Briefings in Bioinformatics, 2002, 3(3): 225-235. |
| [19] | Consortium GO. The Gene Ontology (GO) database and informatics resource. Nucleic Acids Research, 2004, 32(Suppl 1): D258-D261. |
| [20] | Lagesen K, Hallin P, Rdland EA, Staerfeldt HH, Rognes T, Ussery DW. RNAmmer: consistent and rapid annotation of ribosomal RNA genes. Nucleic Acids Research, 2007, 35(9): 3100-3108. |
| [21] | Lowe TM, Eddy SR. tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence. Nucleic Acids Research, 1997, 25(5): 955-964. |
| [22] | Darling ACE, Mau B, Blattner FR, Perna NT. Mauve: multiple alignment of conserved genomic sequence with rearrangements. Genome Research, 2004, 14(7): 1394-1403. |
| [23] | Bose M, Barber RD. Prophage finder: a prophage loci prediction tool for prokaryotic genome sequences. In Silico Biology, 2006, 6(3): 223-227. |
| [24] | Asahara T, Mori Y, Zakataeva NP, Livshits VA, Yoshida KI, Matsuno K. Accumulation of gene-targeted Bacillus subtilis mutations that enhance fermentative inosine production. Applied Microbiology and Biotechnology, 2010, 87(6): 2195-2207. |
| [25] | Wang HH, Isaacs FJ, Carr PA, Sun ZZ, Xu G, Forest CR, Church GM. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460(7257): 894-898. |
 2015, Vol. 55
2015, Vol. 55