中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 沙月霞. 2018
- Yuexia Sha. 2018
- 不同水稻组织内生细菌的群落多样性
- Diversity of bacterial endophytic community in different rice tissues
- 微生物学报, 58(12): 2216-2228
- Acta Microbiologica Sinica, 58(12): 2216-2228
-
文章历史
- 收稿日期:2018-04-02
- 修回日期:2018-07-08
- 网络出版日期:2018-07-23
植物内生菌是重要的微生物资源,其生活史的一定阶段或全部阶段能够定殖在健康植物细胞间隙或细胞内[1],寄主植物受侵染后并不表现病症,可以从严格表面消毒的植物组织中分离得到内生菌或者直接从植物组织内扩增微生物特征DNA片段[2]。植物内生菌多样性的研究可以揭示微生物与寄主植物的互作[3]、评价寄主植物内环境的稳定性[4],一部分植物内生菌能够提高植物抗病性与抗虫性[5]、抗逆境能力[6],在微生物肥料的应用[7]、生防种质资源的研制及利用[8]等技术领域具有重要意义。内生细菌是一类重要的内生微生物,评价和衡量。水稻植株生长的内环境是否稳定和健康的重要指标之一就是具有稳定的内生细菌群落多样性。相关研究表明内生细菌对于水稻植株具有多种生态功能,例如增强水稻抗逆境能力[9]、抗病虫害能力[10]、自身合成或者促进植物合成多种植物生长激素[11-12]和提高植物氮吸收与氮代谢水平[13-14]等,往往与寄主植物形成一种互惠互利的共生关系[15]。在水稻不同生育期,内生细菌动态变化及多样性不同[16],分蘖期菌群分布均匀、种类较多,开花期物种相对单一[17]。成熟期种子内生细菌种类比灌浆期种子丰富[18-19],内生细菌群落结构具有丰富的多样性[20]。与本研究不同的是,已有的研究主要集中在水稻根系、茎部和叶片可培养的内生细菌群落,只有较少的研究报道了水稻根部不可培养内生细菌多样性[21]。本研究的特点在于不仅研究了水稻根、茎和叶部可培养内生细菌的群落多样性,还研究了水稻根、茎和叶部不可培养内生细菌的群落多样性。本文采用高通量测序技术,研究宁夏水稻宁粳43号内生细菌的物种组成、群落结构及多样性,为进一步探讨植物内生细菌的生态功能以及植物与微生物的互作机制提供依据。
1 材料和方法 1.1 温室种植水稻试验地点在宁夏农林科学院植物保护研究所温室。温室种植水稻的土壤配比为:自然土:营养土=3:1。自然土从宁夏银川市贺兰山农牧场采集,土壤类型为灰钙土,有机质平均含量为0.57%,碳酸钙为14.64%,全盐量分别为0.61%,平均含氮量是0.05%,速效磷是5.5 ppm,阳离子交换量7.60 meg/100 g土。
挑选健康水稻种子宁粳43号,用1%次氯酸钠溶液消毒10 min,70%酒精冲洗3次,无菌水冲洗5次,在28 ℃人工气候箱催芽,待芽长到1 cm时播种于塑料小水桶里(水桶底部直径18 cm×上部直径24.5 cm×高21.0 cm)。每个水桶里播种5粒种子,土层厚度8 cm。
1.2 样本采集水稻分蘖期(种植后60 d左右)采集样本,取样时把水稻植株从塑料盆中轻轻取出,不要伤害根系,用无菌水持续冲洗浮土,直至所有肉眼可见异物全部冲洗干净。把洗干净的完整水稻植株放入取样袋中,用冰盒将采集的样品带到实验室。根部样本采集完整根组织,茎部样本采集距离茎基部3 cm的组织,叶部样本采集中部大约10 cm叶段。试验设置3组样本处理,每组样本4个重复处理,分别是叶部样本(L):L1、L2、L3、L4;茎部样本(S):S1、S2、S3、S4;根部样本(R):R1、R2、R3、R4。
1.3 基因组DNA抽提样本在70%酒精中浸泡1 min,重复3次,然后用无菌水反复清洗。严格表面消毒后的样本提取基因组总DNA,然后利用1%琼脂糖凝胶电泳检测抽提的基因组DNA。
1.4 PCR扩增扩增引物为799F:5′-barcode-AACMGGATTA GTAGATACCCKG-3′,1392R:5′-ACGGGCGGTG TGTRC-3′,1193R:5′-ACGTCATCCCCACCTTC C-3′[22],对细菌16S rRNA基因V5–V7高变区进行PCR扩增,引物由上海美吉生物医药科技有限公司设计合成,测序区域合成带有barcode的特异引物。PCR正式试验采用20 μL反应体系:10×PCR Buffer 2.0 μL,2.5 mmol/L dNTPs 2.0 μL,5 μmol/L正向引物0.8 μL,5 μmol/L反向引物0.8 μL,rTaq Polymerase 0.2 μL,BSA 0.2 μL,Template DNA 10 ng,补ddH2O至20 μL。PCR扩增条件:95 ℃ 3 min;循环数×(95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s);72 ℃ 10 min,10 ℃直到反应结束。第一轮扩增引物:799F-1392R 27 cycles,退火55 ℃。第二轮扩增引物:799F-1193R 13 cycles,退火55 ℃。
对第二轮PCR扩增产物用2%琼脂糖凝胶电泳检测,切胶回收PCR产物,Tris-HCl洗脱;2%琼脂糖电泳检测。将PCR产物用QuantiFluor TM- ST蓝色荧光定量系统(Promega公司)进行检测定量,之后进行相应比例的混合。混合后的产物用Illumina公司的Miseq 2×300平台测序。
1.5 数据处理与分析高通量测序得到的原始数据进行质量控制和软件拼接,过滤掉低质量的序列。将有效序列相似性在≥97%序列聚类成为单元,1个单元为1个OTU (Operational Taxonomic Units)。再用QIIME软件进行单样品组成分析,计算样品的Coverage、Sobs、Simpson、Shannon指数等[23-25]。Simpson指数值越大,说明群落多样性越低。Shannon值越大,说明群落多样性越高。Coverage反映测序结果是否代表了样本中微生物的真实情况,其数值越高表明样本中序列被测出的概率越高。相似性分析(analysis of similarities)可以判断样本分组是否有意义,反映样本组内与组间的差异。相似性分析中statistic为R值,范围为-1到+1。如果R值越接近1,说明样本组间的差异越大于组内差异;如果R值越接近-1,说明样本组间和组内的微生物种群没有显著性差异。
2 结果和分析 2.1 水稻样本内生细菌序列统计和多样性本研究采用16S rRNA基因测序技术对水稻植株内生细菌群落多样性进行了分析,明确水稻不同组织部位内生细菌群落结构组成特点,结果见表 1。
| Sample | Sobs | Shannon | Simpson | Coverage/% | Shannon even | Number of sequences | OTUs |
| S | 12.67±2.08 | 0.97±0.26 | 0.50±0.14 | 100 | 0.38±0.09 | 14761 | 411 |
| R | 6.67±1.53 | 0.32±0.18 | 0.84±0.07 | 100 | 0.17±0.05 | 20787 | 174 |
| L | 16.67±2.89 | 1.27±0.11 | 0.35±0.09 | 100 | 0.46±0.07 | 15438 | 610 |
| S means stem treatment; R means root treatment; L means leaf treatment. OTU: the similar effective sequence more than 97% sequence is grouped into unit. | |||||||
宁夏水稻内生细菌叶部、茎部和根部分别获得有效序列数为15438、14761和20787,产生的OTUs分别是610、411和174。根部获得有效序列数最多,但是产生的OTUs最少,可能是有些细菌在数据库里无法找到。宁粳43号水稻植株内生细菌具有丰富的群落多样性,不同组织部位多样性指数不同,结果见表 1。覆盖度测序结果可以代表样本中微生物的真实情况。水稻叶部、茎部和根部内生细菌的覆盖度均为100%,说明取样合理,检测结果能够真实反映水稻叶部、茎部和根部内生细菌群落。比较3组样本的群落丰富度指数、香农指数、优势度指数和均匀度指数,叶部内生细菌群落的丰富度、群落多样性、优势物种多样性和均匀度最高,茎部次之,根部的最低。
稀释曲线往往用来检验测序结果是否可以真实准确地覆盖整个类群,其测序量反映了样品的测序深度。图 1结果显示,水稻根部内生细菌随着测序数量的增加,OTU的变化趋势近似一条直线,而茎部和叶部内生细菌群落在序列数低于3000时OTU变化较大,然后随测序数量的增加OTU变化基本趋于平缓。表明植物内生细菌的种群基本上都在水稻叶部和茎部中检测到,但是在根部只检测出部分细菌种群,仍有部分细菌群落的种群尚未被检测到。

|
| 图 1 水稻不同组织内生细菌的稀释曲线 Figure 1 Rarefaction curve for bacterial endophytic OTUs in rice different tissues. S means stem treatment; R means root treatment; L means leaf treatment. |
样本分组相似性分析结果显示(表 2),不同分组对样品差异的解释度R值接近1,说明3组样本之间的差异(P < 0.001)大于组内差异(P > 0.001)。
| Method | Statistic/R | P value | Permutation_number |
| Analysis of similarities | 0.6543 | 0.008 | 999 |
2.2 水稻叶部内生细菌群落结构组成
对水稻叶部内生细菌610个OTUs的分类结果表明,叶部内生细菌15438个序列中。图 2显示,宁粳43号水稻叶部内生细菌至少隶属于22个不同的细菌门,40个纲,103个目,198个科,399个属。在门水平,有4个细菌门的相对丰度≥1%,为叶部内生细菌中的优势菌群。其中变形菌门(Proteobacteria)的丰度为31.28%,放线菌门(Actinobacteria)的丰度为24.60%,拟杆菌门(Bacteroidetes)的丰度为23.86%,厚壁菌门(Firmicutes)的相对丰度为18.89%,其余18个门所占比例均低于1%,共占1.36%。

|
| 图 2 门水平水稻叶部内生细菌菌群组成 Figure 2 Bacterial endophytic composition of rice leaf at phylum level. |
在纲水平至少隶属于40个纲(表 3),相对丰度≥1%的纲有7个。其中放线菌门的放线菌纲(Actinobacteria) 24.60%、拟杆菌门的拟杆菌纲(Bacteroidia) 22.98%、变形菌门的β-变形菌纲(Betaproteobacteria) 11.65%、厚壁菌门的芽胞杆菌纲(Bacilli) 11.51%和变形菌门α-变形菌纲(Alphaproteobacteria) 10.78%所占比例最高。其次为γ-变形菌纲(Gammaproteobacteria) 8.32%和梭菌纲(Clostridia) 6.77%等。
| Class | Relative abundance/% | Member of phylum |
| Actinobacteria | 24.60 | Actinobacteria |
| Bacteroidia | 22.98 | Bacteroidetes |
| Betaproteobacteria | 11.65 | Proteobacteria |
| Bacilli | 11.51 | Firmicutes |
| Alphaproteobacteria | 10.78 | Proteobacteria |
| Gammaproteobacteria | 8.32 | Proteobacteria |
| Clostridia | 6.77 | Firmicutes |
| Others | 3.39 |
在103个目中,相对丰度≥1%的目有11个。其中拟杆菌目(Bacteroidales) 22.98%和棒状杆菌目(Corynebacteriales) 22.16%所占比例较高,其次为乳酸杆菌目(Lactobacillales) 9.90%、立克次氏体目(Rickettsiales) 8.64%、伯克霍尔德氏菌目(Burkholderiales) 7.63%、梭菌目(Clostridiales) 6.77%等。在隶属的169个科中,相对丰度≥1%的科有15个,排在前4位的是诺卡氏菌科(Nocardiaceae) 21.00%、拟杆菌科(Bacteroidales) 10.10%、诺卡氏菌科(Lactobacillaceae) 9.19%和伯克氏菌科(Burkholderiaceae) 4.24%。
叶部内生细菌在属水平上,全部细菌序列至少有399个属,相对丰度≥1%的属有13个,其中能够准确分类的属有10个。优势菌属主要包含红球菌属(Rhodococcus) 21.00%、乳酸杆菌属(Lactobacillus) 9.19%、拟杆菌属(Bacteroides) 5.31%、罗尔斯通菌属(Ralstonia) 3.13%和气单胞菌属(Aeromonas) 2.17%等。
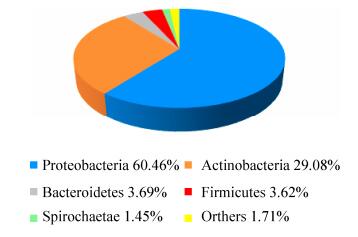
2.3 水稻茎部内生细菌群落结构组成对水稻茎部内生细菌411个OTUs的分类结果表明,茎部内生细菌14761个序列中,隶属于19门31纲85目169科306属。在门水平至少隶属于19个门(图 3),其相对丰度≥1.00%的共5个门。其中变形菌门60.45%、放线菌门29.08%、拟杆菌门3.69%、厚壁菌门3.62%和螺旋体门(Spirochaetae) 1.45%为茎部内生细菌中的优势菌种,其余14个门所占比例均低于1.00%,共占1.71%。
|
| 图 3 门水平水稻茎部内生细菌菌群组成 Figure 3 Bacterial endophytic composition of rice stem at phylum level. |
在纲水平至少隶属于31个纲(表 4),相对丰度≥1%的纲有10个。其中变形菌门的β-变形菌纲31.88%和α-变形菌纲19.32%、放线菌门的放线菌纲29.08%所占比例最高。其次为γ-变形菌纲7.67%和芽胞杆菌纲2.33%等。
| Class | Relative abundance/% | Member of phylum |
| Betaproteobacteria | 31.88 | Proteobacteria |
| Actinobacteria | 29.08 | Actinobacteria |
| Alphaproteobacteria | 19.32 | Proteobacteria |
| Gammaproteobacteria | 7.67 | Proteobacteria |
| Bacilli | 2.33 | Firmicutes |
| Flavobacteriia | 1.71 | Bacteroidetes |
| Deltaproteobacteria | 1.58 | Proteobacteria |
| Spirochaetes | 1.45 | Spirochaetes |
| Bacteroidia | 1.31 | Bacteroidetes |
| Clostridia | 1.28 | Firmicutes |
| Others | 2.38 |
在85个目中,相对丰度≥1%的目有16个。其中伯克霍尔德氏菌目27.45%、棒状杆菌目19.88%、根瘤菌目(Rhizobiales) 12.26%所占比例最高,其次为丙酸杆菌目(Propionibacteriales) 5.92%、肠杆菌目(Enterobacteriales) 3.53%和立克次氏体目2.92%等。在169个科中,相对丰度≥1%的有15个,排在前5位的是诺卡氏菌科19.27%、伯克氏菌科14.94%、丛毛单胞菌科(Comamonadaceae) 11.14%、甲基杆菌科(Methylobacteriaceae) 8.40%和丙酸杆菌科(Propionibacteriaceae) 5.69%。
茎部内生细菌在属水平上,全部细菌序列至少隶属于306个属,有17个细菌属的相对丰度≥1%,其中能够准确分类的属有12个。水稻茎部内生细菌主要有红球菌属19.25%、罗尔斯通菌属13.52%、丙酸杆菌属(Propoinicicella) 5.57%和肠杆菌属(Enterobacter) 3.12%等。
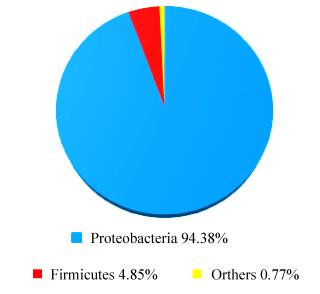
2.4 水稻根部内生细菌群落结构组成对水稻根部内生细菌174个OTUs的分类结果表明,根部内生细菌20787个序列中,隶属于9门19纲44目82科140属。在门水平至少隶属于9个门(图 4),其相对丰度≥1%的共2个门。其中变形菌门94.38%和厚壁菌门4.85%为根部内生细菌中的优势菌种,其余7个门所占比例均低于1.00%,共占0.77%。
|
| 图 4 门水平水稻根部内生细菌菌群组成 Figure 4 Bacterial endophytic composition of rice root at phylum level. |
在纲水平至少隶属于19个纲(表 5),相对丰度≥1%的纲有3个。其中变形菌门的γ-变形菌纲(93.00%)所占比例最高,其次为梭菌纲(3.51%)和芽胞杆菌纲(1.33%)等。在目水平隶属于44个目,相对丰度≥1%的目有3个。其中肠杆菌目丰度最高,占92.82%,其次为梭菌目占3.51%,芽胞杆菌目占1.33%。在82个科中,相对丰度≥1%的科有3个,排在前3位的是肠杆菌科(Enterobacteriaceae) 92.82%、梭菌科(Clostridiaceae) 3.20%和类芽胞杆菌科(Paenibacillaceae) 1.13%。
| Class | Relative abundance/% | Member of phylum |
| Gammaproteobacteria | 93.00 | Proteobacteria |
| Clostridia | 3.51 | Firmicutes |
| Bacilli | 1.33 | Firmicutes |
| others | 2.16 |
根部内生细菌在属水平上,全部细菌序列至少隶属于40个属,有4个细菌属的相对丰度≥1%,其中能够准确分类的属有4个。水稻根部内生细菌主要包含肠杆菌属81.13%、埃希氏杆菌属(Escherichia) 10.89%、多粘类芽胞杆菌属(Paenibacillus) 1.13%和梭菌属(Clostridium) 3.10%等。
2.5 水稻不同组织部位内生细菌的共有和独有物种Venn图用于比较样本中共有和独有的物种(如OTU)数目组成情况。图 5显示,水稻不同组织部位的内生细菌物种中既有共有物种,也有独有的物种。水稻叶部的OTU数量是610个,茎部的OTU数量是411个,根部的OTU数量是174个。叶部、茎部和根部内生细菌的共有OTU物种是78个,叶部独有的物种数是376个,茎部独有物种数是163个,根部独有物种数是22个。除共有物种和独有物种外,叶部与茎部、根部之间有相同或相似物种,茎部与根部之间也有相同或相似物种。

|
| 图 5 水稻不同组织内生细菌物种的Venn分析 Figure 5 Venn species analysis of bacterial endophytes in rice different tissues. S means stem treatment; R means root treatment; L means leaf treatment. |
2.6 水稻内生细菌群落物种相关性网络
物种相关性网络可以表达物种之间是否具有相关性,如果物种与物种之间具有相关性,它们之间就有连线,圆圈越大表示物种的丰度越高。图 6显示水稻内生细菌的物种相关性,变形菌门的细菌属丰度最高,而且与其他水稻组织部位的细菌都有相关性;长螺旋细菌属Spirochaetae属于螺旋体门,只与变形菌门有相关性,与其他物种没有相关性;酸杆菌门Acidobacteria的丰度比较低,与变形菌门、放线菌门具有较近的相关性。放线菌门丰度最高,与变形菌门、酸杆菌门、拟杆菌门Bacteroidetes之间均存在相关性。

|
| 图 6 水稻不同组织内生细菌的物种网络分析 Figure 6 Network analysis of bacterial endophytes in rice different tissues on genus level. |
2.7 水稻不同组织内生细菌群落的功能预测分析
KEGG数据库(Kyoto Encyclopedia of Genes and Genomes)是可以对基因组信息和功能信息进行系统分析的数据库。表 6表明,水稻内生细菌参与了各种代谢通路、合成通路、膜转运、信号传递以及细胞周期相关通路,还有一些细菌可以引起人类疾病,其中根部中参与各种功能活动的物种丰度最高,其次是茎部内生细菌的物种,叶部中调控各种代谢的物种丰度最低。根系中不可分类的内生细菌物种是茎部、叶部的5.6倍。
| Category | S | R | L |
| Cellular processes | 480913 | 2177900 | 405661 |
| Environmental information processing | 2390024 | 12875261 | 1740282 |
| Genetic information processing | 2347231 | 9415806 | 2088326 |
| Human diseases | 144600 | 658429 | 143498 |
| Metabolism | 8865536 | 27211652 | 6332467 |
| None | 31483 | 102592 | 21554 |
| Organismal systems | 156044 | 271851 | 113784 |
| Unclassified | 2037658 | 11398218 | 1620576 |
3 讨论
水稻是世界上最重要的粮食作物之一,是全世界一半人口的主粮食品。我国水稻产业的可持续发展对国民经济和粮食产量发挥着举足轻重的作用。本研究采用的水稻品种为宁粳43号,其植株内生细菌群落多样性丰富,优势度较低,均匀度一般,说明宁粳43号在温室种植的环境中植物内环境较稳定和较健康。温室种植采用的自然土为黑垆土,偏碱性,试验期间气温较高且湿度较低,对宁粳43号内生细菌群落多样性影响较大。
植物内生细菌生活在寄主植物体内,具有非常重要的生态功能[26-27],包括促进植物生长发育、降解重金属污染[28]、降解农药残留、防治病虫草害[29]、研制生物有机肥[7]等。水稻内生细菌群落由可培养和不可培养的内生细菌群落构成,可培养内生细菌可以在实验室分离培养,进而研究报道较多;不可培养内生细菌由于不能在实验室分离培养,需要借助分子生物学手段,故而较少有人研究报道。但是不可培养内生细菌对于整个水稻内生细菌群落的稳定性至关重要,只有了解水稻中可培养和不可培养内生细菌的群落及其多样性,才能系统评价水稻内生细菌群落的多样性与水稻健康状况。微生物多样性测序技术具有高通量、操作简便、快速、数据精确、成本价格低等特点。本研究首次采用微生物多样性测序技术对宁夏水稻宁粳43号叶部、茎部和根部内生细菌群落及其多样性进行了较为详细的研究,列出了水稻品种宁粳43号叶片、茎和根系内生细菌群落所属的门、纲、目、科、属不同分类水平上的优势类群及其相对丰度。此次测序结果表明,水稻品种宁粳43号内生细菌群落在目、科和属水平上仍有较大比例的微生物不能明确分类,但在门和纲水平上获得比较明确的分类名称信息。估计与测序序列长度、测序区间、比对的数据库以及16S rRNA基因本身的局限性等有关。
微生物群落多样性是否稳定会直接影响微生物的生态功能和整个生态系统[30]。本研究表明,与根系内生细菌群落及多样性比较,宁夏水稻品种宁粳43号叶部内生细菌群落的丰富度、群落多样性、均匀度、优势度明显较高,优势物种多样性较低,茎部内生细菌群落及多样性介于叶片和根系之间。变形菌门是宁粳43号丰度最高的优势类群,其中在根系中的丰度最高。变形菌门是植物内生细菌最普遍的菌门,本研究结果与国内外大多数研究结果一致[14-15, 31]。放线菌门是水稻宁粳43号叶部和茎部内生细菌中第二优势类群,说明宁夏环境干旱、高温等气候条件适合于放线菌的生存与繁殖。放线菌可以产生大量的、种类繁多的抗生素,是非常重要的生防微生物。本研究结果表明水稻宁粳43号中具有广泛的抗生素产生菌资源。
水稻内生细菌群落多样性与水稻品种、生育期、环境和气候条件有密切关系。有很多研究表明水稻内生细菌菌群中伯克霍尔德氏菌属、草螺菌属(Herbaspirillum)、根瘤菌(Rhizobium)、甲基杆菌属(Methylobacterium)和芽胞杆菌属为根部优势菌[32],伯克霍尔德氏菌属、固氮螺菌属(Azospirillum)和草螺菌属为茎部优势菌[17],成团泛菌属(Pantoea)、芽胞杆菌属和鞘氨醇单胞菌属(Sphingomonas)为水稻种子中的优势菌群[33-35]。本研究结果与已有研究差异较大,估计与水稻品种、实验土壤、温室温度湿度等环境因素有关。水稻品种的基因型能够影响水稻内生细菌的群落多样性[18, 36],本研究采用的水稻品种为宁粳43号,它的基因型与已有研究中的水稻品种基因型不同,这是引起研究结果差异的主要因素。水稻植株具有一定的营养成分,这些营养成分为内生细菌的生长和繁殖提供必需的营养物质,土壤条件以及栽培环境会影响到植株体内的营养成分,营养成分的积累及其变化对植株联合细菌群落具有重要的影响[17, 35, 37]。宁夏水稻主栽品种宁粳43号内生细菌菌群红球菌属和乳酸杆菌属为叶部优势菌,红球菌属和罗尔斯通菌属为茎部优势菌,肠杆菌属和埃希氏杆菌属为根部优势菌。水稻等谷类作物中丝状真菌常常产生毒性较高的代谢产物霉菌毒素,红球菌属能够有效抑制水稻霉菌毒素的危害,降低稻米的霉变[38]。乳酸杆菌属是对植株健康有益的一类微生物。罗尔斯通菌产生的毒素杀虫效果比较明显,常常用来生物防治害虫[39],对于水稻害虫虫口密度的降低具有重要意义。肠杆菌属普遍存在于土壤中,埃希氏杆菌属也在土壤中发现,据推测本研究根系中的肠杆菌属和埃希氏杆菌属可能来源于土壤。
水稻不同组织部位既有共有物种,又有独有物种。从结果可以推测,土壤中的微生物可以进入水稻根系,部分内生细菌从水稻根系向上部茎部、叶部迁移,根部、茎部和叶部之间产生共有物种。但是随着水稻植株的生长发育,土壤中的一些微生物进入水稻根系后没有继续沿维管束系统扩展,成为根部的独有物种。叶围微生物从气孔或者叶片表皮毛进入水稻叶片[40]成为叶部的独有物种。而水稻茎部由于长时间浸泡在灌溉水中,水中的一些微生物通过伤口或者自然孔口进入茎部成为水稻茎部的独有物种。水稻内生细菌可以调控各种代谢通路、合成通路、膜转运、信号传递、细胞周期以及疾病相关通路等,其中根系参与代谢调控的细菌丰度最高,其次是茎部和叶部。根系中不可分类的内生细菌的物种数量远远大于茎部和叶部。
| [1] | Verma SC, Singh A, Chowdhury SP, Tripathi AK. Endophytic colonization ability of two deep-water rice endophytes, Pantoea sp. and Ochrobactrum sp. using green fluorescent protein reporter. Biotechnology Letters, 2004, 26(5): 425-429. DOI:10.1023/B:BILE.0000018263.94440.ab |
| [2] | Bayman P, Lebrón LL, Tremblay RL, Lodge DJ. Variation in endophytic fungi from roots and leaves of Lepanthes (Orchidaceae). New Phytologist, 1997, 135(1): 143-149. DOI:10.1046/j.1469-8137.1997.00618.x |
| [3] | Rosenblueth M, Martínez-Romero E. Bacterial endophytes and their interactions with hosts. Molecular Plant-Microbe Interactions, 2006, 19(8): 827-837. DOI:10.1094/MPMI-19-0827 |
| [4] | Taghavi S, Garafola C, Monchy S, Newman L, Hoffman A, Weyens N, Barac T, Vangronsveld J, van der Lelie D. Genome survey and characterization of endophytic bacteria exhibiting a beneficial effect on growth and development of poplar trees. Applied and Environmental Microbiology, 2009, 75(3): 748-757. DOI:10.1128/AEM.02239-08 |
| [5] | Ownley BH, Gwinn KD, Vega FE. Endophytic fungal entomopathogens with activity against plant pathogens: ecology and evolution. BioControl, 2010, 55(1): 113-128. DOI:10.1007/s10526-009-9241-x |
| [6] | Pham VTK, Rediers H, Ghequire MGK, Nguyen HH, de Mot R, Vanderleyden J, Spaepen S. The plant growth-promoting effect of the nitrogen-fixing endophyte Pseudomonas stutzeri A15. Archives of Microbiology, 2017, 199(3): 513-517. DOI:10.1007/s00203-016-1332-3 |
| [7] | Defez R, Andreozzi A, Bianco C. The overproduction of indole-3-acetic acid (IAA) in endophytes upregulates nitrogen fixation in both bacterial cultures and inoculated rice plants. Microbial Ecology, 2017, 74(2): 441-452. DOI:10.1007/s00248-017-0948-4 |
| [8] | Xu T, Li Y, Zeng XD, Yang XL, Yang YZ, Yuan SS, Hu XC, Zeng JR, Wang ZZ, Liu Q, Liu YQ, Liao HD, Tong CY, Liu XM, Zhu YH. Isolation and evaluation of endophytic Streptomyces endus OsiSh-2 with potential application for biocontrol of rice blast disease. Journal of the Science of food and Agriculture, 2017, 97(4): 1149-1157. DOI:10.1002/jsfa.2017.97.issue-4 |
| [9] | Shahzad R, Khan AL, Bilal S, Waqas M, Kang SM, Lee IJ. Inoculation of abscisic acid-producing endophytic bacteria enhances salinity stress tolerance in Oryza sativa. Environmental and Experimental Botany, 2017, 136: 68-77. DOI:10.1016/j.envexpbot.2017.01.010 |
| [10] | Liu Y, Bai FR, Li N, Wang WP, Cheng C. Identification of endophytic bacterial strain RSE1 from seeds of super hybrid rice Shenliangyou 5814 (Oryza sativa L., ) and evaluation of its antagonistic activity. Plant Growth Regulation, 2017, 82(3): 403-408. DOI:10.1007/s10725-017-0265-4 |
| [11] | Shylla A, Shivaprakash MK, Shashidhar HE, Vishwakarma P, Sudradhar M. Production of phytohormones by endophytic bacteria isolated from aerobic rice. Journal of Pure and Applied Microbiology, 2016, 10(3): 2127-2133. |
| [12] | Shahzad R, Waqas M, Khan AL, Al-Hosni K, Kang SM, Seo WC, Lee IJ. Indoleacetic acid production and plant growth promoting potential of bacterial endophytes isolated from rice (Oryza sativa L.) seeds. Acta Biologica Hungarica, 2017, 68(2): 175-186. DOI:10.1556/018.68.2017.2.5 |
| [13] |
Yang B, Chen Y, Li X, Ren CG, Dai CC. Research progress on endophyte-promoted plant nitrogen assimilation and metabolism. Acta Ecologica Sinica, 2013, 33(9): 2656-2664.
(in Chinese) 杨波, 陈晏, 李霞, 任承钢, 戴传超. 植物内生菌促进宿主氮吸收与代谢研究进展. 生态学报, 2013, 33(9): 2656-2664. |
| [14] | Rangjaroen C, Rerkasem B, Teaumroong N, Sungthong R, Lumyong S. Comparative study of endophytic and endophytic diazotrophic bacterial communities across rice landraces grown in the highlands of northern Thailand. Archives of Microbiology, 2014, 196(1): 35-49. |
| [15] | Bodenhausen N, Horton MW, Bergelson J. Bacterial communities associated with the leaves and the roots of Arabidopsis thaliana. PLoS One, 2013, 8(2): e56329. DOI:10.1371/journal.pone.0056329 |
| [16] | Rangjaroen C, Sungthong R, Rerkasem B, Teaumroong N, Noisangiam R, Lumyong S. Untapped endophytic colonization and plant growth-promoting potential of the genus Novosphingobium to optimize rice cultivation. Microbes and Environments, 2017, 32(1): 84-87. DOI:10.1264/jsme2.ME16112 |
| [17] |
Wang XJ, Jia RZ, Guo YL, Xu L, Zuo J, Kong H, Guo AP. Diversity of culturable endobacterial communities in rice (Oryza sativa L.) stem at different growth stages. Chinese Journal of Tropical Crops, 2015, 36(6): 1078-1085.
(in Chinese) 王雪君, 贾瑞宗, 郭运玲, 徐林, 左娇, 孔华, 郭安平. 水稻4个生长时期茎部可培养内生菌多样性分析. 热带作物学报, 2015, 36(6): 1078-1085. DOI:10.3969/j.issn.1000-2561.2015.06.011 |
| [18] |
Li NN, Li N, Cao YH, Zhang X, Xiao M, Liu Y, Wang WP. Diversity of endophytic bacterial communities in three parental seeds of hybrid rice (Oryza sativa L.) at maturity stage. Journal of Food Science and Technology, 2017, 35(4): 56-64.
(in Chinese) 李南南, 黎妮, 曹艳花, 张欣, 肖明, 刘洋, 王伟平. 3个杂交水稻亲本成熟期种子内生细菌多样性研究. 食品科学技术学报, 2017, 35(4): 56-64. |
| [19] |
Li NN, Li N, Zhao R, Yuan LP, Xiao M, Liu Y, Wang WP. Diversity of endophytic bacterial communities in parental seeds of outstanding hybrid rice (Oryza sativa L.) in the milk. Journal of Microbiology, 2017, 37(2): 20-25.
(in Chinese) 李南南, 黎妮, 赵燃, 袁隆平, 肖明, 刘洋, 王伟平. 优异杂交水稻亲本灌浆期种子内生细菌多样性研究. 微生物学杂志, 2017, 37(2): 20-25. DOI:10.3969/j.issn.1005-7021.2017.02.003 |
| [20] |
Jiang XY, Gao JS, Xu FH, Cao YH, Tang X, Zhang XX. Diversity of endophytic bacteria in rice seeds and their secretion of indole acetic acid. Acta Microbiologica Sinica, 2013, 53(3): 269-275.
(in Chinese) 姜晓宇, 高菊生, 徐凤花, 曹艳花, 唐雪, 张晓霞. 水稻种子内生细菌多样性及其分泌植物生长素能力的测定. 微生物学报, 2013, 53(3): 269-275. |
| [21] | Romero FM, Marina M, Pieckenstain FL. The communities of tomato (Solanum lycopersicum L.) leaf endophytic bacteria, analyzed by 16S-ribosomal RNA gene pyrosequencing. FEMS Microbiology Letters, 2014, 351(2): 187-194. DOI:10.1111/1574-6968.12377 |
| [22] | Bulgarelli D, Rott M, Schlaeppi K, van Themaat EVL, Ahmadinejad N, Assenza F, Rauf P, Huettel B, Reinhardt R, Schmelzer E, Peplies J, Gloeckner FO, Amann R, Eickhorst T, Schulze-Lefert P. Revealing structure and assembly cues for Arabidopsis root-inhabiting bacterial microbiota. Nature, 2012, 488(7409): 91-95. DOI:10.1038/nature11336 |
| [23] | Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Pe a AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [24] | Kemp PF, Aller JY. Bacterial diversity in aquatic and other environments: what 16S rDNA libraries can tell us. FEMS Microbiology Ecology, 2004, 47(2): 161-177. DOI:10.1016/S0168-6496(03)00257-5 |
| [25] | Lu L, Xing DF, Ren NQ. Pyrosequencing reveals highly diverse microbial communities in microbial electrolysis cells involved in enhanced H2 production from waste activated sludge. Water Research, 2012, 46(7): 2425-2434. DOI:10.1016/j.watres.2012.02.005 |
| [26] | Koomnok C, Teaumroong N, Rerkasem B, Lumyong S. Diazotroph endophytic bacteria in cultivated and wild rice in Thailand. Science Asia, 2007, 33(4): 429-435. DOI:10.2306/scienceasia1513-1874.2007.33.429 |
| [27] | Tharek M, Dzulaikha K, Salwani S, Amir HG, Najimudin N. Ascending endophytic migration of locally isolated diazotroph, Enterobacter sp. strain USML2 in rice. Biotechnology, 2011, 10(6): 521-527. DOI:10.3923/biotech.2011.521.527 |
| [28] | Klayraung S, Niamsup P, Poonnoy P, Topoonyanont N. Diversity and control of bacterial contamination of plants propagated in temporary immersion bioreactor system. Acta Horticulturae, 2017(1155): 439-446. |
| [29] |
Li QQ, Jiao C, Nong Q, Yuan GQ, Lin W, Huang YL. Dynamic distribution of endophytic bacteria in rice from Guangxi and their antagonism to the pathogen of rice sheath blight. Chinese Journal of Biological Control, 2010, 26(3): 312-319.
(in Chinese) 黎起秦, 焦成, 农倩, 袁高庆, 林纬, 黄永禄. 广西水稻内生细菌的动态分布及其对水稻纹枯病菌的拮抗作用. 中国生物防治, 2010, 26(3): 312-319. |
| [30] | Prasanna R, Nain L, Pandey AK, Saxena AK. Microbial diversity and multidimensional interactions in the rice ecosystem. Archives of Agronomy and Soil Science, 2012, 58(7): 723-744. DOI:10.1080/03650340.2010.537325 |
| [31] | Shi YW, Lou K, Li C, Wang L, Zhao ZY, Zhao S, Tian CY. Illumina-based analysis of bacterial diversity related to halophytes Salicornia europaea and Sueada aralocaspica. Journal of Microbiology, 2015, 53(10): 678-685. DOI:10.1007/s12275-015-5080-x |
| [32] | Sun L, Qiu FB, Zhang XX, Dai X, Dong XZ, Song W. Endophytic bacterial diversity in rice (Oryza sativa L.) roots estimated by 16S rDNA sequence analysis. Microbial Ecology, 2008, 55(3): 415-424. DOI:10.1007/s00248-007-9287-1 |
| [33] | Mano H, Morisaki H. Endophytic bacteria in the rice plant. Microbes and Environments, 2008, 23(2): 109-117. DOI:10.1264/jsme2.23.109 |
| [34] | Mano H, Tanaka F, Nakamura C, Kaga H, Morisaki H. Culturable endophytic bacterial flora of the maturing leaves and roots of rice plants (Oryza sativa) cultivated in a paddy field. Microbes and Environments, 2007, 22(2): 175-185. DOI:10.1264/jsme2.22.175 |
| [35] |
Zou YY, Liu L, Liu Y, Zhao L, Deng QY, Wu J, Zhuang W, Song W. Diversity of indigenous bacterial communities in Oryza sativa seeds of different varieties. Chinese Journal of Plant Ecology, 2012, 36(8): 880-890.
(in Chinese) 邹媛媛, 刘琳, 刘洋, 赵亮, 邓启云, 吴俊, 庄文, 宋未. 不同水稻品种种子固有细菌群落的多样性. 植物生态学报, 2012, 36(8): 880-890. |
| [36] |
Liu Y, Zhao R, Li N, Cao YH, Zhang C, Bai FR, Zhang X, Yuan LP, Wang WP, Cheng C. Diversity of endophytic bacterial communities in seeds of super hybrid rice (Oryza sativa L.). Food and Fermentation Industries, 2016, 42(1): 31-36.
(in Chinese) 刘洋, 赵燃, 黎妮, 曹艳花, 张超, 白飞荣, 张欣, 袁隆平, 王伟平, 程池. 超级杂交水稻种子内生细菌群落结构及其多样性. 食品与发酵工业, 2016, 42(1): 31-36. |
| [37] | Nelson EB. Microbial dynamics and interactions in the spermosphere. Annual Review of Phytopathology, 2004, 42(1): 271-309. DOI:10.1146/annurev.phyto.42.121603.131041 |
| [38] |
Qiu ZB, Wang R, Zhang Y, Wu Q, Xie BJ, Yang JF, Chen JG, Sun ZD. Recent progress in studies of Rhodococcus and its application of in biodegradation. Food Science, 2016, 37(7): 254-258.
(in Chinese) 邱孜博, 汪荣, 张杨, 吴茜, 谢笔钧, 杨季芳, 陈吉刚, 孙智达. 红球菌及其生物降解作用研究进展. 食品科学, 2016, 37(7): 254-258. |
| [39] |
Zhang YZ, Bao ZX, Liu GX, Liu YT. Biophysical and biochemical properties of Ralstonia eutropha H16 novel toxin protein Reu. Journal of Henan Agricultural Sciences, 2010, 39(3): 55-58.
(in Chinese) 张一折, 包振霞, 刘国祥, 刘永涛. 罗尔斯通氏菌菌株H16新毒素蛋白Reu理化性质的研究. 河南农业科学, 2010, 39(3): 55-58. DOI:10.3969/j.issn.1004-3268.2010.03.016 |
| [40] | 沙月霞.防治稻瘟病芽胞杆菌的筛选及生防机制研究.中国农业大学博士学位论文, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10019-1015564028.htm |
 2018, Vol. 58
2018, Vol. 58